Załącznik 1
Instrukcja szczegółowa sporządzania raportów okresowych
o sytuacji epidemiologicznej w szpitalu za 2016 rok
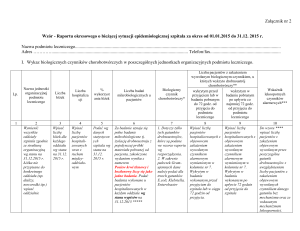
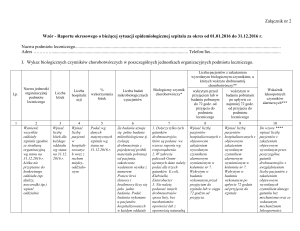
1. W raporcie nie należy pozostawiać pustych pól, w wersji papierowej w przypadku braku
danych należy wpisać symbol „x” a w przypadku wartości zerowej, wpisać „0”. Natomiast
w wersji elektronicznej w każdej z ww. sytuacji wpisać „0”, gdyż to umożliwi zbiorcze
sumowanie danych.
2. W kolumnie nr 5 należy podać wskaźnik wykorzystania łóżek obliczony na podstawie
liczby osobo-dni wyliczonej z uwzględnieniem pierwszego dnia pobytu (data przyjęcia)
bez uwzględnienia dnia wypisu.
3. W kolumnie numer 7 należy wpisywać gatunki drobnoustrojów z nabytymi
mechanizmami lekooporności, podane w załączniku nr 4 rozporządzenia, o ile
występowały objawowe zakażenia tymi czynnikami. Naturalna lekooporność
drobnoustrojów nie spełnia ustawowego kryterium czynnika alarmowego, z tego względu
należy pominąć te gatunki bakterii, które wykazują naturalną oporność na różne
antybiotyki.
4. Lista drobnoustrojów alarmowych, które należy uwzględnić w raporcie, o ile występują:
1) gronkowiec złocisty (Staphylococcus aureus) oporny na metycylinę (MRSA) lub
glikopeptydy (VISA lub VRSA) lub oksazolidynony;
2) enterokoki (Enterococcus spp.) oporne na glikopeptydy (VRE) lub oksazolidynony;
3) pałeczki Gram-ujemne Enterobacteriaceae spp. wytwarzające beta-laktamazy
o rozszerzonym spektrum substratowym (np. ESBL, AMPc,) lub oporne na karbapenemy
(w mechaniźmie wytwarzania karbapenemaz, KPC, NDM) lub inne dwie grupy leków lub
polimyksyny.
W raportach w wersji elektronicznej, dane należy ograniczyć do trzech gatunków tj. E.coli
spp, Klebsiella spp, Enterobacter spp ze zsumowanymi nabytymi mechanizmami oporności,
oprócz oporności na karbapenemy, którą należy wyszczególnić w oddzielnej pozycji. W
przypadku Klebsiella NDM(+) należy wykazać zarówno zakażenia objawowe jak również
w odrębnej kolumnie liczbę pacjentów z kolonizacją.
4) pałeczka ropy błękitnej (Pseudomonas aeruginosa) oporna na karbapenemy lub inne
dwie grupy leków lub polimyksyny;
5) pałeczki niefermentujące Acinetobacter spp. oporne na karbapenemy lub inne dwie
grupy leków lub polimyksyny;
6) szczepy chorobotwórcze laseczki beztlenowej Clostridium difficile - dla tego gatunku
nie podaje się danych dotyczących lekowrażliwości, rozpoznanie ustala się głównie na
1
podstawie występowania biegunki oraz dodatniego wyniku badania toksyny A/B ew.
toksynotwórczości szczepu potwierdzonej innymi metodami;
7) laseczka beztlenowa Clostridium perfringens - dla tego gatunku nie podaje się danych
dotyczących lekowrażliwości;
8) dwoinka zapalenia płuc (Streptococcus pneumoniae) oporna na cefalosporyny
III generacji lub penicylinę;
9) grzyby Candida oporne na flukonazol lub inne leki z grupy azoli lub kandyn. W zakresie
grzybów opornych na flukonazol należy podać dane wyłącznie dotyczące gatunków
z nabytą opornością na flukonazol lub inne leki z grupy azoli tj. Candida albicans.
10) grzyby Aspergillus; dla tego gatunku drobnoustroju nie podaje się danych dotyczących
lekowrażliwości;
11) wirusy, NORO, ROTA, RSV - dla wirusów nie podaje się danych dotyczących
lekooporności.
5. Czas podany w kolumnach nr 8 i 9 należy liczyć od momentu przyjęcia do szpitala do
chwili pobrania materiału na badania mikrobiologiczne.
6. W kolumnie nr 8 wpisuje się liczbę pacjentów, u których stwierdzono objawowe zakażenie
czynnikiem alarmowym wymienionym w kolumnie nr 7 w posiewie pobranym w zakresie
0-72 godziny od momentu przyjęcia, w kolumnie nr 9 wpisuje się liczbę pacjentów, u których
stwierdzono objawowe zakażenie czynnikiem alarmowym wymienionym w kolumnie nr 7
w posiewie pobranym po 72 godzinach od momentu przyjęcia.
7. W kolumnie nr 10 do wyliczenia wskaźnika lekooporności konieczne jest uzyskanie
danych dotyczących co najmniej 30 izolatów pochodzących z zakażeń objawowych
i wyliczenie według wzoru podanego pod tabelą w formularzu. Jeśli łączna liczba pacjentów
z zakażeniem wywołanym określonym gatunkiem drobnoustroju na poszczególnych
oddziałach jest niższa niż 30, to należy podać jeden wspólny wskaźnik oporności dla całego
szpitala zaznaczając to w raporcie, na końcu tabeli.
W przypadkach, gdy łączna liczba pacjentów z zakażeniem wywołanym czynnikiem danego
gatunku jest niższa niż 30, nie należy wyliczać wskaźnika, a w tabeli
w wersji papierowej i elektronicznej wstawić „0” wyjaśniając pod tabelą, że liczebność
szczepów jest zbyt mała, aby wyliczyć w/w wskaźnik. Do wyliczenia tego wskaźnika
konieczna jest znajomość liczby pacjentów z zakażeniem objawowym wywołanym
czynnikiem biologicznym określonego gatunku, niewykazującym danego mechanizmu
lekooporności. Optymalne jest, aby było to potwierdzone na podstawie indywidualnej analizy
każdego przypadku, jednak w sytuacji dużej liczby badań może to być zbyt czasochłonne,
dlatego możliwe jest zastosowanie procedury uproszczonej, polegającej na przyjęciu wykazu
materiałów uznanych za istotne kliniczne i podstawieniu do w/w wzoru liczby pacjentów, od
których wyizolowano z w/w materiału drobnoustrój podany w kolumnie nr 7. Pod tabelą
należy podać w komentarzu, jaką metodą wyliczono w/w wskaźnik tj. czy na podstawie
2
indywidualnej analizy czy procedury uproszczonej. Zgodnie z aktualną wiedzą, za materiały
istotne klinicznie uznaje się:
materiał z kanału szyjki macicy od pacjentki z zakażeniem narządu rodnego
kał od chorego z biegunką infekcyjną,
krew od pacjenta z zakażeniem inwazyjnym,
jałowe płyny z jam ciała (mózgowo-rdzeniowy, otrzewnowy, stawowy, opłucnowy)
od pacjenta z zakażeniem inwazyjnym,
materiał ze świeżo zakażonej rany (z wyłączeniem materiału m.in. z odleżyn,
owrzodzeń, oparzeń pobieranego jako powierzchowny wymaz),
aspirat tchawiczy lub materiał bronchoskopowy od chorego z zapaleniem płuc,
mocz od chorego z zakażeniem układu moczowego
materiał śródoperacyjny od pacjenta z zakażeniem w obrębie operowanego
narządu/okolicy.
Nie podaje się wskaźnika lekooporności dla następujących gatunków czynników
alarmowych: Clostridium difficile, Clostridium perfringens, Aspergillus, rotawirus, noro
wirus.
3