Struktura i funkcja białek
(I mgr)
dr Katarzyna Śmietana
[email protected]
http://www.protein.pl/protein/downup/struk-funk/
Jeremy M. Berg, John L. Tymoczko, Lubert Stryer „Biochemia”
Carl Branden, John Tooze “Introduction to Protein Structure”
Wykład 1
- cztery główne typy funkcjonalne białek
- aminokwasy: wzory, właściwości, szczególne cechy kodu genetycznego,
- struktury II, III, IV rzędowe białek, mapa Ramachandrana,
- preferencje do występowania w określonych strukturach II-rzędowych,
- wiązanie peptydowe: cechy charakterystyczne, kąty,
- typy oddziaływań, odległość, przykłady,
- elementy stabilizujące struktury białek,
- efekt hydrofobowy (zwijanie białek, oddziaływanie białko-białko)
Wykład 2
- motywy strukturalne i funkcjonalne (przykłady), domeny alfa, beta,
alfa/beta, alfa + beta
- oligomeryzacja białek (typy, przykłady)
- rozpoznanie, komplementarność i centra aktywne, elastyczność białek,
białka szkieletowe,
- domena PDZ, cechy charakterystyczne wiązania ligandów, struktura,
specyficzność
Wykład 3
- poziomy kontroli funkcji białek
- mechanizmy kierowania i regulacji białek
- kontrola funkcji białek: degradacja, kowalencyjne modyfikacje, proteoliza,
lipidacja, metylacja, N-acetylacja, sumoilacja, nitrozylacja, fosforylacja
białek, przykłady, wpływ na strukturę/aktywność
- molekularne przełączniki
-- cykl aktywności białek G, minimalna domena G, zasada działania, białka
GAP, GEF, GDI – budowa i mechanizm działania, model działania
mięśniowej miozyny i kinezyny
- kontrola funkcji białek przez modyfikacje kowalencyjne (fosforylacja,
lipidacja, metylacja, N-acetylacja, sumoilacja, nitrozylacja)
- degradacja, proteoliza i składanie białek, czterostopniowy mechanizm
splicingu białek
Wykład 4
Od sekwencji do funkcji:
-- sekwencje homologiczne, dopasowanie sekwencyjne, motywy
strukturalne i funkcjonalne (przykład), zasada 40%
-- mikromacierze DNA, żele 2D, Y2H
-- modelowanie homologiczne, profile-based threading, metody Rosetta,
metoda GRID, THEMATICS
-- sekwencje kameleonowe
Biosynteza białka - etapy, funkcje poszczególnych IF, EF i RF, ogólny opis
struktury krystalicznej rybosomu, czym jest PTC, synteza wiązania
peptydowego, tunel wyjściowy, L22 i SecM
Wykład 5
- kinazy białkowe, struktura przestrzenna, mechanizm aktywacji i działania
kinaz Src i układu Cdk2-cyklina A
- dwuskładnikowy mechanizm sygnalizacyjny bakterii
- przeciwciała, MHC I i II, receptor T: budowa, genetyczne i strukturalne
podłoże różnorodności
- p53, ogólne informacje, budowa domeny oligomeryzacyjnej i wiążącej DNA,
kluczowe reszty, białka natywnie niezwinięte
- struktura i funkcja bakteriorodopsyny, poryn, kanału potasowego,
- trzy typy białek fibrylarnych,
- kolagen, filamenty pośrednie, włókna amyloidowe: budowa i cechy
szczególne,
- foldy metastabilne – priony, amyloidy i serpiny
- białka opiekuńcze
- podstawowa jednostka symetrii wirusów sferycznych, budowa wybranych
wirusów
Wykład 6
?
Białka
• stanowią aż 75% suchej masy tkanek miękkich naszego ciała
• z gr. proteios - pierwszorzędny, o największym znaczeniu
• białka są polimerami zbudowanymi z zestawu 20 różnych
aminokwasów połączonych wiązaniami peptydowymi -> już
dla stuaminokwasowego białka liczba możliwych sekwencji
aminokwasowych (20100) przekracza 10130
• znaczne zróżnicowanie właściwości fizykochemicznych
poszczególnych reszt aminokwasowych umożliwia tworzenie
przez łańcuchy białkowe bardzo różnorodnych struktur
trójwymiarowych odznaczających się często dużym stopniem
komplikacji – pozwala to na pełnienie przez białka tak wielu
różnych funkcji
• różnorodność białek jest widoczna już w ich rozmiarach –
znane są białka o masach od tysiąca do ponad miliona Da
Białka są najbardziej różnorodnymi i wielofunkcyjnymi
cząsteczkami występującymi w komórce
Wiązanie różnorodność
rozpoznawanych
cząsteczek;
komplementarność
kształtu i oddziaływań
polarnych
Kataliza przyspieszanie reakcji
do 17 rzędów wielkości,
odpowiednie ułożenie
grup reaktywnych,
stabilizacja stanu
przejściowego, kataliza
kwasowo-zasadowa
Molekularny
przełącznik zmiana konformacyjna
pod wpływem pH
lub wiązania liganda
przełącza funkcję
Białka strukturalne specyficzna asocjacja
podjednostek i białek
pozwala na spontaniczne
powstawanie nawet bardzo
złożonych struktur
Wyróżniamy cztery poziomy organizacji struktury białek
Od właściwości łańcuchów bocznych zależy wkład różnych reszt
aminokwasowych w zwijanie i funkcje białek
w pH 7 końcowe grupy łańcucha polipeptydowego
(aminowa i karboksylowa) są zjonizowane
-zaangażowane tylko w oddziaływania van der Waalsa
-unikanie wody i pakowanie się na siebie są podstawą efektu hydrofobowego
-Ala i Leu faworyzują powstawanie helisy w przeciwieństwie do proliny
-aromatyczny pierścień Phe czasami uczestniczy w słabych polarnych oddziaływaniach
-mogą tworzyć wiązania wodorowe między sobą, z łańcuchem głównym i innymi
cząsteczkami polarnymi (m.in. z wodą)
-niektóre dysponują ładunkiem (w zależności od pH i otoczenia)
- aa hydrofilowe tworzą wiązania wodorowe ze sobą,
z łańcuchem głównym, z polarnymi związkami
organicznymi i z wodą
- Glu i Asp pKa 5 (ujemnie naładowane w pH 7),
ale w otoczeniu hydrofobowym pKa może wzrosnąć
powyżej 7 i wtedy służą jako donory protonu
- Lys pKa 10 (w pH 7 dodatnio naładowana),
ale w otoczeniu hydrofobowym może spaść poniżej 6
i wtedy Lys staje się akceptorem protonu
- His ma dwie grupy –N-H, każda o pKa około 6
-- gdy jedna traci proton, pKa drugiej staje się większa niż 10
-- gdy obie są uprotonowane His jest ujemnie naładowana
(bardzo rzadko)
-- gdy jedna jest uprotonowana His jest neutralna i zdolna
do przyjęcia bądź oddania protonu
- Arg jest prawie zawsze uprotonowana
- Grupa –SH cysteiny jest najsilniejszym nukleofilem
- Ser, Thr, Gln i Asn są akceptorami i donorami protonu
-- aa amfipatyczne najczęściej uczestniczą w tworzeniu
interfejsów między elementami hydrofobowymi a polarnymi
- Tyr zwykle nie jonizuje w pH 7 (pKa=9). Grupa –OH jest
donorem i akceptorem wiązania wodorowego,
a aromatyczny pierścień Tyr uczestniczy w słabych
oddziaływaniach polarnych
- Met jest najmniej polarna z grupy aa amfipatycznych,
ale jej siarka jest często uczestniczy w oddziaływaniach
z jonami metali
Sposób organizacji kodu genetycznego odzwierciedla wzajemne
podobieństwa aminokwasów
Więcej białek niż genów u Eukariontów:
-alternatywny splicing
-editing RNA
Częstość podstawień aminokwasowych w tym samym
białku pochodzącym z różnych organizmów
„conservative substitutions”
Tworzenie i hydroliza wiązania peptydowego
Wiązanie kowalencyjne
tworzone pomiędzy kwasem
karboksylowym i grupą aminową
dwóch α-aminokwasów
z uwolnieniem jednej cząsteczki
wody. Wiązania amidowe są
bardzo stabilne w środowisku
wodnym w pH bliskim 7.
W komórce tworzenie i hydroliza
wiązań peptydowych
kontrolowane są
enzymatycznie.
Rozciągnięty łańcuch polipeptydowy
Rezonans – delokalizacja elektronów
w obrębie wiązania peptydowego powoduje:
-częściowy charakter wiązania podwójnego
(CNO w jednej płaszczyźnie - dwa kąty
torsyjne - ograniczona możliwość rotacji
i konformacji; podwyższona stabilność)
-polarność wiązania peptydowego, moment
dipolowy (możliwe oddziaływania)
Oddziaływania stabilizujące białko
oddziaływania z wodą lub za jej pośrednictwem
C=O i N-H we wnętrzu białka nie mogą tworzyć wiązań wodorowych
z cząsteczkami wody, więc tworzą je między sobą prowadząc do powstania
struktur II rzędowych i stabilizacji struktury
Wykres Ramachandrana
Prawie we wszystkich białkach
łańcuch główny przyjmuje taką
konformację, w której kąty
torsyjne phi i psi powtarzają
się, tworząc regularne wzory –
elementy struktury
drugorzędowej
Ograniczenia steryczne
określają możliwe typy
struktury drugorzędowej
Zgięcie beta (beta/hairpin/reverse turn)
najprostszy element struktury II-rzędowej
-NH
-O
n+3
(n+2)
Część grup C=O i N-H nie tworzy wiązań wodorowych
w obrębie łańcucha, dlatego zgięcia występują
najczęściej na powierzchni cząsteczki białka
Obecność zgięć beta pozwala ograniczyć rozmiary
białka i nadać mu bardziej zwartą strukturę
Helisa alfa – najpopularniejsza struktura II-rzędowa
wiązania wodorowe pomiędzy C=O i N-H
znajdującymi się blisko siebie w sekwencji
3.6 reszty
na skręt
n+4
Parametry elementów helikalnych
n+4
n+3
n+5
Amfipatyczność helis alfa
-łańcuchy boczne „wystają”
co 100o
-reszty oddalone od siebie o 3-4
aminokwasy grupują się po tej
samej stronie helisy
-helisy amfipatyczne stabilizują
pakowanie helisa-helisa
i często występują
na powierzchni białka
Helisy zawierające reszty proliny
Struktura kolagenu
helisa lewoskrętna
(Gly-X-Y)n gdzie X, Y to Pro, Pro-OH lub (rzadziej) Lys
Helisy poliprolinowe
wszystkie proliny w formie trans
tworzą lewoskrętną helisę (3 reszty na skręt);
motyw w białkach sygnalnych rozpoznawany przez domeny SH3
Struktury beta
bardziej stabilna
nie są eksponowane do rozpuszczalnika,
najczęściej „przeplatane” helisami
Silnie rozciągnięty, zwykle lekko skręcony łańcuch polipeptydowy
odległość między resztami 3,3 Å
Możliwość utworzenia struktury amfipatycznej
Kompleks Rap–Raf
pakowanie helisy
na równoległe włókno beta
międzycząsteczkowa struktura beta jako interfejs
oddziaływania białko-białko
Baryłka beta (b-barrel)
-włókna beta, ze względu
na konfiguracje L aminokwasów,
mają tendencję do prawoskrętu
-aminokwasy rozgałęzione
na węglu beta (Val, Ile) łatwo
akomodują się w strukturze
beta (w porównaniu z gęsto
upakowaną helisą)
białko wiążące retinol z niepolarnym
ligandem związanym we wnętrzu baryłki
Przewidywanie struktury drugorzędowej
- trafność przewidywań
zaledwie ok. 70%
- najłatwiej określić
położenie helis (tworzone
dzięki oddziaływaniom
reszt sąsiadujących
ze sobą w sekwencji)
- najtrudniej przewidzieć
granice pętli i miejsca zgięć
Tworzenie struktury trzeciorzędowej
Intermediaty zwijania (barnaza)
Efekt hydrofobowy
-minimalizacja powierzchni
hydrofobowej dostępnej
dla rozpuszczalnika
-zbliżenie polaryzowalnych
grup hydrofobowych
umożliwia powstanie między
nimi oddziaływań van der
Waalsa
-tym samym „wciągniecie”
polarnych grup C=O i N-H
łańcucha głównego staje się
siłą sprawczą powstania
struktur drugorzędowych
Porównanie struktur TIM (izomerazy triozofosforanowej)
i DHFR (reduktazy kw. dihydrofoliowego)
-podobne elementy struktury drugorzędowej
mogą tworzyć białka o całkiem różnych
strukturach III°
-wynika to ze sposobu łączenia elementów występowania pętli lub zgięć
Pętle
-pętle w białkach zwykle
ulokowane są na powierzchni,
eksponowane do rozpuszczalnika
-ponieważ ich wkład w stabilizację
struktury białka jest niewielki,
w obrębie pętli łatwiej tolerowane
są mutacje
-dzięki temu pętle łatwo
dostosowują się do powierzchni
ligandów i często tworzą interfejsy
oddziaływań białko-białko
Pierwsza powłoka hydratacyjna elastazy
-cząsteczki wody ściśle związane z białkiem
stanowią część jego struktury
Wnętrze zwiniętego białka
- atomy we wnętrzu są upakowane
prawie jak w ciele stałym
-kanały i szczeliny pozwalają
jednak na ruch atomów
i zapewniają elastyczność
-jeśli w rdzeniu znajdują się
większe przestrzenie,
to wypełnione są cząsteczkami
wody, które mogą oddziaływać
z okolicznymi grupami polarnymi
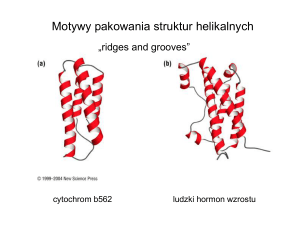
Motywy pakowania struktur helikalnych
„ridges and grooves”
cytochrom b562
ludzki hormon wzrostu
Symulowana struktura fragmentu dwuwarstwy lipidowej
20 x 1,5 Å - helisa
8-9 x 3.5 Å - włókno beta
-otoczenie hydrofobowe
powoduje, że wszystkie polarne
grupy (w tym C=O i N-H
łańcucha głównego) muszą
zostać „zagrzebane” we wnętrzu
białka – odwrotnie niż
w środowisku wodnym
Fragment kompleksu cytochromu bc1
elementy przezbłonowe najczęściej są helikalne,
ze względu na możliwość lokalnego utworzenia
wiązań wodorowych w obrębie łańcucha głównego
Profil hydrofobowości białka DctB z Rhizobium meliloti
Błonowe białka beta
Struktura transportera FhuA
-włókno przezbłonowe = 8-9 reszt
-zwykle pod kątem => trudniejsze
do przewidzenia
-zamknięte baryłki aby zaspokoić HB
-często tworzą kanały
Struktura bakteryjnego kanału potasowego
homotetramer
Zorganizowane cząsteczki wody na eksponowanej
powierzchni hydrofobowej rybonukleazy A
Pojedyncze słabe
oddziaływanie uwalnia
około 4-13 kJ/mol energii
swobodnej
Zwinięte natywnie białko jest termodynamicznym kompromisem.
Stabilność można zdefiniować jako utratę energii swobodnej,
będącą sumą efektów entalpowych i entropowych.
Dla większości białek różnica energii między stanem rozwiniętym
a zwiniętym jest marginalna, około 21-42 kJ/mol.
Stabilność, denaturacja, termofilność
-marginalna stabilność dopuszcza duży
stopień swobody (niezbędny
do wzajemnego dopasowania podczas
oddziaływań z ligandami czy katalizy)
-stan zdenaturowany rozpoznajemy
po utracie aktywności
biologicznej/biochemicznej lub zmianie
sygnału np. dichroizmu kołowego
-denaturację można wymusić wysoką
temperaturą oraz chemicznymi
denaturantami (chlorowodorek
guanidyny, mocznik, SDS), które
współzawodniczą z polarnymi grupami
białka o wiązania wodorowe
-stabilizację białek można uzyskać
poprzez skracanie pętli, dodawanie
mostków solnych itp.
Struktura BPTI
stabilizacja przez mostki S-S
-zwykle struktura białka jest
stabilizowana przede wszystkim
przez oddziaływania
niekowalencyjne
-w niektórych przypadkach istotną
rolę mogą też pełnić oddziaływania
kowalencyjne, np. tworzenie
mostków disulfidowych
-dotyczy to głównie białek
zewnątrzkomórkowych, ponieważ
warunki panujące w komórce są
silnie redukujące
Fragment struktury subtylizyny
stabilizacja przez koordynację metalu (Ca2+)
-Kd wiązania metalu
w zakresie od mM
(bardzo słabe) do nM
(bardzo mocne)
-w koordynacji mogą
uczestniczyć cząsteczki
wody
-w niektórych
przypadkach białka
strukturyzują się
wyłącznie w obecności
jonów metalu, a ich
usunięcie prowadzi
do denaturacji
Stabilizacja przez wiązanie kofaktora
dotyczy miejsc aktywnych
DaAT/pirydoksal
mioglobina/hem+żelazo
cytochrom c/hem+żelazo
oksydaza
poliaminowa
/PQQ
Modyfikacje potranslacyjne
wpływające na stabilność białka
SUMOylation
S-nitrosylation
Domeny strukturalne
domeny tetrameryzacyjne
domeny wiążące DNA
Diagram tetrameru Lac represora wiążącego się do DNA
-białka fibrylarne/globularne
-domena – zwarty obszar w strukturze białka, zwykle
(ale nie zawsze) tworzony przez ciągłą sekwencję aa,
często posiadający zdolność zwijania się
i funkcjonowania w sposób niezależny od reszty białka
-domeny mają nie więcej niż 250 aa (49% w granicach
51-150aa)
Domeny strukturalne
struktura racemazy alaniny
- nie wszystkie domeny budowane są przez ciągły
odcinek sekwencji
- rdzenie hydrofobowe domen białkowych
Domeny strukturalne
-białka wielodomenowe
ewoluowały przez fuzję
genów kodujących
pojedyncze białka
-im dawniej nastąpiła
duplikacja, tym trudniej
dostrzec podobieństwo
(coraz więcej
nagromadzonych
mutacji)
monomer
(tioesteraza)
homodimer
(dehydraza tioestrowa)
Struktura gamma-krystaliny z soczewki oka
podwójna duplikacja
w obrębie tej samej domeny
Struktury syntazy Trp i dehydratazy galaktonianiu
Podobna alfa/beta baryłka (żółta)
mimo całkowitego braku
pokrewieństwa oraz podobieństwa
sekwencji czy funkcji obu białek
-ograniczona liczba sposobów zwijania
-domain fold – topograficzny układ elementów struktury
drugorzędowej, charakterystyczny dla danej domeny
Diagram aranżacji domen w białkach
zaangażowanych w sygnalizację
komórkową
-funkcjonowanie poszczególnych
modułów może (ale nie musi) zależeć
od ich kolejności w łańcuchu
polipeptydowym => możliwe
zamienianie i dokładanie domen
-wyodrębniamy rodziny białek
w zależności od architektury
domenowej
Struktura reduktazy aldozy (redukcja, NADPH)
i fosfotriesterazy (hydroliza, jon metalu)
ten sam sposób zwinięcia – zupełnie inna funkcja i mechanizm katalizy
Struktura aminotransferaz
-katalizują tą samą reakcję
-brak podobieństwa sekwencji
i struktury przestrzennej,
z wyjątkiem bardzo podobnych
miejsc aktywnych
Na następnym wykładzie:
-motywy strukturalne i funkcjonalne (przykłady), domeny alfa, beta,
alfa/beta, alfa + beta
- oligomeryzacja białek (typy, przykłady)
- rozpoznanie, komplementarność i centra aktywne,
- elastyczność białek, białka szkieletowe,
- domena PDZ, cechy charakterystyczne wiązania ligandów, struktura,
specyficzność