Nr wniosku: 168919, nr raportu: 15150. Kierownik (z rap.): dr inż. Karolina Susek
Gatunki rodzaju Lupinus (łubin) należą do rodziny Fabaceae i charakteryzują się cennymi
właściwościami biologicznymi, wykorzystywanymi w rolnictwie, żywieniu zwierząt,
a ostatnio coraz częściej również ludzi. Ze względu na ich geograficzne występowanie, można
wyróżnić łubiny Starego Świata oraz Nowego Świata. Gatunki łubinów wykazują się dużym
zróżnicowaniem nie tylko geograficznym, ale również genomowym. Liczba chromosomów
(2n=32-52), podstawowa liczba chromosomów (x=6-9, 13) a także wielkość genomów (0,97
pg/2C – 2,68 pg/2C) łubinów uwidaczniają złożoną organizację i ewolucję genomów Lupinus.
W ramach projektu podjęto próbę odpowiedzi na pytanie czy i jakie zmiany w genomach
roślinnych zachodziły przez wiele milionów lat ewolucji. W celu identyfikacji przemian
chromosomowych (tj. delecja, duplikacja, insercja) przeprowadzono porównawcze analizy
cytogenetyczne obejmujące analizę kariotypów (zestaw chromosomów w komórce
somatycznej) łubinów Starego Świata, tzn. występujących wokół basenu Morza Śródziemnego
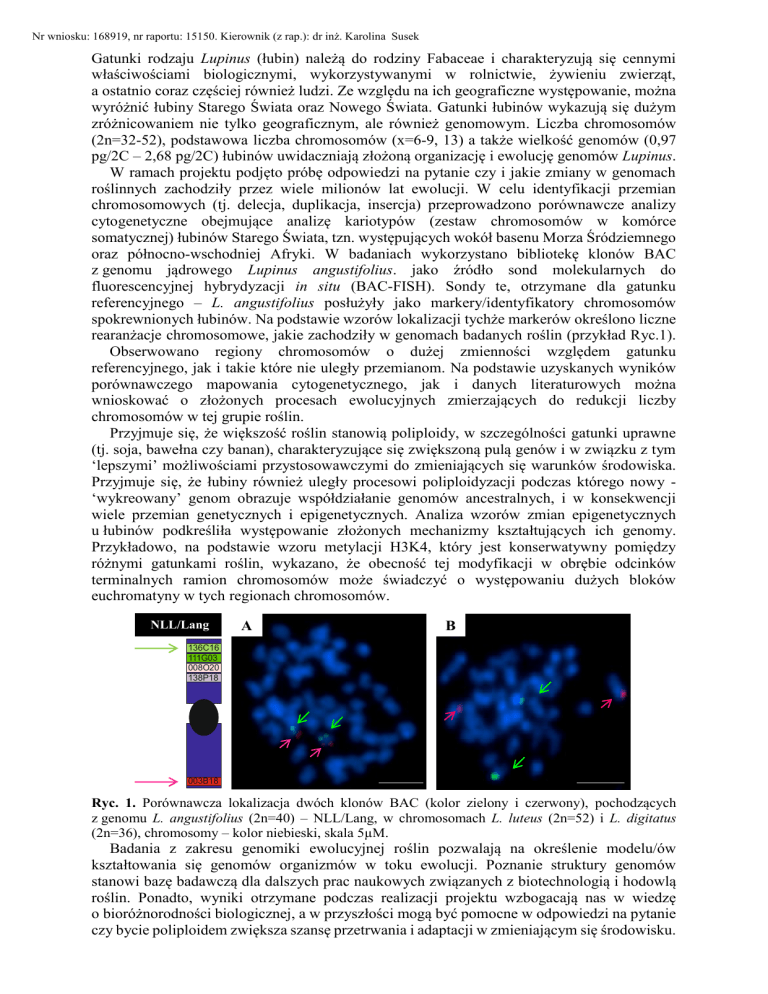
oraz północno-wschodniej Afryki. W badaniach wykorzystano bibliotekę klonów BAC
z genomu jądrowego Lupinus angustifolius. jako źródło sond molekularnych do
fluorescencyjnej hybrydyzacji in situ (BAC-FISH). Sondy te, otrzymane dla gatunku
referencyjnego – L. angustifolius posłużyły jako markery/identyfikatory chromosomów
spokrewnionych łubinów. Na podstawie wzorów lokalizacji tychże markerów określono liczne
rearanżacje chromosomowe, jakie zachodziły w genomach badanych roślin (przykład Ryc.1).
Obserwowano regiony chromosomów o dużej zmienności względem gatunku
referencyjnego, jak i takie które nie uległy przemianom. Na podstawie uzyskanych wyników
porównawczego mapowania cytogenetycznego, jak i danych literaturowych można
wnioskować o złożonych procesach ewolucyjnych zmierzających do redukcji liczby
chromosomów w tej grupie roślin.
Przyjmuje się, że większość roślin stanowią poliploidy, w szczególności gatunki uprawne
(tj. soja, bawełna czy banan), charakteryzujące się zwiększoną pulą genów i w związku z tym
‘lepszymi’ możliwościami przystosowawczymi do zmieniających się warunków środowiska.
Przyjmuje się, że łubiny również uległy procesowi poliploidyzacji podczas którego nowy ‘wykreowany’ genom obrazuje współdziałanie genomów ancestralnych, i w konsekwencji
wiele przemian genetycznych i epigenetycznych. Analiza wzorów zmian epigenetycznych
u łubinów podkreśliła występowanie złożonych mechanizmy kształtujących ich genomy.
Przykładowo, na podstawie wzoru metylacji H3K4, który jest konserwatywny pomiędzy
różnymi gatunkami roślin, wykazano, że obecność tej modyfikacji w obrębie odcinków
terminalnych ramion chromosomów może świadczyć o występowaniu dużych bloków
euchromatyny w tych regionach chromosomów.
NLL/Lang NLL/Lang
A
B
136C16
111G03
008O20
138P18
003B18
Ryc. 1. Porównawcza lokalizacja dwóch klonów BAC (kolor zielony i czerwony), pochodzących
z genomu L. angustifolius (2n=40) – NLL/Lang, w chromosomach L. luteus (2n=52) i L. digitatus
(2n=36), chromosomy – kolor niebieski, skala 5µM.
Badania z zakresu genomiki ewolucyjnej roślin pozwalają na określenie modelu/ów
kształtowania się genomów organizmów w toku ewolucji. Poznanie struktury genomów
stanowi bazę badawczą dla dalszych prac naukowych związanych z biotechnologią i hodowlą
roślin. Ponadto, wyniki otrzymane podczas realizacji projektu wzbogacają nas w wiedzę
o bioróżnorodności biologicznej, a w przyszłości mogą być pomocne w odpowiedzi na pytanie
czy bycie poliploidem zwiększa szansę przetrwania i adaptacji w zmieniającym się środowisku.










