Struktura i ewolucja genomów roślinnych
Liczba chromosomów
• Metody ustalania: analiza w mikroskopie
świetlnym gniecionych preparatów komórek.
• Rośliny nasienne: od: 2n=4 (np.Haplopappus
gracilis) do 2n=ok.600 (Voanioala gerardii)
Poliploidy
• Wnioski na podstawie analizy kariotypów.
• 50-70% roślin kwiatowych przeszło podwojenie liczby
chromosomów co najmniej raz w swojej historii
ewolucyjnej.
• Poliploidy częste wśród roślin użytkowych: pszenica,
rzepak, ziemniak, bawełna.
• Auto- i allopolipolidy.
Rozmiar genomów
•
Metody ustalania: Kinetyka
reasocjacji DNA, pomiary
objętości jądra kom., oceny na
podst, próbek klonów z bibliotek
genomowych, mikrodesytometria
jąder po barwieniu odcz.
Feulgena, cytometria
przepływowa izolowanych jąder
po barwieniu jodkiem propiodyny.
•
Na podstawie analiz >2800
gatunków roślin nasiennych:
Haploidalny genom od 0.1 pg do125
pg.
Ok. 50% roślin kwiatowych: 0.1 – 3.5
pg.
Arabidopsis 125 Mb DNA – Fritillaria
assyriaca – 120 000 Mb.
Różnice także miedzy gatunkami z
jednej rodziny: np. Poaceae :450
Mb (ryż), 750 Mb (sorgo), 2500
Mb (kukurydza), 5000 Mb
(jęczmień), 16 000Mb (pszenica)
WEWNĘTRZNA STRUKTURA GENOMÓW
• Paralogia – bliskie podobieństwo nie allelicznych segmentów
chromosomów lub sekwencji DNA w obrębie gatunku, wskazuje na
bliskie pokrewieństwo ewolucyjne. Paralogami są np. dwa różne
ludzkie geny a-globinowe.
• Ortologia – bliskie podobieństwo segmentów chromosomów lub
sekwencji DNA pomiędzy gatunkami. Ortologami są np. główny
ludzki gen determinujący płeć SRY i jego odpowiednik (ortolog) u
myszy – gen Sry.
• Homologia – ogólny termin określający podobieństwa sekwencji
wskazujące na wspólne pochodzenie ewolucyjne (wewnątrz- lub
pomiędzy gatunkami).
Kinetyka reasocjacji DNA
•
•
•
DNA cięty na odcinki ok.. 300 pz,
denaturowany, następnie
inkubowany w różnych stężeniach
w ciągu różnych okresów czasu.
Analiza - pomiar
niezreasocjowanego
(jednoniciowego) DNA
(chromatografia na
hydroksyapatycie)
C0 – stężenie jednoniciowego
DNA ( w molach nukleotydów/litr)
na początku reakcji, t – czas
reasocjacji w sekundach
Złożoność sekwencyjna genomów roślinnych na podstawie
analizy kinetyk reasocjacji
• Złożone z sekwencji powtarzalnych i jednokopijnych
• Różnice w rozmiarach genomów głównie z powodu różnej ilości
sekwencji powtarzalnych (istotny także poziom ploidalności).
• Podział sekwencji powtarzalnych: a) ułożone tandemowe
(większość w centromerach, telomerach) i b) rozrzucone po
genomie.
• W klasie b) najczęściej transpozony. W Arabidopsis stanowią
ok.10% genomu, w kukurydzy - 50%
Organizacja centromerów w Arabidopsis
Rodzaje sekwencji w genomach roślin-I
• Elementy transpozonowe: (na przykładzie
Arabidopsis) - copia- i gypsy-like long
terminal repeat (LTR) retrotransposons,
long interspersed nuclear elements
(LINEs); short interspersed nuclear
elements (SINEs), hobo/Activator /Tam3
(hAT)-like elements, CACTA-like elements
and miniature inverted-repeat
transposable elements (MITEs).
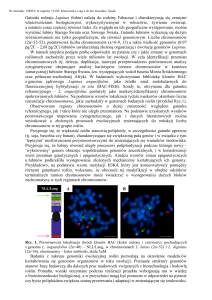
Model obszarów genowych w genomach roślin
•
•
Strzałki = geny (eksony
zaznaczone na czarno)
MITE =miniature inverted
transposable elements
•
•
LTR = long terminal repeat
SSR = simple sequence repeat
Struktura chromosomów Arabidopsis
Rodzaje duplikacji w Arabidopsis
Porównanie rejonów zduplikowanych/Arabidopsis
Relacje między gatunkowe/syntenia
• Syntenia = podobieństwo genów i ich ułożenia w
chromosomach
Porównanie rejonów ortologicznych obszaru adh w genomach sorgo i
kukurydzy (zacienienia= silne podobieństwo sekwencji)
Syntenia: Arabidopsis/ryż