Lab. 5. Wyznaczanie krzywej standardowej metodą real-time PCR. Analiza HRM (High
Resolution Melting).
Doświadczenie: Wyznaczanie krzywej standardowej dla reakcji PCR
Określanie liczby kopii matrycowego DNA metodą real-time PCR
Wykorzystanie DyNAmo™ SYBR® Green qPCR Kit do wrażliwego PCR ilościowego w
czasie rzeczywistym real-time PCR. Master miks zawiera zmodyfikowaną polimerazę DNA
Thermus brockianus i składniki niezbędne do qPCR. Trzeba dodać jedynie matrycę DNA i
startery. Krzywe amplifikacji poniżej pokazują na możliwości Kitu do detekcji produktów o
niskiej liczbie kopii i niskie wartości C(t)
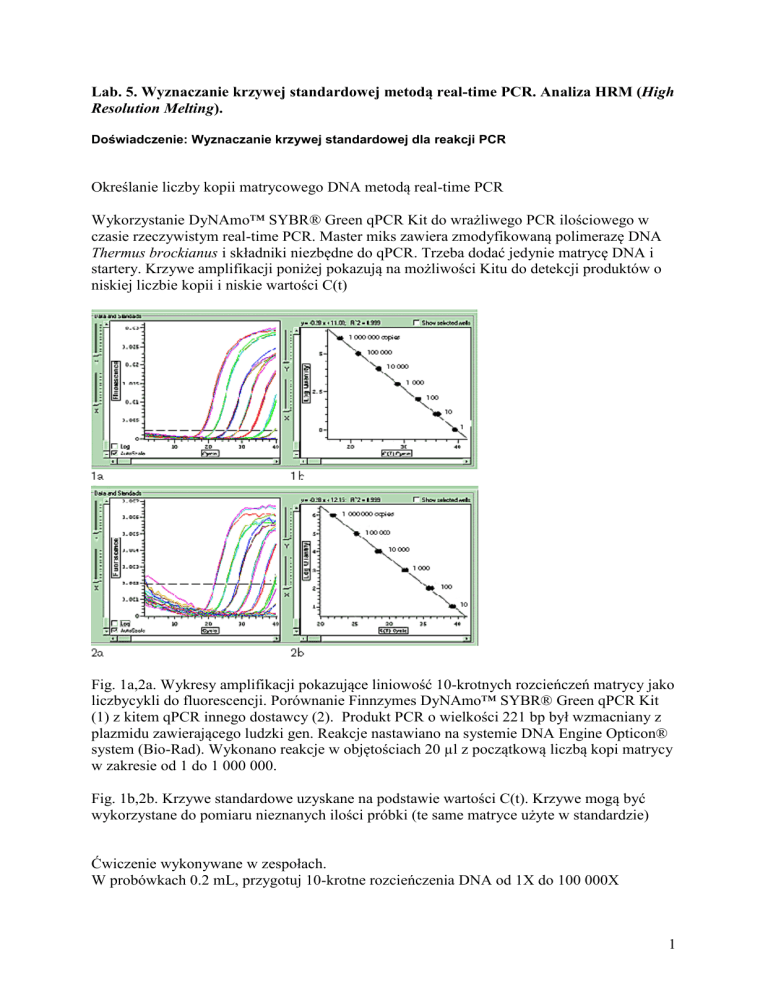
Fig. 1a,2a. Wykresy amplifikacji pokazujące liniowość 10-krotnych rozcieńczeń matrycy jako
liczbycykli do fluorescencji. Porównanie Finnzymes DyNAmo™ SYBR® Green qPCR Kit
(1) z kitem qPCR innego dostawcy (2). Produkt PCR o wielkości 221 bp był wzmacniany z
plazmidu zawierającego ludzki gen. Reakcje nastawiano na systemie DNA Engine Opticon®
system (Bio-Rad). Wykonano reakcje w objętościach 20 µl z początkową liczbą kopi matrycy
w zakresie od 1 do 1 000 000.
Fig. 1b,2b. Krzywe standardowe uzyskane na podstawie wartości C(t). Krzywe mogą być
wykorzystane do pomiaru nieznanych ilości próbki (te same matryce użyte w standardzie)
Ćwiczenie wykonywane w zespołach.
W probówkach 0.2 mL, przygotuj 10-krotne rozcieńczenia DNA od 1X do 100 000X
1
składnik
DNA (ze stoku)
bufor PCR (X)
MgCl2 (uM)
dNTP + dUTP (uM)
Thermus brockianus
DyNAmo
Master Mix SYBR Green I dye
Starter1-L (uM)
Starter1-R (uM)
Starter2-L (uM)
Starter2-R (uM)
woda
DyNAmo
vol
St0 StK
reakcji
10
20
2
1
20
5
1,5
2000
200
1
1
10
10
10
10
1
rozcien
20
20
20
20
20
0,2
0,2
0,2
0,2
0,1
0,06
0,1
ile 1X
1
5
0,02
0,02
0,02
0,02
6
0,5
0,5
0,5
0,5
4
DNA – wybrane stoki DNA z pszenicy zawierające translokację ‘pontin’ wykrywane za
pomocą starterów scs265 RiL + scs253 RiL
Układ doświadczalny: (seria 6 rozcieńczeń + kontrola negatywna) 3X do wyznaczenia
krzywej, 3-4 bibliotekix3powtórzenia
Kapa
PCR premix
Startery FiR
składnik
premix (X)
starter (X)
DNA ng/L
woda
St0
StK
2
10
1
1
vol reakcji
1
15
15
1
15
4
15
rozcien
ile 1X
7.5
1.5
4
2
95stC 5’; 35x(95stC 30”, 60stC 45”)65-95stC melt
Myco
Fermentas
Startery FiR
składnik
bufor (X)
starter (X)
MgCl2 (uM)
dNTP (mM)
TAQ U/L
DNA ng/L
evagreen
woda
St0
StK
10
10
25
2000
5
1
20
1
1
1
2,5
200
0,6
4
1
vol
reakcji
15
15
15
15
15
15
15
15
rozcien
ile 1X
Etapy analizy danych:
Wprowadzamy do arkusza Excela i postępujemy zgodnie z instrukcją
Startery PCR
Należy zaprojektować uważnie by zminimalizować niespecyficzne przyłączanie starterów i
tworzenie dimerów. Fluorescencja SYBR Green wzrasta po związaniu barwnika do
jakiegokolwiek dwuniciowego DNA. Tworzenie dimerów starterów i niespecyficzne wiązanie
może kształtować wydajność amplifikacji dla każdej stosowanej reakcji. Należy stosować się
do standardowych uwag w czasie projektowania starterów w celu uniknięcia tworzenia
dimerów starterów i struktur szpilki do włosów. Stężenie optymalne w reakcji 0,5µM (od 0.1
do 1 µM).
2
Traktowanie UNG (UDG)
Z powodu dużej wrażliwości qPCR nawet niewielkie ilości zanieczyszczającego DNA mogą
prowadzić do wyników fałszywie dodatnich. Jeśli dUTP jest stosowane we wszystkich
reakcjach qPCR, mżna zapobiegać przenoszeniu zanieczyszczeń dzięki traktowaniu próbek
UNG (N-glikozydaza urydynowa) przed PCR. Stosowany kit zawiera dUTP. UNG trawi
DNA zawierające dU i trawione DNA nie może działać jako matryca do qPCR. UNG jest
inaktywowana poprzez denaturację w PCR. Etap traktowania UNG (2 min w 50°C) nie ma
negatywnego wpływu na PCR przy wykorzystywaniu polimerazy DNA typu hot-start, która
nie ulega aktywacji w 50°C. W celu ograniczania ryzyka zanieczyszczenia ogólnie probówki
lub płytki zawierające produktu reakcji nie powinny być otwierane lub analizowane na żelu
agarozowym w tym samym laboratorium w którym nastawiane są reakcje.
Uruchomienie Eco
1. włącz netbook i czekaj aż Windows się w pełni uruchomi
2. zamknij klapę i włącz Eco
3. poczekaj 20 min, żeby dioda przestała migać; nie próbuj zaczynać analizy lub
zakładać płytki na blok do czasu gotowości aparatu.
4. uruchom program Eco i potwierdź komunikację z aparatem Eco
qPCR
Czystość kwasów nukleinowych jest ważna dla qPCR, zanieczyszczenia mogą niekorzystnie
wpływać na wykrywanie fluorescencji
Obliczanie ilości DNA
Obliczenie absolutnej ilości jest wykonywane poprzez nanoszenie próbek o nieznanym
stężeniu na krzywą standardową wyznaczoną na podstawie serii rozcieńczeń matrycy o
znanym stężeniu. Typowo krzywa standardowa jest wykresem cyklu krytycznego (threshold
cycle, C(t)) do logarytmu ilości DNA. Do określania ilości DNA w nieznanych próbkach
stosowana jest liniowa analiza regresji. Spadek równania jest związany z wydajnością reakcji
PCR. Wydajność reakcji PCR powinna być jednakowa dla standardu oraz mierzonych próbek.
Wydajność PCR dla próbek można ocenić wykonując serię rozcieńczeń.
Dla wykresu gdzie C(t) jest na osi y i log(DNA copy#) na osi x:
Wydajność PCR = ((10-1/nachylenie)-1)x 100%
Nachylenie wynoszące -3.322 odpowiada 100% wydajności
Dla wykresu gdzie log(DNA copy#) jest na osi Y a C(t) na osi x:
Wydajność PCR = = ((10-1xnachylenie)-1)x 100%
Nachylenie wynoszące -0.301 odpowiada 100% wydajności.
3
Obliczenie względnej ilości stosowane jest do określenia współczynnika pomiędzy ilością
docelowej cząsteczki a kalibratorem (kalibrator – zdrowa lub nietraktowana tkanka).
Najczęstszym zastosowaniem dla tej metody jest analiza ekspresji genu i porównanie
poziomu ekspresji genu w różnych próbkach. Ilość cząsteczki docelowej jest normalizowana
w odniesieniu do genu referencyjnego.
Jeśli wydajność amplifikacji genu referencyjnego jest taka sama jak wydajność enu
docelowego, do oznaczenia względnej ilości można stosować porównawczą metodę ∆∆C(t).
Zarówno dane dla badanej próbki jak i kalibratora są początkowo normalizowane w stosunku
do zmienności w ilości I jakości próbki. Znormalizowane wartości ∆C(t), są początkowo
obliczne z poniższych wzorów:
∆C(t)próbki = C(t)target - C(t)reference
∆C(t)kalibratora = C(t)target - C(t)reference
∆∆C(t) jest określana przy wykorzystaniu następującego wzoru:
∆∆C(t) = ∆C(t)próbki –∆C(t)kalibratora
Ekspresja docelowego genu znormalizowanego do genu referencyjnego względem kalibratora
= 2-∆∆C(t).
Wybrane zagadnienia z diagnostyki mikrobiologicznej człowieka
Borrelia burgdorferi
Wykrywane organizmy: B. afzelii, B. garini i B. burgdorferi sensu stricto sp. z "Borrelia
burgdorferi sensu lato sp." wywołują zakaźną chorobę wielonarządową boreliozę. Wektorem
przenoszącym chorobę jest kleszcz z rodzaju Ixodes.
Choroba przebiega w trzech etapach. Pierwszym etapem jest typowe wystąpienia rumienia
wędrującego, w drugim etapie choroba jest przenoszona z w krwioobiegu i może prowadzić
do zakażenia ośrodkowego układu nerwowego (aseptyczne zapalenie opon mózgowych....), w
trzecim etapie następuje upośledzenie funkcji stawów i niedowłady kończyn.
Diagnostyka boreliozy
Stosowane są metody pośrednie wykrywania przeciwciał surowicy IgM i IgG lub przeciwciał
wytwarzanych w rdzeniu kręgowym metodami ELISA lub immunoblotingu.
Ze względu na długotrwałe utrzymywanie się przeciwciał surowicy, stosunkowo wysoką
seroprewalencję (zdrowe osoby w populacji z wynikiem dodatnim na podstawie testów
serologicznych) u osób mieszkających w rejonach endemicznych, występowanie
seronegatywnych form boreliozy Lyme’a, możliwości występowania reakcji krzyżowych lub
brak możliwości zbadania materiałów klinicznych, po biopsji (skóry), płynu stawowego, itp.
wskazane jest uzupełnienie metod serologicznych.
Informacje o patogenie
Diagnostyka PCR jest jedyną klinicznie dostępną metodą detekcji bezpośredniej boreliozy i
doskonale nadaje się jako uzupełnienie dotychczasowego zakresu badań serologicznych z
4
każdego rodzaju materiału klinicznego. W pierwszym, ostrym stadium choroby, boreliozę
można wykryć w próbkach biopsji skóry a później, w fazie rozprowadzania z krwią w
próbkach krwi obwodowej i moczu. W późniejszych stadiach choroby metoda PCR może być
wykorzystana do wykrywania boreliozy w płynie mózgowo-rdzeniowym lub płynie
stawowym, a także w ciele szklistym oka. Próbki kliniczne do wykrywania B. burgdorferi
metodą PCR muszą być pobrane w odpowiednich stadiach choroby. Można również
profilaktycznie sprawdzić usuniętego kleszcza.
Metoda jakościowa (1)
Detekcja polega na amplifikacji sekwencji DNA genu kodującego flagelinę metodą nestedPCR w 1 probówce z polimerazą typu „hot start’ (pozwala na minimalizację reakcji
niespecyficznej) z rozdziałem na żelu agarozowym. W miksie reakcyjnym znajduje się
standard wewnętrzny w celu kontroli możliwej inhibicji reakcji PCR i glikozyalza uracylowa
DNA do kontroli zanieczyszczenia reakcji PCR produktami amplifikacji.
Pobieranie i przechowywanie próbek
Biopsja skóry z miejsc wysypki. Próbki powinny być umieszczone w probówkach "na
sucho" bez środków do transportu w temperaturze 4° C i przebadana w ciągu 24 godzin.
Próbka krwi obwodowej pobrana do EDTA i transportowana do laboratorium w
temperaturze +4° C w ciągu 24 godzin. Próbki płynu mózgowo-rdzeniowego i mazi
stawowej z chorych stawów, mocz należy pobrać próbki do probówek bez środka
transportującego i transportować w temperaturze +4° C w ciągu 24 godzin lub
długoterminowo zakonserwowane w temperaturze -20 do -80° C. Jeśli wymagane jest badanie
usuniętego kleszcza, należy go zachować w sterylnych warunkach, w temperaturze -20 do 80° C natychmiast po wyjęciu z rany i transportować do laboratorium jak najszybciej. W
przypadku dłuższego przechowywania wszystkie próbki powinny zostać zamrożone w
temperaturze -20° C.
Izolacja DNA
Do izolacji wykorzystywane są zestawy: PathogenFree DNA isolation kit (GeneProof);
QIAamp DNA Blood Mini Kit (QIAGEN); NucleoSpin Blood (Macherey-Nagel).
Amplifikacja PCR
Dodać 36 l MasterMix-u i 4 l izolowanego DNA lub 4 l kontroli pozytywnej do probówki
PCR do łącznej objętości 40 l. (próbki w temp 0-15°C). Amplifikować zgodnie z
warunkami:
dekontaminacja UDG: 37 °C/2 min;
denaturacja wstępna: 96 °C/10 min;
denaturacja 96 °C/10 sec., przyłączanie 68 °C/10 sec., wydłużanie 72 °C/40 sec.
liczba cykli 30
denaturacja 96 °C/10 sec., przyłączanie 54 °C/10 sec., wydłużanie 72 °C/30 sec.
liczba cykli 45
wydłużanie końcowe 72 °C/2 min.
Rozdział agarozowy
Produkty rozdzielić na 2% żelu agarozowym (5V/cm) z bromkiem etydyny (5 g/1 ml) i
obserwować w świetle UV. Ładować przynajmniej 10 l produktu amplifikacji.
Ocena detekcji
5
Wynik dodatni wykrywany jest produkt wielkości 276 bp, i produkt standardu wewnętrznego
420 bp (Fig. 1). Wynik ujemny – wykrywany jest jedynie produkt amplifikacji dla standardu
wewnętrznego 420 bp. W przypadku inhibicji PCR nie nastąpi amplifikacja żadnego z
produktów – badanie należy powtórzyć.
Fig. 1. Wykrywanie produktów na 2% żelu agarozowym. Fragmenty 276 bp pochodzą z
docelowego genu flageliny Borrelia burgdorferi sensu lato, fragment 420 bp pochodzi ze
standardu wewnętrznego. Ścieżka No. 1 – amplifikacja 320 krętków, Ścieżka No. 2 - 32
krętki, Ścieżka No. 3 - 3 krętki, Ścieżka No. 4 – 0.3 krętka/reakcję, M - 100 bp marker masy.
Liczba kopii “Pn” wzmacnianej sekwencji po “n” cyklach PCR wynosi:
Pn = Po (1+ E)n,
gdzie Po jest początkową liczbą kopii sekwencji docelowej a E jest wydajnością amplifikacji
(0 ≤ E ≤ 1). Na żelach agarozowych barwionych bromkiem etydyny można wykryć 25
ng DNA, co odpowiada 1011 kopi fragmentu 200 pz; obliczenia teoretyczne pokazały,
że rozpoczynając od 10 cząsteczek matrycy na początku reakcji PCR, żeby produkt
był możliwy do detekcji po 35 cyklach ogólna wydajność reakcji powinna wynosić
ponad 0.9.
http://www.geneproof.com
Mem Inst Oswaldo Cruz, Rio de Janeiro, Vol. 97(6): 897-900, September 2002
Przykładowe startery
gatunek
B.afzelii
Sekwencje starterów (5'
3')
Pozycjea
Temperatura przyłączania
(°C)
42
GCATGCAAGTCAAACGGA
59-76
ATATAGTTTCCAACATAGC
648-630
B.garinii
GGGATGTAGCAATACATCT
74-92 44
ATATAGTTTCCAACATAGT
648-630
B.burgdorferi GGGATGTAGCAATACATTC
74-92 48
ATATAGTTTCCAACATAGG
648-630
VS116
GCAAGTCAAACGGGATGTAGT
63-83 52
GTATTTTATGCATAGACTTATATG 612-589
a
In the 16S rRNA sequence of B. burgdorferi sensu stricto (in nucleotides)
Journal of Clinical Microbiology, November 1998, p. 3355-3358, Vol. 36, No. 11
6
Metoda ilościowa (2)
wariant A) ze standardem wewnętrznym w MasterMixie PCR Kit ISIN - zestaw pozwala na
kontrolę inhibicji reakcji PCR
wariant B) ze standardem wewnętrznym oddzielnie PCR Kit ISEX - zestaw pozwala na
kontrolę inhibicji reakcji PCR i kontrolę wydajności izolacji DNA. Standard wewnętrzny
jest dodawany do próbki przeznaczonej do izolacji DNA tak, żeby w objętości końcowej było
w proporcji 1 µl w tym 0.1 µl standardu wewnętrznego
7
Amplifikacja PCR
1. Dodać 30 l MasterMix i 10 l izolowanego DNA lub 10 l kontroli pozytywnej do
probówki PCR, do końcowej objętości 40 l.
2. Zamknąć probówki, krótko zwirować i umieścić w urządzeniu do real-time PCR. Warunki
amplifikacji:
dekontaminacja UDG: 37 °C/2 min;
denaturacja wstępna: 96 °C/10 min;
denaturacja 96 °C/5 sec., przyłączanie 60 °C/40 sec. – czytanie sygnału fluorescencji,
wydłużanie 72 °C/20 sec.
liczba cykli 45
Ocena ilościowa obecności patogena
8
Wykrywanie gatunków z rodzaju Mycoplasma
Zasada wykrywania
Zestaw zaprojektowano do wykrywania genomowego DNA gatunków z rodzaju Mycoplasma
przy zastosowaniu reakcji łańcuchowej polimerazy (PCR). Metoda ta bazuje na wykrywaniu
wielokrotnych kopii sekwencji genu kodującego bakteryjną podjednostkę 16S RNA,
specyficzną dla gatunków Mycoplasma spp. Czułość zestawu do detekcji PCR pozwala na
wykrywanie pojedynczych kopii genomowego DNA Mycoplasma spp. w reakcji. Zapewnia
to czułe i uniwersalne wykrywanie wszystkich klinicznie istotnych ludzkich mykoplazm i
ureaplazm włączając w to M. pneumoniae, M. hominis, M. genitalium, U. urealitica i M.
fermentans. Zestaw pozwala na wykrywanie wszystkich szczepów Mycoplasma
zanieczyszczających kultury tkankowe i roztwory do kultur tkankowych (e.g. kultury
wirusów, BSA) i może służyć do kontroli zanieczyszczeń produktów przygotowywanych z
kultur tkankowych (np. szczepionki).
Metoda pozwala na wykrywanie inhibicji reakcji i i dostarcza poprawnych warunków nawet
w obecności inhibitorów reakcji PCR. Metoda pozwala na wykrywanie wszystkich znanych
gatunków Mycoplasma (National Center for Biotechnology Information U.S.; 2006) i jest
przystosowana do detekcji następujących szczepów o znaczeniu przemysłowym lub
weterynaryjnym: Acholeplasma axanthum, Mycoplasma alvi, Mycoplasma arginini,
Mycoplasma buccale, Mycoplasma cavipharyngis, Mycoplasma cloacale, Mycoplasma
fastidiosum, M. fermentans, Mycoplasma genitalium, Mycoplasma hominis, Mycoplasma
salivarium, Mycoplasma pulmonis, M. orale, Mycoplasma penetrans, M.pirum, M.
pneumoniae, Ureaplasma diversum, Ureaplasma parvum, Ureaplasma urealyticum,
Acholeplasma sp., etc. Elementy zestawu: polimeraza “hot start” standard wewnętrzny do
kontroli inhibicji reakcji PCR, glukozylazę uracylową DNA (UDG).
Przebieg oznaczenia
Pobieranie i przechowywanie próbek
Próbki płynów ustrojowych, krwi, wymazy z pochwy i cewki moczowej, krwi obwodowej,
itd. Próbki (za wyjątkiem krwi) powinny być pobrane do sterylnych probówek bez żadnych
środków do transportu, transportowane w ciągu 12 godzin w +4°C. Pobieranie krwi na EDTA
i transport w +4°C. Badanie nasienia jak u Chlamydia (przynajmniej 200µl wydzieliny,
transportowane w ciągu 24 godzin w +4°C. lub przechowywanej poniżej min. -20°C do czasu
analizy).
Izolacja DNA
jak w Borrelia burgdorferi
Amplifikacja PCR
Dodać 36 l MasterMix-u i 4 l izolowanego DNA lub 4 l kontroli pozytywnej do probówki
PCR do łącznej objętości 40 l. (próbki w temp 0-15°C). Amplifikować zgodnie z
warunkami:
dekontaminacja UDG: 37 °C/2 min;
denaturacja wstępna: 96 °C/15 min;
denaturacja 96 °C/20 sec., przyłączanie
liczba cykli 45
wydłużanie końcowe 72 °C/2 min.
62 °C/20 sec., wydłużanie
72 °C/40 sec.
9
Rozdział agarozowy
Produkty rozdzielić na 2% żelu agarozowym (5V/cm) z bromkiem etydyny (5 g/1 ml) i
obserwować w świetle UV. Ładować przynajmniej 10 l produktu amplifikacji.
Ocena detekcji
Wynik dodatni wykrywany jest produkt wielkości 270 bp i produkt standardu wewnętrznego
603 bp (Fig. 2). Wynik ujemny – wykrywany jest jedynie produkt amplifikacji dla standardu
wewnętrznego 603 bp. W przypadku inhibicji PCR nie nastąpi amplifikacja żadnego z
produktów – badanie należy powtórzyć.
Fig. 2. Wykrywanie produktów na 2% żelu agarozowym. Fragmenty 270 bp pochodzą z
docelowego genu 16S rDNA gatunków Mycoplasma, fragment 603 bp pochodzi ze standardu
wewnętrznego. Ścieżki No. 1-5 – wyniki dodatnie wykrywania Mycoplasma, No. 6-9 –
wyniki negatywne, M - 100 bp marker masy.
Zawartość MasterMixu:
Chlorek magnezu bezwodny
Chlorek potasu
siarczan amonu
trihydroksymetyloaminometan (TRIS)
kwas deoksyrybonukleinowy
dATP, dUTP, dGTP, dCTP,
polimeraza Taq,
glicerol,
woda
Kontrola dodatnia
kwas deoksyrybonukleinowy
trihydroksymetyloaminometan
kwas etylenodiamino trichlorooctowy (Neutrol TE)
woda
10
ĆWICZENIE PRAKTYCZNE:
Wykrywanie Mycoplasma z wymazów śluzówki
Test można stosować do detekcji mykoplazm w kulturach tkankowych
Przygotuj PCR w grupach
Startery:
Myco_L (GPO-3) gggAgCAAACAggATTAgATACCCT
Myco_R (MGSO) TgCACCATCTgTCACTCTgTTAACCTC
Myco
Fermentas
Myco_L
Myco_R
składnik
bufor (X)
starter1 (M)
starter1 (M)
MgCl2 (uM)
dNTP (mM)
spermidyna (mM)
TAQ U/L
DNA ng/L
woda
St0
vol
reakcji
StK
10
10
10
25
2000
10
1
10
1
1
0,4
0,4
2,5
200
0,4
0,6
25
rozcien
ile 1X
25
25
25
25
25
25
25
25
25
Cykl:
touchdown: SSR55B
Rozdziel produkty na 2% żelu agarozowym.
Produkt o wielkości 270 bp jest wykrywany na żelu agarozowym 2% barwieniem EtBr.
Biotynylowana sonda GPO-4, 59-CTTAAAGGAATTGACGGGAACCCG-39 może być
wykorzystywana do hybrydyzacji z produktem PCR w celu potwierdzenia identyczności
sekwencji.
Ossewaarde J.M., De Vries A., Bestebroer T., Angulo A.F. 1996. Application of a
Mycoplasma Group-Specific PCR for Monitoring Decontamination of Mycoplasma-Infected
Chlamydia sp. Strains. Applied And Environmental Microbiology, 328–331
11










