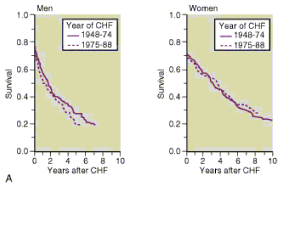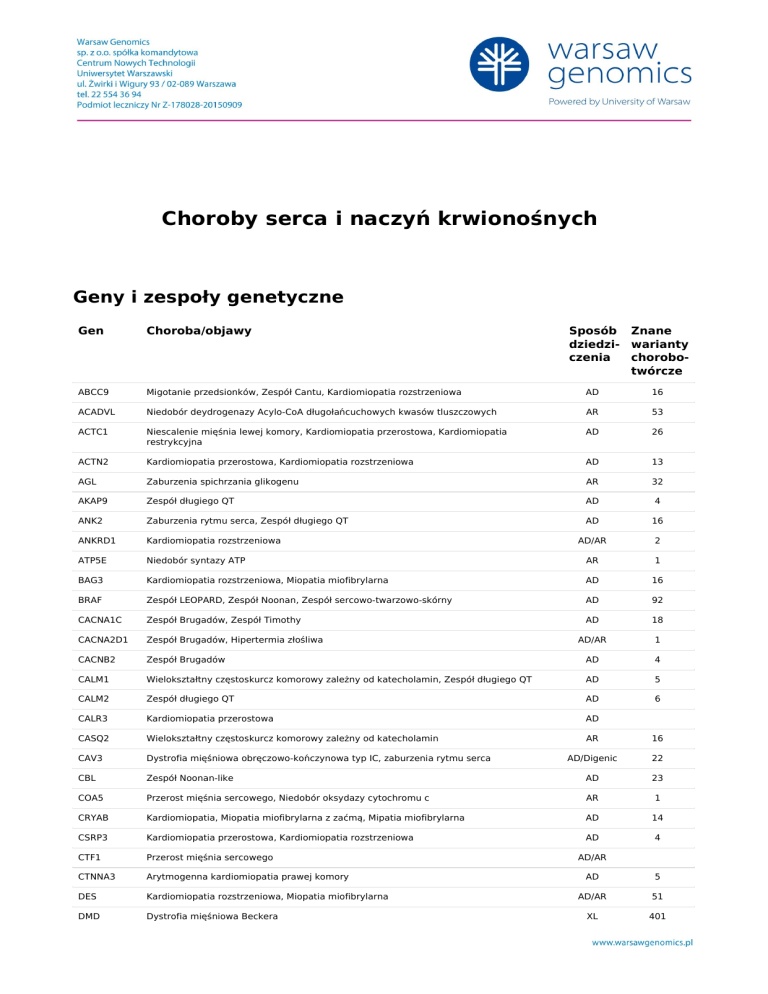
Choroby serca i naczyń krwionośnych
Geny i zespoły genetyczne
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
ABCC9
Migotanie przedsionków, Zespół Cantu, Kardiomiopatia rozstrzeniowa
AD
16
ACADVL
Niedobór deydrogenazy Acylo-CoA długołańcuchowych kwasów tluszczowych
AR
53
ACTC1
Niescalenie mięśnia lewej komory, Kardiomiopatia przerostowa, Kardiomiopatia
restrykcyjna
AD
26
ACTN2
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa
AD
13
AGL
Zaburzenia spichrzania glikogenu
AR
32
AKAP9
Zespół długiego QT
AD
4
ANK2
Zaburzenia rytmu serca, Zespół długiego QT
AD
16
ANKRD1
Kardiomiopatia rozstrzeniowa
AD/AR
2
ATP5E
Niedobór syntazy ATP
AR
1
BAG3
Kardiomiopatia rozstrzeniowa, Miopatia miofibrylarna
AD
16
BRAF
Zespół LEOPARD, Zespół Noonan, Zespół sercowo-twarzowo-skórny
AD
92
CACNA1C
Zespół Brugadów, Zespół Timothy
AD
18
CACNA2D1
Zespół Brugadów, Hipertermia złośliwa
AD/AR
1
CACNB2
Zespół Brugadów
AD
4
CALM1
Wielokształtny częstoskurcz komorowy zależny od katecholamin, Zespół długiego QT
AD
5
CALM2
Zespół długiego QT
AD
6
CALR3
Kardiomiopatia przerostowa
AD
CASQ2
Wielokształtny częstoskurcz komorowy zależny od katecholamin
AR
16
CAV3
Dystrofia mięśniowa obręczowo-kończynowa typ IC, zaburzenia rytmu serca
AD/Digenic
22
CBL
Zespół Noonan-like
AD
23
COA5
Przerost mięśnia sercowego, Niedobór oksydazy cytochromu c
AR
1
CRYAB
Kardiomiopatia, Miopatia miofibrylarna z zaćmą, Mipatia miofibrylarna
AD
14
CSRP3
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa
AD
4
CTF1
Przerost mięśnia sercowego
CTNNA3
Arytmogenna kardiomiopatia prawej komory
DES
Kardiomiopatia rozstrzeniowa, Miopatia miofibrylarna
DMD
Dystrofia mięśniowa Beckera
AD/AR
AD
5
AD/AR
51
XL
401
Gen
Choroba/objawy
DMPK
Dystrofia miotoniczna typ 1
DNAJC19
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
AD/AR
139
Kwasica 3-metyloglutakonowa
AR
3
DNM1L
Przerost mięśnia sercowego
AD
3
DOLK
Zaburzenia glikozylacji
AR
7
DPP6
Zaburzenia rytmu serca
AD
6
DSC2
Arytmogenna kardiomiopatia prawej komory
AD/AR
20
DSG2
Arytmogenna kardiomiopatia prawej komory, Kardiomiopatia rozstrzeniowa
AD
36
DSP
Arytmogenna kardiomiopatia prawej komory, Kardiomiopatia rozstrzeniowa
AD/AR
99
DTNA
Przerost mięśnia sercowego
AD
2
EMD
Dystrofia Emery'ego-Dreiffusa
XL
22
EYA4
Kardiomiopatia rozstrzeniowa
AD
6
FHL1
Myopathy with postural muscle atrophy, Emery-Dreifuss muscular dystrophy, Reducing
bod myopathy
XL
18
FHL2
Przerost mięśnia sercowego
AD/AR
FKTN
Muscular dystrophy-dystroglycanopathy, Kardiomiopatia rozstrzeniowa, Muscular
dystrophy-dystroglycanopathy (limb-girdle)
AD/AR
23
FOXRED1
Leigh syndrome, Mitochondrial complex I deficiency
AR
10
FXN
Ataksja Friedreicha
AR
9
GAA
Glycogen storage disease
AR
61
GATAD1
Kardiomiopatia rozstrzeniowa
AR
4
GLA
Fabry disease
XL
372
GLB1
GM1-gangliosidosis, Mucopolysaccharidosis (Morquio syndrome)
AR
52
GPD1L
Zaburzenia rytmu serca
AD
3
GUSB
Mucopolysaccharidosis
AR
23
HCN4
zespół chorej zatoki, Zespół Brugadów
AD
9
HFE
Hemochromatosis, choroba Alzheimera, postać późna
AR/Digenic
7
HRAS
Zespół Costello, Miopatia z nadmiarem wrzecionek mięśniowych, Neuroblastoma, Rak
pęcherza moczowego
AD
29
ILK
Przerost mięśnia sercowego
AD/AR
JPH2
Kardiomiopatia przerostowa
AD
5
JUP
Arytmogenna kardiomiopatia prawej komory
AD/AR
12
KCND3
Zespół Brugadów
AD
6
KCNE1
Zespół długiego QT, Zespół Jervella i Lange-Nielsena
AD/AR/Dig
enic
20
KCNE1L
Zaburzenia rytmu serca
KCNE2
Zespół długiego QT, Migotanie przedsionków
AD
14
KCNE3
Zespół Brugadów
AD
1
KCNH2
Zespół krótkiego QT, Zespół długiego QT
AD/AR
1
AD/AR
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
KCNJ2
Zespół krótkiego QT, Zespół Andersena syndrome, Zespół długiego QT, Migotanie
przedsionków
AD
64
KCNJ5
Zespół długiego QT, Hiperaldosteronizm
AD
8
KCNJ8
Zaburzenia rytmu serca
AD/AR
1
KCNQ1
Zespół krótkiego QT, Zespół długiego QT, Migotanie przedsionków, Zespół Jervella i LangeNielsena
AD/AR/Dig
enic
400
KRAS
Zespół Noonan, Zespół sercowo-twarzowo-skórny
AD
45
LAMA4
Przerost mięśnia sercowego
AD
5
LAMP2
Choroba Danona
XL
46
LDB3
Kardiomiopatia rozstrzeniowa, Miopatia miofibrylarna
AD
10
LMNA
Zespół serce-ręka, Dystrofia kończynowo-obręczowa, Lipodystrofia, Dystrofia Emery'egoDreiffusa, Kardiomiopatia rozstrzeniowa, Progeria Hutchinsona-Gilforda
AD/AR
170
MAP2K1
Zespół sercowo-twarzowo-skórny
AD
14
MAP2K2
Zespół sercowo-twarzowo-skórny
AD
14
MIB1
Przerost mięśnia sercowego
AD
2
MRPL3
Przerost mięśnia sercowego
AR
1
MYBPC3
Niescalenie mięśnia lewej komory, Kardiomiopatia przerostowa, Kardiomiopatia
rozstrzeniowa
AD/AR
408
MYH6
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa, Miopatia
AD
8
MYH7
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa, Miopatia
AD/AR
320
MYL2
Kardiomiopatia przerostowa
AD
23
MYL3
Kardiomiopatia przerostowa
AD/AR
14
MYLK2
Przerost mięśnia sercowego
AD/Digenic
3
MYOM1
Przerost mięśnia sercowego
AD/AR
2
MYOZ2
Przerost mięśnia sercowego
AD
1
MYPN
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa, Kardiomiopatia restrykcyjna
AD
7
NEBL
Przerost mięśnia sercowego
NEXN
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa
NOS1AP
Zespół Romano-Ward
NRAS
Zespół Noonan
PDLIM3
Przerost mięśnia sercowego
PKP2
Arytmogenna kardiomiopatia prawej komory
PLN
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa
PRKAG2
AD/AR
AD
6
AD/AR
AD
16
AD/AR
AD
93
AD/AR
8
Kardiomiopatia przerostowa, Zespół Wolffa-Parkinsona-White'a
AD
17
PSEN1
Kardiomiopatia rozstrzeniowa
AD
46
PSEN2
Kardiomiopatia rozstrzeniowa, Kardiomiopatia związana z ciążą i połogiem
AD
7
PTPN11
Zespół Noonan, Zespół LEOPARD
AD
120
RAF1
Zespół Noonan, Zespół LEOPARD, Kardiomiopatia rozstrzeniowa
AD
32
RANGRF
Zaburzenia rytmu serca
AD/AR
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
RBM20
Kardiomiopatia rozstrzeniowa
AD
13
RYR2
Arytmogenna kardiomiopatia prawej komory, Wielokształtny częstoskurcz komorowy
zależny od katecholamin
AD
123
SCN1B
Zespół Brugadów, Padaczka uogólniona z napadami gorączkowymi typu 1
AD
12
SCN2B
Zespół Brugadów
AD
2
SCN3B
Rodzinne migotanie przedsionków, Zespół Brugadów
AD
4
SCN4B
Zespół długiego QT
AD
4
SCN5A
Zespół Brugadów, Zespół długiego QT, Kardiomiopatia rozstrzeniowa, Rodzinny
postępujacy blok serca,
AD/AR/Dig
enic
517
SCO2
Zespół Leigh, Kardiomiopatia przerostowa, Myopia
AD/AR
18
SDHA
Guzy stromalne przewodu pokarmowego, Zespół Leigh, Paraganglioma, Kardiomiopatia
rozstrzeniowa
AD/AR
14
SGCD
Kardiomiopatia rozstrzeniowa, Dystrofia kończynowo-obręczowa
AR
10
SHOC2
Zespół Noonan-like
AD
1
SLC25A3
Niedobór mitochondrialnych przenośników grup fosforanowych
AR
2
SLMAP
Zespół Brugadów
SNTA1
Zespół długiego QT
AD
2
SOS1
Zespół Noonan
AD
39
SPRED1
Zespół Legiusa
AD
11
SYNE1
Przerost mięśnia sercowego
AD/AR
22
SYNE2
Przerost mięśnia sercowego
AD
2
TAZ
Kwasica 3 metylo-glutarowa (Zespół Bartha)
XL
32
TCAP
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa, Dystrofia kończynowoobręczowa
AD/AR
9
TGFB3
Zespół Loyesa-Dietza, Arytmogenna kardiomiopatia prawej komory
AD
8
TMEM43
Arytmogenna kardiomiopatia prawej komory
AD
5
TMEM70
Niedobór mitochondrialnego kompleksu V
AR
8
TMPO
Przerost mięśnia sercowego
AD
TNNC1
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa
AD
12
TNNI3
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa
AD/AR
58
TNNT2
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa, Niescalenie mięśnia lewej
komory
AD
60
TPM1
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa
AD
37
TRDN
Wielokształtny częstoskurcz komorowy zależny od katecholamin
AR
3
TRIM63
Przerost mięśnia sercowego
TRPM4
Postępujący rodzinny blok serca
AD
4
TSFM
Złożone zaburzenia fosforylacji oksydacyjnej
AR
5
TTN
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa
AD
385
TTR
Hipertyroksynemia związana z zaburzeniami transttyretyny
AD
51
AD/AR
AD/AR
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
TXNRD2
Przerost mięśnia sercowego
VCL
Kardiomiopatia przerostowa, Kardiomiopatia rozstrzeniowa
AD
12
XK
Zespół McLeoda
XL
9
AD/AR
Metodologia
Informacja na temat metody badania:
W pierwszej kolejności, z pobranej próbki krwi lub z bloczka parafinowego izolowany jest kwas
deoksyrybonukleinowy (DNA), którego jakość i ilość jest określana w analizie
spektrofotometrycznej i fluorymetrycznej. Po mechanicznej lub enzymatycznej fragmentacji,
DNA jest wykorzystywany do stworzenia biblioteki, umożliwiającej oznaczenie, a następnie
zsekwencjonowanie i analizę genów, które zostały wybrane w ramach zleconego panelu.
Otrzymana biblioteka jest sekwencjonowana na sekwenatorze nowej generacji. Otrzymane
wyniki zostają następnie poddane analizie bioinformatycznej i interpretacji klinicznej. Warianty
genetyczne są identyfikowane z wykorzystaniem Burrows-Wheeler Aligner. Test umożliwia
wykrycie 100% substytucji i 95% małych insercji i delecji.
Informacja na temat klasyfikacji wariantów:
W raporcie z badania przedstawiana jest informacja na temat wariantów zaklasyfikowanych
jako warianty „potencjalnie patogenne” i „patogenne”, z uwagi na ich potencjalne znaczenie
kliniczne. Zidentyfikowane warianty są klasyfikowane do następujących kategorii:
Wariant patogenny: znaleziona zmiana w sekwencji genu ma bezpośredni związek
z powstawaniem choroby. Równocześnie, niektóre zmiany patogenne mogą nie mieć pełnej
penetracji, tj. pojedyncza zmiana może być niewystarczająca do wywołania pełnoobjawowej
choroby.
Wariant potencjalnie patogenny: znaleziona zmiana w sekwencji genu jest z dużym
prawdopodobieństwem związana z powstawaniem choroby, jednakże udowodnienie tego
związku nie jest możliwe w oparciu o aktualnie dostępne dane naukowe. Potwierdzenie
patogenności wariantu wymaga dodatkowych badań i dowodów; nie można wykluczyć, że
dalsze badania wykażą, że znaleziona zmiana ma niewielkie lub żadne znaczenie kliniczne.
Wariant o nieznanej patogenności: w oparciu o aktualnie dostępne dane naukowe nie ma
możliwości określenia znaczenia znalezionej zmiany.
Wariant potencjalnie łagodny: znaleziona zmiana w sekwencji genu najprawdopodobniej nie
ma związku z powstawaniem choroby, jednakże w oparciu o aktualnie dostępne dane naukowe
nie ma możliwości potwierdzenia łagodności zmiany. Potwierdzenie klinicznego znaczenia
wariantu wymaga dodatkowych badań i dowodów; nie można wykluczyć, że dalsze badania
wykażą, że znaleziona zmiana ma znaczenie kliniczne i prowadzi do rozwinięcia choroby
Wariant łagodny: znaleziona zmiana nie ma związku z powstawaniem choroby
Zidentyfikowane warianty genetyczne klasyfikowane są w oparciu o wytyczne opracowane
przez American College of Medical Genetics and Genomics i American Association for Molecular
Pathology (S. Richards, Genet Med. 2015 May;17(5):405-24). W klasyfikacji wariantów brane są
pod uwagę następujące kryteria:
wcześniejsza identyfikacja wariantu u osób obciążonych chorobą
wpływ wariantu na powstawanie funkcjonalnego produktu genu:
określony w analizach bioinformatycznych
potwierdzony w badaniach in vitro/in vivo
lokalizacja wariantu (ekson/intron, domena funkcjonalna)
zmiana de novo/dziedziczna
częstość występowania wariantu w populacji ogólnej (każdy wariant występujący
z częstością >5% zgodnie z Exome Sequencing Project, 1000 Genomes Project lub
Exome Aggregation Consortium jest klasyfikowany jako zmiana łagodna)
częstość występowania wariantu w populacji ogólnej w stosunku do populacji osób
chorych
Ostateczna klasyfikacja wariantów prowadzona jest w oparciu o sumę wymienionych kryteriów.
Przeszukiwane bazy danych obejmują: 1000GP, ClinVar, ConsensusPathDB, Exome Aggregation
Consortium, Exome Variant Server, FATHMM, GO (Gene Ontology), GTEx (Genotype-Tissue
Expression), GWAS (Genome Wide Association Study), HGMD, KEGG, MetaLR, MetaSVM,
MutationAssessor, MutationTaster, OMIM, PolyPhen-2, PROVEAN, SIFT, SnpEff, dbNSFP, UniProt,
VEP (Variant Effect Predictor).
Ograniczenia badania:
Wszystkie technologie sekwencjonowania mają swoje ograniczenia. Zlecane badanie jest
wykonywane z wykorzystaniem sekwencjonowania nowej generacji (NGS) i ma na celu
zbadanie regionów kodujących i splicingowych zleconych genów. Chociaż stosowane techniki
sekwencjonowania oraz późniejsze analizy bioinformatyczne są ukierunkowane na ograniczenie
znaczenia sekwencji pseudogenów, to jednak obecność wysoce homologicznych sekwencji
genowych może nadal sporadycznie zakłócać zdolność identyfikacji patogennych alleli, jak
i delecji/duplikacji. Sekwencjonowanie Sangera jest metodą wykorzystywaną do potwierdzania
wariantów, które uzyskały niższe parametry jakości. Analizy delecji/duplikacji wskazują na
zmiany ilościowe DNA obejmujące minimum jeden ekson i zawsze wymagają potwierdzenia
innymi metodami (qPCR lub MLPA). Wykonane analizy nie są przeznaczone do wykrywania
pewnych typów zmian genomowych, jak translokacje, inwersje, mutacje dynamiczne (np.
zwiększenie ilości powtórzeń trzynukleotydowych), zmian w regionach regulatorowych czy
intronowych. Jeśli raportowane jest zwiększenie liczby powtórzeń dwu- czy trzynukleotydowych,
to trzeba założyć, że dokładna liczba powtórzeń nie jest precyzyjna. Przeprowadzane badanie
nie jest przeznaczone do wykrywania mozaikowatości somatycznych, a analizy mutacji
somatycznych powinny być prowadzone w kontekście sekwencji DNA germinalnego.
Nie ma możliwości wykluczenia obecności mutacji w genach i rejonach innych niż objęte
wykonywanym badaniem, a także zmian liczby kopii genu. Raport z badania zawiera informację
na temat zmian w sekwencji genów zidentyfikowanych w oparciu o porównanie z aktualnymi
sekwencjami referencyjnymi zdeponowanymi w bazach danych NCBI Nucleotide i Ensembl.
Testy są opracowywane w Warsaw Genomics do celów klinicznych. Wszystkie otrzymywane
wyniki badań są interpretowane i analizowane przez ekspertów naukowych i medycznych
Warsaw Genomics.
Jak zlecić badanie
Informacja na temat metody badania:
Badanie można zlecić bezpośrednio na stronie internetowej Warsaw Genomics, poprzez
zaznaczenie wybranego testu. Zalecamy jednak, by przed każdym badaniem skonsultować się
z lekarzem, który pomoże w wybraniu odpowiedniego testu diagnostycznego, wyjaśni
możliwości i ograniczenia testów genetycznych a także przedstawi możliwe wyniki
i konsekwencje przeprowadzenia badania.
Czas realizacji badania: od 4 do 10 tygodni. Będziemy informować o kolejnych etapach analizy.
KONSULTACJA LEKARSKA
REJESTRACJA
Wybranie właściwego testu
genetycznego lub
indywidualnego zestawu
genów
Wypełnienie formularza
zlecenia testu.
Formularz wypełnia pacjent
lub wybrany przez niego
lekarz.
OPŁACENIE TESTU
Po wykonaniu testu
POBRANIE KRWI
Łącznie należy pobrać 4ml
krwi do jednej probówki
z EDTA (takiej jak na
morfologie). Pobraną krew
można przechowywać
w lodówce (w temp. +4st C)
do 7 dni
WYNIK BADANIA
zostanie przekazany osobie
zlecającej test – pacjentowi
lub wybranemu przez niego
lekarzowi
Jak przekazać materiał do badania?
Badanie genetyczne z krwi:
PRZESŁANIE PRÓBKI
KRWI NA NASZ ADRES
Próbkę można dostarczyć
osobiście lub kurierem
(w temperaturze pokojowej)
w ciągu 48 godzin.
Szczegółowa instrukcja
pakowania próbki
i zamówienia kuriera jest
dostępna tutaj. Do próbki
dołączamy wydrukowany
i podpisany formularz
zlecenia testu.
KONSULTACJA LEKARSKA
1. Krew należy pobrać do jednej probówki z EDTA (nie pobierać do probówek na skrzep,
ani na heparynę litową). Pobranie krwi może nastąpić w dowolnej godzinie, pacjent nie
musi być na czczo:
osoba dorosła - ok. 4 ml krwi żylnej do izolacji DNA (pobraną krew należy
dokładnie wymieszać z antykoagulantem i przechowywać w temperaturze 4°C)
dzieci – ok. 4 ml (minimalna ilość 2 ml) krwi żylnej do izolacji DNA (pobraną
krew należy dokładnie wymieszać z antykoagulantem i przechowywać
w temperaturze 4°C)
niemowlę – ok. 1,5 - 2 ml krwi żylnej do izolacji DNA (pobraną krew należy
dokładnie wymieszać z antykoagulantem i przechowywać w temperaturze 4°C)
2. Probówkę należy opisać imieniem i nazwiskiem i zabezpieczyć (można zakleić taśmą
klejącą).
3. Zabezpieczoną probówkę wraz z wypełnionym i podpisanym formularzem zlecenia
badania należy zapakować i wysłać zgodnie z instrukcją dostępną pod adresem:
https://badamygeny.pl/BADAMY_GENY/docs/instrukcja-wysylki-probki-krwi.pdf
Badanie genetyczne z bloczka parafinowego (profilowanie
nowotworu):
1. Należy pobrać łącznie 4 ml krwi do jednej probówki z EDTA (nie pobierać do probówek
na skrzep, ani na heparynę litową). Pobranie krwi może nastąpić w dowolnej godzinie,
pacjent nie musi być na czczo. Pobraną krew należy dokładnie wymieszać
z antykoagulantem i przechowywać w temperaturze 4°C.
2. Probówkę należy opisać imieniem i nazwiskiem i zabezpieczyć (można zakleić taśmą
klejącą).
3. Uzyskanie tkanki nowotworowej do badania w postaci:
bloczka parafinowego zawierającego wycinek nowotworu wraz z uzyskanym
z bloczka preparatem histopatologicznym (szkiełkiem) umożliwiającym
zlokalizowanie fragmentu tkanki nowotworowej,
albo wycinka tkanki nowotworowej z bloczka parafinowego o wymiarach min.
4x4x1mm, zawierającego wyłącznie tkankę nowotworową.
4. Próbkę należy opisać imieniem i nazwiskiem i zabezpieczyć (można zakleić taśmą
klejącą).
5. Zabezpieczony materiał wraz z wypełnionym i podpisanym formularzem zlecenia
badania należy zapakować i wysłać zgodnie z instrukcją dostępną pod adresem:
https://badamygeny.pl/BADAMY_GENY/docs/instrukcja-wysylki-probki-krwi.pdf
Powered by TCPDF (www.tcpdf.org)