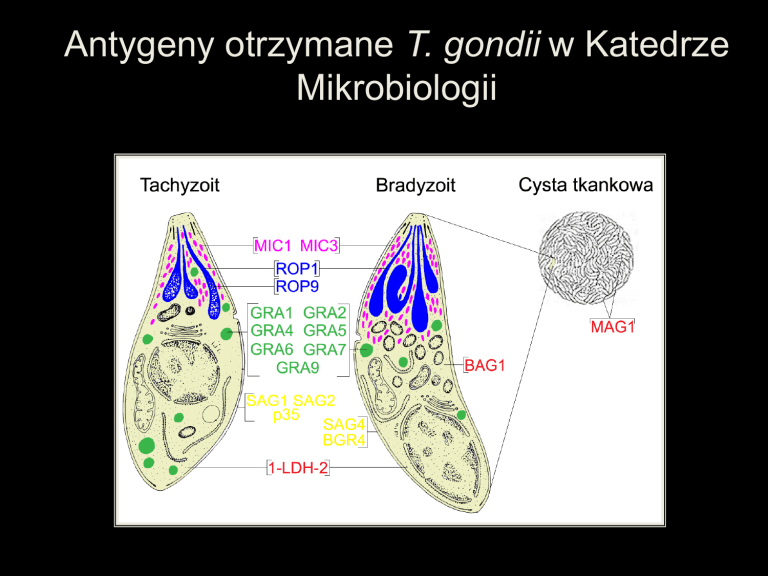
Antygeny otrzymane T. gondii w Katedrze
Mikrobiologii
Diagnostyka toksoplazmozy
Metody diagnostyczne powinny:
potwierdzić lub wykluczyć zarażenie T. gondii;
określić fazę zarażenia (toksoplazmoza wczesna, przewlekła);
u kobiet w ciąży z pierwotną toksoplazmozą potwierdzić lub wykluczyć
zarażenie płodu lub noworodka
METODY
BEZPOŚREDNIE
POŚREDNIE
(mają na celu izolację
pasożyta lub wykrycie jego
antygenu)
(wykorzystują właściwości
immunogenne pasożyta)
Testy serologiczne stosowane w
diagnostyce toksoplazmozy
Test barwny (antygen żywy / IgG)
Immunofluorescencja pośrednia (antygen utrwalony formaliną / IgG, IgM)
Aglutynacja bezpośrednia (antygen utrwalony formaliną / IgG + IgM)
Test ISAGA (antygen utrwalony formaliną / IgM, IgA, IgE)
Odczyn wiązania dopełniacza (antygen cytoplazmatyczny / IgG + IgM)
Test hemaglutynacji (antygen cytoplazmatyczny + błonowy / IgG + IgM)
Test ELIFA (antygen cytoplazmatyczny / IgG, IgM, IgA, IgE)
Awidność przeciwciał (antygen cytoplazmatyczny + błonowy / IgG)
Test ELISA:
pośredni (antygen cytoplazmatyczny + błonowy / IgG)
bezpośredni (antygen cytoplazmatyczny + błonowy / IgM i IgA)
Schemat reakcji PCR
(amplifikacja DNA genu bag1)
ekson 1
ekson 2
PCR 1
ex1
279 pz
ex2
ekson 4
ekson 3
PCR 2
PCR 3
ex4
ex3
232 pz
243 pz
PCR 4
ex3
ex4
437 pz
Konstrukcja plazmidu rekombinantowego
Produkt PCR 1
pUC19
EcoRI
2686 pz
MCS
EcoRI
ex1
ex2
SmaI
EcoRI / SmaI
Produkt PCR 4
HindIII
pUC19/ex1ex2
2943 pz
ex3
SmaI / HindIII
SmaI
CCT CCC CCC GGG
Pro
Pro
Pro
pUC19/BAG1
3080 pz
ex4
Konstrukcja plazmidu rekombinantowego
pUC19/BAG1
3080 pz
BglII / HindIII
pUET1
2856 pz
bag1 (694 pz)
MCS
BglII
HindIII
Klonowanie do wektora
ekspresyjnego pUET1 w miejsca
restrykcyjne BglII i HindIII DNA
produktu PCR
pUET1/BAG1
3465 pz
Immunoidentyfikacja i oczyszczanie
rekombinantowego białka BAG1
M 1 2 3 4
(A)
(B)
M 1
2
3 4
(C)
M 1 2
3 4 5 6
Rys. (A) Wynik testu Western blotting z użyciem przeciwciał skierowanych na domenę S-Tag;
(B) Rozdział elektroforetyczny białek zawartych we frakcjach zebranych lizatów w 12% żelu
poliakrylamidowym (SDS-PAGE); (C) Rozdział elektroforetyczny białek zawartych we frakcjach
uzyskanych podczas oczyszczania białka rekombinantowego BAG1 w 12% żelu
poliakrylamidowym (SDS-PAGE).
RODZINA
NAZWA
FORMA
ROZMOJOWA
MARKER
TEST
Antygeny
powierzchniowe
(SAG)
SAG1-His
SAG2-His
SAG4-His
P35-His
BSR4-His
tachyzoit
tachyzoit / bradyzoit
bradyzoit
tachyzoit
bradyzoit
w/p
późna
wczesna
późna
ELISA / Western b.
ELISA / Western b.
ELISA / Western b.
ELISA / Western b.
Western blotting
Antygeny granul
o dużej gęstości
(GRA)
GRA1-His
GRA2-His / GRA2ex2
GRA4-His
GRA5-His / GRA5-Trx
GRA6-His
GRA7-His
GRA9-His
tachyzoit / bradyzoit
tachyzoit / bradyzoit
tachyzoit / bradyzoit
tachyzoit / bradyzoit
tachyzoit / bradyzoit
tachyzoit / bradyzoit
tachyzoit / bradyzoit
Antygeny roptrii
(ROP)
ROP1-His
ROP9-His
tachyzoit / bradyzoit
tachyzoit
wczesna
nie badano
ELISA / Western b.
-
Antygeny
mikronem
(MIC)
MIC1-His
MIC3-His / MIC3-Trx
tachyzoit / bradyzoit
tachyzoit / bradyzoit
wczesna
wczesna
Western blotting
Western blotting
Enzymy
LDH1-His
LDH2-His
tachyzoit
bradyzoit
nie badano
nie badano
-
Inne
MAG1-His
BAG1-His
bradyzoit
bradyzoit
wczesna
późna
ELISA / Western b.
Western blotting
wczesna
ELISA / Western b.
ELISA / Western b.
Western blotting
Western blotting
ELISA / Western b.
ELISA / Western b.
Western blotting
Test Western blotting
M 1 2 3 4
M 1 2 3 4
M – marker wielkości
1 – MAG1
2 – GRA5
3 – GRA9
4 – GRA4
IgM +, IgG + / niska awidność
Toksoplazmoza wczesna (ostra)
M 1 2 3 4
IgM -, IgG >300 IU/ml / wysoka awidność
Toksoplazmoza przewlekła
IgM +, IgG + / wysoka awidność
Toksoplazmoza przewlekła ???
M 1 2 3 4
IgM -, IgG 48 IU/ml / wysoka awidność
Toksoplazmoza przewlekła
Test Western blotting
M 1 2
3
4
M – marker wielkości
M 1 2 3 4
1 – MIC3
2 – MAG1
3 – SAG4
4 – p35
IgM +, IgG + /
niska awidność
Toksoplazmoza wczesna (ostra)
IgM -, IgG >300 IU/ml /
wysoka awidność
Toksoplazmoza przewlekła
Test Western blotting
1 PACJENT
M 1 2
M 1 2 3
3
M – marker wielkości
1 – MAG1
2 – p35
3 – SAG4
1 pobranie surowicy
TOKSOPLAZMOZA WCZESNA
2 pobranie surowicy
(8 miesięcy później)
TOKSOPLAZMOZA PÓŹNA
DNA szczepionki
DNA szczepionki
Charakterystyka szczepionek
Ściśle zdefiniowany skład
• zawierają geny kodujące określone antygeny białkowe lub sekwencje
kodujące determinanty antygenowe
Bezpieczeństwo
• brak możliwości wywołania infekcji
• możliwość wywołania choroby autoimmunologicznej
indukcja produkcji autoprzeciwciał skierowanych przeciwko komórkom, w
których następuje biosynteza obcego antygenu
• możliwość integracji DNA plazmidowego z ludzkim DNA
Skuteczność
• wywołują zarówno humoralną jak i komórkową odpowiedź
immunologiczną
• uzależniona od rodzaju kodowanego antygenu
• uzależniona od poziomu ekspresji antygenu w komórce docelowej
DNA szczepionki
Charakterystyka szczepionek
• możliwość stosowania tylko jednej dawki szczepionki
długotrwała prezentacja antygenu systemowi immunologicznemu – biosynteza
antygenu in vivo
• możliwość stosowania jako szczepionki profilaktyczne lub terapeutyczne
ochrona przed infekcją
stymulacja układu immunologicznego u osobników zainfekowanych
• możliwość stosowania u niemowląt posiadających jeszcze matczyne
przeciwciała
DNA plazmidowe nie jest rozpoznawane i neutralizowane przez przeciwciała
długotrwała prezentacja antygenu umożliwia rozwój prawidłowej odpowiedzi
immunologicznej
• możliwość wywoływania odporności na różne szczepy tego samego
wirusa lub bakterii
możliwość wywołania odpowiedzi immunologicznej przeciwko
zakonserwowanym antygenom niż antygeny powierzchniowe
bardziej
DNA szczepionki
Charakterystyka szczepionek
Możliwość stosowania jako szczepionki skojarzone lub poliwalentne
•
•
kilka plazmidów kodujących różne antygeny
jeden plazmid kodujący kilka antygenów
Możliwość dostarczania DNA do organizmu różnymi sposobami
Łatwa formulacja szczepionek
•
brak konieczności stosowania adiuwantów, substancji konserwujących i
stabilizujących
Stabilność termiczna
•
liofilizowane lub sprecypitowane DNA można bardzo długo przechowywać
w temperaturze pokojowej
Niski koszt produkcji
•
•
możliwość produkcji dużych ilości plazmidowego DNA w komórkach
bakteryjnych
łatwa i tania procedura oczyszczania plazmidowego DNA - liza alkaliczna
DNA szczepionki
Sposoby formulacji i podawania szczepionek DNA
domięśniowo (iniekcja; DNA w roztworze soli fizjologicznej)
•
wydajność dostarczania
regenerujących się mięśni
DNA
wzrasta,
jeżeli
jest
podawane
do
śródskórnie (iniekcja; DNA w roztworze soli fizjologicznej)
dożylnie (iniekcja, DNA wewnątrz liposomów)
przez błony śluzowe
•
•
donosowo; DNA w roztworze soli fizjologicznej, w postaci aerozolu
doustnie; mikrokapsułki z biodegradowalnych polimerów zawierające DNA
przez skórę
•
•
skaryfikacja; DNA w roztworze soli fizjologicznej
dostarczanie za pomocą pistoletu genowego, cząsteczki złota opłaszczone
DNA (DNA dostarczane bezpośrednio do wnętrza komórek)
DNA szczepionki
Wektory plazmidowe stosowane do produkcji DNA
szczepionek (wektory wahadłowe)
bakteryjne ori replikacji
•
zapewnia dużą liczbę kopii plazmidowego
DNA w komórce bakteryjnej
prokariotyczny marker selekcyjny
•
umożliwia łatwą identyfikację komórek
bakteryjnych transformowanych DNA
plazmidowym
miejsce
(MCS)
•
wielokrotnego
klonowania
umożliwia łatwe wprowadzenie
kodującego antygen w obręb
plazmidowego
genu
DNA
DNA szczepionki
Wektory plazmidowe stosowane do produkcji DNA
szczepionek (wektory wahadłowe)
elementy regulujące transkrypcję
organizmach eukariotycznych
•
•
w
silne promotory pochodzenia eukariotycznego
lub wirusowego
promotor CMV ludzkiego wirusa cytomegalii
promotor SV40 małpiego wirusa S40
promotor CKM mięśniowo-specyficznej kinazy
kreatyny
eukariotyczne
lub
wirusowe terminatory
transkrypcji
zapewniające
poliadenylację
mRNA
sygnał poliadenylacji wirusa S40
sygnał poliadenylacji bydlęcego hormonu
wzrostu (BGH)
Odpowiedź immunologiczna indukowana przez
szczepionki DNA
DNA szczepionki
Możliwości zwiększenia skuteczności szczepionek DNA
zwiększenie poziomu ekspresji genu kodującego antygen
• nowe silne promotory
• promotory tkankowo-specyficzne
• sekwencja Kozaka inicjacji translacji w komórkach eukariotycznych
(-6GCCAGCCAUGGG+4)
• codon usage – stosowanie kodonów preferowanych przez komórki ssacze
• multimeryzacja genu kodującego antygen
zwiększenie immunogenności DNA szczepionki
• wprowadzenie do wektora plazmidowego immunogennych sekwencji CpG
• koekspresja antygenu z cząsteczkami immunostymulującymi
Szczepionki DNA poddawane próbom klinicznym
Kodowane
białko
Odpowiedź
humoralna
Odpowiedź
komórkowa
HIV
gp160, białka
regulatorowe,
białka rdzenia,
enzymy
nie badano
+
HBV
HBsAg
+
+
HSV
glikoproteina
HSV
w ocenie
w ocenie
hemaglutynina
w ocenie
w ocenie
nie badano
+
Patogen
wirus grypy
Plasmodium sp. CSP, Spf66
Metody genotypowe najczęściej stosowane w
badaniach epidemiologicznych
• Makrorestrykcyjna analiza genomowego DNA połączona z
elektroforezą pulsacyjną (REA/PFGE)
• Multilocus Sequence Typing (MLST)
• Mikromacierze DNA
• Analiza polimorfizmu długości fragmentów restrykcyjnych
amplifikowanego DNA (PCR/RFLP)
• Repetytywny ekstrageniczny palindromowy PCR (Rep-PCR)
• Przypadkowe amplifikowanie polimorficznego DNA (RAPD)
• Rybotypowanie
• Metody oparte o ligację adaptorów
Z wymienionych największą moc dyskryminacyjną mają techniki
polegające na analizie całego materiału genetycznego komórki
„Złoty standard”
w typowaniu mikroorganizmów”
Analiza restrykcyjna chromosomalnego DNA
połączona z elektroforezą pulsową (RFLP-PFGE)
Technika ta polega na poddaniu nienaruszonego DNA
genomowego trawieniu enzymatycznemu z zastosowaniem
restryktaz.
Rozdziału otrzymanych fragmentów restrykcyjnych dokonuje się
w agarozowej elektroforezie pulsowej (ang. PFGE – Pulsed Field
Gel Elektrophresis).
Analiza polimorfizmu długości fragmentów restrykcyjnych
możliwa dzięki elektroforezie pulsowej jest obecnie metodą
standardową w epidemiologii szpitalnej.
Krawczyk, B., Lewandowski, K., Bronk, M.,
Samet, A., Myjak, P., Kur, J.: Evaluation of a
novel method based on amplification of DNA
fragments surrounding rare restriction sites
(ADSRRS fingerprinting) for typing strains of
vancomycin-resistant Enterococcus faecium. J.
Microbiol. Meth. 52 (2003) 341-351.
Trudna w wykonaniu,
kosztowna, wymaga
skomplikowanej aparatury
zachowania stałych
warunków analizy.
Cechy efektywnej metody typowania szczepów:
• Wysoki stopień dyskryminacji
• Powtarzalność
• Zdolność do typowania wszystkich szczepów
• Prosta w użyciu
• Szybkość
• Mało kosztowna
• Wystandaryzowana
Potrzeba poszukiwania nowych metod typowania genetycznego, które powinny
charakteryzować się prostotą wykonania, porównywalną siłą dyskryminacji do
RFLP/PFGE, niskimi kosztami oraz łatwością adaptacji do badań rutynowych
METODY FINGERPRINTING
Wiele obecnie stosowanych technik typowania molekularnego
opiera się na analizie całego genomowego DNA
Metody te wykorzystują:
analizę restrykcyjną
amplifikację fragmentów DNA in vitro, przy zastosowaniu reakcji PCR
Rozdział powstałych fragmentów DNA o różnej długości uzyskuje się przy
pomocy technik elektroforetycznych.
Otrzymany w ten sposób profil prążków jest charakterystyczny zarówno
dla wybranej metody jak i dla badanej próby.
Różnice we wzorach świadczą o odmienności genetycznej badanych
organizmów.
wzór elektroforetyczny
=
„odcisk palca”
Metody typowania molekularnego oparte na
technice ang. Ligation Mediated PCR
AFLP- Amplified Fragment Length Polymorphism,
IRS- PCR – Infrequent Restriction Site PCR,
ADSRRS- fingerprinting – Amplification of DNA Fragments
Surrounding Rare Restriction Sites
PCR- MP – PCR Melting Profiles.
LM PCR (ang. Ligation – Mediated
Polimerase Chain Reaction)
Technika ta umożliwia analizę całego genomowego DNA
zarówno prokariotycznego jak i eukariotycznego, bez
potrzeby znajomości jego sekwencji
Etapy:
trawienie restrykcyjne
ligacja adaptorów
reakcja pre –PCR
reakcja PCR
elektroforeza produktów amplifikacji
Trawienie restrykcyjne
5’ - P
G GATC C
C CTAG G
5’ -
P GATCC
5’ - G
CCTAG GP - 5’ CCTAG GP - 5’
P
G GATCC
G GATCC
CCTAG G P - 5’
Ligacja adaptorów oligonukleotydowych
5’ - CTAG
CTAG - 5’
5’ - P GATC
CTAG
P
- 5’
Ligacja adaptorów oligonukleotydowych
5’ - P GATC
CTAG - 5’
5’ - CTAG
CTAG
P
- 5’
Ligacja adaptorów oligonukleotydowych
P
GATC
CTAG
CTAG
CTAG
P
Ligacja adaptorów oligonukleotydowych
GATC
CTAG
CTAG
CTAG
Reakcja pre - PCR
GATC
CTAG
CTAG
CTAG
Reakcja PCR
GATC
CTAG
Elektroforeza w żelu poliakrylamidowym
1
2
3
Td [ºC]
100
75
Td [ºC]
100
75
PCR MP – PCR Melting Profile
Ograniczoną reprezentację genomu można
otrzymać stosując LM PCR z niższą niż
zwykle temperaturą denaturacji w trakcie
reakcji PCR
PCR MP- cechy
Zalety:
Metoda nie wymaga znajomości analizowanej sekwencji;
Wady:
Prosta analiza wyników rozdziału w żelu
Termocykler
gradientowy;
poliakryloamidowym
wybarwionych
bromkiem etydyny;
Kalibracja
Ten sam adaptor
i enzymtermocyklera;
restrykcyjny może być użyty do
analizy DNA różnych gatunków organizmów;
Wysoki potencjał różnicujący;
Metoda szybka i stosunkowo tania ;
Bardzo dobra powtarzalność uzyskiwanych wyników;
PCR MP - PCR Melting Profile
Etap wstępny
Gradient temperatury denaturacji
Wybór odpowiedniej temperatury
denaturacji
87,7oC
PCR MP w wykrywaniu zakażeń szpitalnych na
poszczególnych oddziałach szpitala
Krawczyk, B., Samet, A., Leibner, J.,
Śledzińska, A., Kur, J.: Evaluation of a
PCR melting profile (PCR MP)
technique for bacterial strain
differentiation. J. Clin. Microbiol. 44
(2006) 2327-2332.
H2
PCR MP w badaniu indywidualnego pacjenta
Sytuacja epidemiologiczna indywidualnego pacjenta
Nr
pacjenta
Data
Materiał
Genotyp
6
23.09. 04
krew
H1
6
05.02. 05
krew
H11
6
05.02. 05
krew
H12
6
05.02. 05
krew
H13
6
07.02. 05
kał
H13
6
07.02. 05
kał
H12
6
10.05. 05
krew
H19
6
10.05. 05
kał
H19
6
19.05.05
krew
H19
6
19.05.05
krew
H19
6
22.05.05
krew
H24
6
22.05.05
krew
H24
6
25.05.05
krew
H24
6
25.05.05
krew
H24
6
27.05.05
krew
H24
6
08.07.05
krew
H24
PCR MP w badaniu indywidualnego pacjenta
Badanie transmisji (translokacji) bakterii
Co to jest translokacja bakterii?
migracja bakterii pochodzących z prawidłowej flory bakteryjnej i
ich toksyn przez ścianę przewodu pokarmowego i układu
moczowego, co w konsekwencji może wywoływać bakteriemię,
zakażenia narządowe, a nawet posocznicę oraz zapalenie
otrzewnej u chorych z marskością wątroby.
PCR MP w badaniu indywidualnego pacjenta
Badanie transmisji (translokacji) bakterii
Nr translokacji Nr izolatu
1
2
3
4
5
6
7
8
9
10
11
Data
Źródło
Nr pacjenta
izolacji
izolacji
12
07.02.05
13
07.02.05
69
10.05.05
70
10.05.05
39
15.02.05
40
18.02.05
1
12.11.04
2
12.11.04
35
12.02.05
36
13.02.05
37
15.02.05
23
14.12.04
24
15.12.04
45
22.02.05
46
25.02.05
28
01.01.05
29
02.01.05
52
03.03.05
53
04.03.05
33
12.02.05
34
13.02.05
67
03.05.05
68
03.05.05
6
6
24
1
23
15
26
17
31
22
40
Genotyp
krew
H13
kał
H13
krew
H20
kał
H20
krew
H15
kał
H15
krew
T1
mocz
T1
krew
T2
mocz
T2
kał
T2
krew
P1
kał
P1
krew
H17
kał
H17
krew
H9
mocz
H9
krew
T4
mocz
T4
krew
IM1
kał
IM1
krew
UG1
mocz
UG1
PCR MP w badaniu indywidualnego pacjenta
Badanie transmisji (translokacji) bakterii
Gradient temperatury
denaturacji
Potwierdzenie pokrewieństwa
genetycznego
Optymalizacja procedury PCR
MP dla Enterococcus faecium
Optymalizacja - cykl badań, który pozwoli określić
parametry krytyczne poszczególnych etapów metody
oraz wyznaczyć ich wartości optymalne
Cel optymalizacji:
skrócenie czasu przeprowadzania badania
zmniejszenie stopnia skomplikowania przeprowadzania badania
obniżenie kosztów analizy
z zachowaniem jakości i powtarzalności otrzymywanych wyników
Podczas optymalizacji PCR MP badano wpływ zmiany:
ilości, rodzaju i stężenia stosowanych odczynników
czasu trwania poszczególnych etapów
temperatury poszczególnych etapów
na jakość otrzymywanych wyników
Izolacja genomowego DNA
Enterococcus faecium
Trawienie restrykcyjne
genomowego DNA
Reakcja ligacji
W wyniku optymalizacji PCR MP określono:
parametry krytyczne metody
wartości optymalne badanych parametrów
Inaktywacja termiczna
Reakcja PCR
Elektroforeza
poliakrylamidowa
Wizualizacja
Wyznaczenie parametrów krytycznych
Wyznaczenie ilości mieszaniny ligacyjnej w
reakcji PCR
M
0.2
0.4
0.6 0.8
1
1.5
2
3
4
5
6
10
Wyznaczenie aktywności enzymu
restrykcyjnego
M
0.1
0.5
1
2
3
4
5
10
K-
Rys.1. Obraz elektroforetyczny produktów PCR, przy zastosowaniu różnej ilości
enzymu restrykcyjnego w reakcji trawienia; M – marker wielkości DNA (100 –
1000), K- kontrola ujemna, 0.1 – 10 –ilości enzymu w kolejnych próbach
Rys.2. Obraz elektroforetyczny produktów PCR, przy zastosowaniu
różnej ilości mieszaniny ligacyjnej; M – marker wielkości DNA (100 –
1000), K- kontrola ujemna, 0.2 – 10 – objętość mieszaniny ligacyjnej
w kolejnych próbach [ul]
Procedura skrócona
(wg Krawczyk B., Samet A., Leibner.
J, Śledzińska A. Kur J. (2006);
1h; 37°C
500 ng DNA
5U HindIII
Izolacja genomowego DNA
Enterococcus faecium
Trawienie restrykcyjne
genomowego DNA
1h, 16°C
adaptor: 20pmol każdego
oligonukleotydu
Vc= 25 μl
mieszanina ligacyjna 1μl
starter 25 pmol
Polimeraza PwoHis 0.6 U
Cykle: 22
20 min; 37°C
600 - 1000 ng DNA
3U Hind III
20min; 16°C
Reakcja ligacji
1U ligazaT4
10 min; 70°C
Procedura zoptymalizowana
adaptor: 10pmol każdego
oligonukleotydu
0,6U ligazaT4
Inaktywacja termiczna
Reakcja
10 min; PCR
70°C
Elektroforeza
poliakrylamidowa
Wizualizacja
Vc= 25 μl
mieszanina ligacyjna 1μl
starter 25 pmol
Polimeraza PwoHis 0.6 U
Cykle: 22
Weryfikacja zoptymalizowanej procedury PCR MP
Procedura
standardowa
M1 1
2
3
4
Procedura
zoptymalizowana
5
M2
1
2
3 4
5
Analiza porównawcza pięciu różnych szczepów E. faecium
Parametr
Skrócona
procedura
PCR MP
Zoptymalizowan
a procedura PCR
MP
czas badania
8h
6-7h
koszt badania
?
Zredukowany ze
względu na
zmniejszenie ilości
stosowanych
odczynników
jakość
otrzymywanyc
h wyników
bardzo dobra
bardzo dobra
interpretacja
wyników
łatwa
łatwa
Konstrukcja zestawu diagnostycznego
PCR MP unique
Zestaw diagnostyczny PCR MP unique wykorzystuje
zoptymalizowaną technikę PCR MP
Służy do różnicowania i genotypowania drobnoustrojów
w badaniach epidemiologicznych
Zestaw pozwala na przeprowadzenie 100 pełnych analiz,
przy zastosowaniu gotowych do użycia odczynników o
stężeniach i aktywnościach odpowiadającym optymalnym
Walidacja zestawu diagnostycznego
PCR MP unique
Walidacja dowolnej metody badawczej jest to działanie mające na celu
potwierdzenie w sposób udokumentowany i zgodny z założeniami, że
dana metoda badawcza jest odpowiednia dla zamierzonego celu i
pozwala uzyskać zaplanowane (prawidłowe) wyniki z odpowiednią
precyzją i dokładnością.
Walidacja przeprowadzona zgodnie z wytycznymi ICH (The
International Conferencer on Harmonisation of Technical
Requirements for Registration of Pharmaceuticals for Human Use)
oraz monografii EP (European Pharmacopeia) dla metod
analitycznych, wykorzystujących techniki amplifikacji kwasów
nukleinowych (NAT – Nucleic Acid Amplification Techniques)
Cechy walidowane:
specyficzność i selektywność
granica wykrywalności
elastyczność
powtarzalność
precyzja otrzymywanych wyników
Precyzja
Stopień zgodności otrzymanych wyników, gdy dana procedura jest stosowana dla wielokrotnie
powtórzonych, niezależnych oznaczeń.
Miarą precyzji jest stopień zgodności profili elektroforetycznych produktów reakcji PCR
otrzymanych na drodze niezależnych badań tego samego szczepu.
powtarzalność
M
Precyzja oznaczeń wykonanych przez
tą samą osobę
w tym samym laboratorium
na tej samej aparaturze oraz z
wykorzystaniem tych samych odczynników
w krótkim odstępie czasu
odtwarzalność
Precyzja oznaczeń wykonywanych przez:
inną osobę
w tym samym laboratorium
stosując te same odczynniki i aparaturę
3
23.01.2007
1 2
4
K-
M
1
2
3
4
6. 02.2007
K-
M 1
2
3
4
20. 02. 2007
K-
Walidacja zestawu diagnostycznego PCR MP unique podsumowanie
Zoptymalizowana procedura PCR MP jest:
specyficzna
selektywna
charakteryzuje się granicą oznaczalności DNA w ilości 250 ng
elastyczna
uniwersalna
ADSRRS fingerprinting (ang. Amplification of
DNA Surrounding Rare Restriction Sites)
Zjawisko supresji
Badania relacji epidemiologicznych pomiędzy nosicielstwem
Staphylococcus aureus a czyracznością (metoda ADSRRS)
Źródło
izolacji
Numer
izolatu
Oznaczenie
rodziny/pacjenta
PCR MP
genotyp
1
I/A
nos
A’
2
I/B
nos
A’
3
I/B
czyrak
A’
4
II/A
nos
B’
5
II/A
czyrak
B’
6
II/B
nos
C’
7
III/A
nos
D’
8
III/B
czyrak
B’
9
III/B
nos
D’
10
III/B
czyrak
(pacha)
B’
11
IV/A
nos
E’
12
IV/B
nos
F’
13
IV/B
czyrak
F’
Nowa klasa homodimerycznych białek SSB z rodziny
Deinococcaceae /Thermaceae
– charakterystyka molekularna, zastosowanie
SSB
(ang. single stranded DNA - binding protein)
białka wiążące się z jednoniciowym DNA
Funkcje białek SSB
są niezbędne w procesie replikacji DNA
stymulują polimerazy DNA
oddziaływują z innymi białkami replikacyjnymi (np. helikazą)
biorą udział w rekombinacji i naprawie DNA
Występowanie białek SSB
wszystkie żywe organizmy
wirusy
mitochondria
Klasyfikacja białek SSB
ze względu na budowę
► homotetramer:
- większość bakterii
- mitochondria
► heterotrimer:
- archaea
- eukaryota
► dimer:
- Thermus thermophilus (263 aa)
- Thermus aquaticus (264 aa)
- Deinococcus radiodurans (301 aa)
Termostabilność TaqSSB i TthSSB
T.aquaticus i T. thermophilus
rosną w optymalnej temp.
70ºC
Termostabilność białek TaqSSB i TthSSB
w T = 85ºC okres półtrwania wynosi 10 min
Termostabilność DgeoSSB i DmurSSB
95
85
okres półtrwania [min]
okres półtrwania [min]
50
40
30
20
10
0
75
65
55
45
35
25
65
70
75
80
85
Temperatura [°C]
DgeoSSB 61,93 kDa
monomer: 300 aa
90
95
80
85
90
95
100
Temperatura [°C]
DmurSSB 49,51kDa
monomer: 276 aa
105
110
Zastosowanie białek TaqSSB i TthSSB
w reakcji PCR…i nie tylko…
Aplikacje białek SSB
Zastosowanie białek
TaqSSB i TthSSB
Inne techniki
wykorzystujące
ssDNA i RNA
Reakcje PCR
Reakcja
multipleks PCR
Odwrotna
transkrypcja (RT)
Legenda:
Zastosowanie dowiedzione
Zastosowanie potencjalne
Reakcje
sekwencjonowania
trudnych matryc
Reakcja PCR – jedną z najbardziej
popularnych aplikacji termostabilnych białek
SSB
Wykorzystaliśmy białko TthSSB i TaqSSB do amplifikacji fragmentu
DNA wirusa CMV.
Rozdział elektroforetyczny w 2% żelu
agarozowym produktów reakcji PCR
przeprowadzanej w nieobecności TthSSB
(góra) lub z białkiem TthSSB (dół).
Powyżej
ścieżek
wskazano
liczbę
cząsteczek DNA (pCDNA21/CMV) w
każdej amplifikowanej próbce.
Rezultat:
TthSSB podnosi wydajność reakcji
PCR o 4 rzędy wielkości
Wykorzystanie białka TaqSSB w reakcji
multipleks PCR
► Krótkie tandemowe sekwencje repetytywne występujące w ludzkim chromosomie Y (YSTR) wykazują wysoki stopień polimorfizmu i są znacznikiem różnicującym osobniki.
Analiza polimorfizmu ludzkiego Y-STR umożliwia ustalenie ojcostwa i jest szeroko
stosowana w laboratoriach medycyny sądowej. Loci DYS390, DYS392 i DYS393 występujące
w Y-STR są dobrym markerem różnicującym
► W Zakładzie Medycyny Sądowej AM Gdańsk badano wpływ białka TaqSSB na amplifikację
multiplex PCR z dimeryzacją starterów. Do badania wpływu białka TaqSSB na efektywność
amplifikacji układu wybrano amplifikację dupleksu DYS390-DYS392, w której dimeryzacja
starterów zachodzi w trakcie procesu i powstaje nieprawidłowy produkt PCR długości 36 pz,
uniemożliwiający syntezę właściwych produktów PCR w standardowych warunkach
Hybryd utworzony przez startery DYS390 i DYS392
starter DYS390
5’
3’
3’
5’
starter DYS392
Wynik reakcji multiplex PCR
Startery DYS390 = A
Startery DYS392 = B
Startery DYS393 = C
M – wzorzec wielkości DNA (pGEM, Promega)
1 - produkty amplifikacji ze starterami C-A+TaqSSB
2 - produkty amplifikacji ze starterami C-A
3 - produkty amplifikacji ze starterami C-B+TaqSSB
4 - produkty amplifikacji ze starterami C-B
5 - produkty amplifikacji ze starterami A-B+TaqSSB
6 - produkty amplifikacji ze starterami A-B
7 - produkty amplifikacji ze starterami A-B-C+TaqSSB
8 - produkty amplifikacji ze starterami A-B-C
Wnioski dotyczące reakcji PCR
DYS390, DYS392 i DYS393
► Startery DYS390 i DYS392 tworzą bardzo stabilną hybrydę, łącząc się ze sobą
na 3’ końcach, do których polimeraza dosyntetyzowuje nukleotydy i tworzy się
dodatkowy produkt o wielkości 36 pz.
► Na podstawie przeprowadzonego doświadczenia można wnioskować, że
dodatek termostabilnego białka SSB uniemożliwia tworzenie się struktur
drugorzędowych,
zarówno
wewnątrzcząsteczowych,
jak
i
międzycząsteczkowych, dzięki czemu, uzyskuje się większą wydajność reakcji
PCR, a tym samym pozwala na uzyskanie oczekiwanego produktu reakcji PCR.
Wpływ białka TaqSSB na reakcję PCR
Wynik
reakcji PCR modelowego układu, w którym startery wiążą się ze
sobą na końcach 3’
5’ CGACGATTCTGAGTACTTGCA 3’
3’ AGACTCATGAACGTAGCAGCA 5’
M – marker wielkości M100-500
1 – reakcja bez białka TaqSSB
2 – reakcja z dodatkiem 6l białka TaqSSB
3 – reakcja z dodatkiem 8l białka TaqSSB
4 – reakcja z dodatkiem 9l białka TaqSSB
5 – reakcja z dodatkiem 10l białka TaqSSB
6 – reakcja z dodatkiem 11l białka TaqSSB
7 – reakcja z dodatkiem 12l białka TaqSSB
8 – reakcja z dodatkiem 14l białka TaqSSB
stężenie białka TaqSSB – 1.12 g/l
Dodatek innych znanych „wzmacniaczy” reakcji PCR jak np.:
EcoSSB/Formamid/TMAC/DMSO/TritonX-100
nie przyniósł satysfakcjonujących rezultatów !!!










