(pieczęć wydziału)
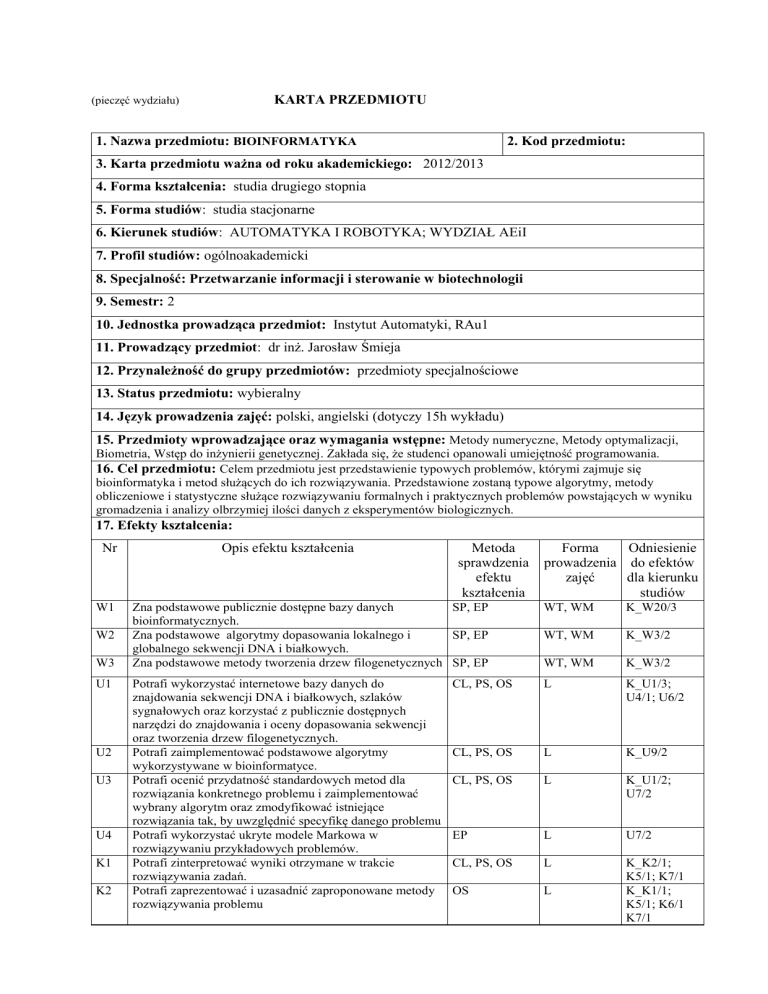
KARTA PRZEDMIOTU
1. Nazwa przedmiotu: BIOINFORMATYKA
2. Kod przedmiotu:
3. Karta przedmiotu ważna od roku akademickiego: 2012/2013
4. Forma kształcenia: studia drugiego stopnia
5. Forma studiów: studia stacjonarne
6. Kierunek studiów: AUTOMATYKA I ROBOTYKA; WYDZIAŁ AEiI
7. Profil studiów: ogólnoakademicki
8. Specjalność: Przetwarzanie informacji i sterowanie w biotechnologii
9. Semestr: 2
10. Jednostka prowadząca przedmiot: Instytut Automatyki, RAu1
11. Prowadzący przedmiot: dr inż. Jarosław Śmieja
12. Przynależność do grupy przedmiotów: przedmioty specjalnościowe
13. Status przedmiotu: wybieralny
14. Język prowadzenia zajęć: polski, angielski (dotyczy 15h wykładu)
15. Przedmioty wprowadzające oraz wymagania wstępne: Metody numeryczne, Metody optymalizacji,
Biometria, Wstęp do inżynierii genetycznej. Zakłada się, że studenci opanowali umiejętność programowania.
16. Cel przedmiotu: Celem przedmiotu jest przedstawienie typowych problemów, którymi zajmuje się
bioinformatyka i metod służących do ich rozwiązywania. Przedstawione zostaną typowe algorytmy, metody
obliczeniowe i statystyczne służące rozwiązywaniu formalnych i praktycznych problemów powstających w wyniku
gromadzenia i analizy olbrzymiej ilości danych z eksperymentów biologicznych.
17. Efekty kształcenia:
Nr
W1
W2
W3
U1
U2
U3
U4
K1
K2
Opis efektu kształcenia
Metoda
sprawdzenia
efektu
kształcenia
Forma
Odniesienie
prowadzenia do efektów
zajęć
dla kierunku
studiów
Zna podstawowe publicznie dostępne bazy danych
SP, EP
bioinformatycznych.
Zna podstawowe algorytmy dopasowania lokalnego i
SP, EP
globalnego sekwencji DNA i białkowych.
Zna podstawowe metody tworzenia drzew filogenetycznych SP, EP
WT, WM
K_W20/3
WT, WM
K_W3/2
WT, WM
K_W3/2
Potrafi wykorzystać internetowe bazy danych do
znajdowania sekwencji DNA i białkowych, szlaków
sygnałowych oraz korzystać z publicznie dostępnych
narzędzi do znajdowania i oceny dopasowania sekwencji
oraz tworzenia drzew filogenetycznych.
Potrafi zaimplementować podstawowe algorytmy
wykorzystywane w bioinformatyce.
Potrafi ocenić przydatność standardowych metod dla
rozwiązania konkretnego problemu i zaimplementować
wybrany algorytm oraz zmodyfikować istniejące
rozwiązania tak, by uwzględnić specyfikę danego problemu
Potrafi wykorzystać ukryte modele Markowa w
rozwiązywaniu przykładowych problemów.
Potrafi zinterpretować wyniki otrzymane w trakcie
rozwiązywania zadań.
Potrafi zaprezentować i uzasadnić zaproponowane metody
rozwiązywania problemu
CL, PS, OS
L
K_U1/3;
U4/1; U6/2
CL, PS, OS
L
K_U9/2
CL, PS, OS
L
K_U1/2;
U7/2
EP
L
U7/2
CL, PS, OS
L
OS
L
K_K2/1;
K5/1; K7/1
K_K1/1;
K5/1; K6/1
K7/1
18. Formy zajęć dydaktycznych i ich wymiar (liczba godzin)
W. : 30
L.: 30
19. Treści kształcenia:
Wykład
1. Zakres i zastosowania bioinformatyki. Przykładowe zagadnienia.
2. Metody badań eksperymentalnych, postać otrzymywanych danych, powtarzalność eksperymentów, źródła
błędów; zagadnienia statystyczne normalizacji i standaryzacji danych.
3. Łańcuchy Markowa, metody MCMC; algorytm Metropolisa-Hastingsa, symulowane wyżarzanie.
4. Ukryte łańcuchy Markowa; Algorytm Viterbiego, algorytm Bauma-Welcha.
5. Wyszukiwanie motywów w sekwencjach DNA. Baza danych PROSITE.
6. Porównywanie sekwencji nukleotydów w DNA oraz aminokwasów w białkach; macierze dopasowania;
algorytm Needlemana-Wunsha i Smitha-Watermana. Kojarzenie wielu sekwencji jednocześnie. Programy
BLAST, CLUSTALW i CLUSTALX.
7. Analiza sekwencji białek. Wyszukiwanie domen i motywów, przewidywanie własności fizyko-chemicznych,
drugo- i trzecio-rzędowej struktury białka; analiza danych z eksperymentow spektroskopowych.
8. Techniki redukcji wymiarowości w analizie danych mikromacierzowych; analiza składowych głównych
(Principal Component Analysis - PCA), rozkład wartości osobliwych (Singular value Decomposition - SVD),
Metoda Częściowych Najmniejszych Kwadratów (Partial-Least –Squares – PLS)
9. Klasyfikacja i klasteryzacja na podstawie pomiarów ekspresji genów; klasyfikatory liniowe, wykorzystanie sieci
neuronowych, maszyny wektorów wspierających (Support Vector Machines – SVM)
10. Metody odtwarzania drzew filogenetycznych; klasyczne metody oceny topologii i metryki drzew. Algorytm
Felsensteina - Churchila.
11. Kinetyka reakcji biochemicznych. Ograniczenia w modelowaniu, zakresy parametrów, podstawowe
nieliniowości; stany ustalone i quasi-ustalone.
12. Sieci genowe i ścieżki sygnałowe. Dodatnie i ujemne sprzężenia zwrotne regulujace dynamikę ścieżek
sygnałowych.
13. Deterministyczne modele ścieżek sygnałowych. Identyfikacja parametrów; ocena własności dynamicznych;
analiza bifurkacyjna.
14. Stochastyczne modelowanie ścieżek sygnałowych. Algorytm Gillespiego.
15. Wykorzystanie bioinformatyki i biologii obliczeniowej w biotechnologii, przemyśle farmaceutycznym i
medycynie.
Zajęcia laboratoryjne
1. · Znajdowanie dopasowania sekwencji DNA
2. · Znajdowanie dopasowania sekwencji białek
3. · Wykorzystanie algorytmu Viterbiego do analizy sekwencji DNA
4. · Wyszukiwanie wzorców w danych mikromacierzowych
5. ·Redukcję wymiarowości w analizie danych mikromacierzowych
6. · Estymację parametrów metodą największej wiarygodności
7. · Metodę symulowanego wyżarzania
8. · Konstruowanie i analizę drzew filogenetycznych
9. · Modelowanie prostych szlaków sygnałowych
20. Egzamin: nie
21. Literatura podstawowa:
1. A.D. Baxevanis, B.F.F. Ouellette (red.): Bioinformatyka. Podręcznik do analizy genów i białek. Wydawnictwo
Naukowe PWN, 2005.
2. J.D. Murray: Wprowadzenie do biomatematyki. Wydawnictwo Naukowe PWN, 2007.
3. Primrose S. B.: Zasady analizy genomu. Przewodnik do mapowania i sekwencjonowania DNA różnych
organizmów. WNT 1999.
4. Materiały pomocnicze do wykładów umieszczane na stronie
http://dydaktyka.zis.ia.polsl.gliwice.pl
5. A. Polański, M. Kimmel, Bioinformatics. Springer-Verlag Berlin Heidelberg, 2007.
22. Literatura uzupełniająca:
1. J. Kączkowski: Podstawy biochemii, WNT 2005.
2. R. Durbin, S.R. Eddy, A. Krogh, G. Mitchison: Biological Sequence Analysis: Probabilistic Models of Proteins
and Nucleic Acids. Cambridge University Press, 1999.
3. G.J. McLachlan, T. Krishnan: The EM algorithm and extensions. John Wiley & Sons, New York, 1997.
4. http://bioinformatics.org/
5. Materiały udostępnione przez Szwajcarski Instytut Bioinformatyki: http://www.isb-sib.ch/ oraz Europejski
Instytut Bioinformatyki: http://www.ebi.ac.uk/
23. Nakład pracy studenta potrzebny do osiągnięcia efektów kształcenia
Lp.
Forma zajęć
1
Wykład
2
Ćwiczenia
3
Laboratorium
4
Projekt
0/0
5
Seminarium
0/0
6
Inne
0/0
Suma godzin
Liczba godzin
kontaktowych / pracy studenta
30/0
0/0
30/30
60/30
24. Suma wszystkich godzin: 90
25. Liczba punktów ECTS: 3
26. Liczba punktów ECTS uzyskanych na zajęciach z bezpośrednim udziałem nauczyciela akademickiego: 2
27. Liczba punktów ECTS uzyskanych na zajęciach o charakterze praktycznym (laboratoria, projekty): 2
26. Uwagi:
Zatwierdzono:
…………………………….
…………………………………………………
(data i podpis prowadzącego)
(data i podpis dyrektora instytutu/kierownika katedry/
Dyrektora Kolegium Języków Obcych/kierownika lub
dyrektora jednostki międzywydziałowej)