Załącznik nr 2
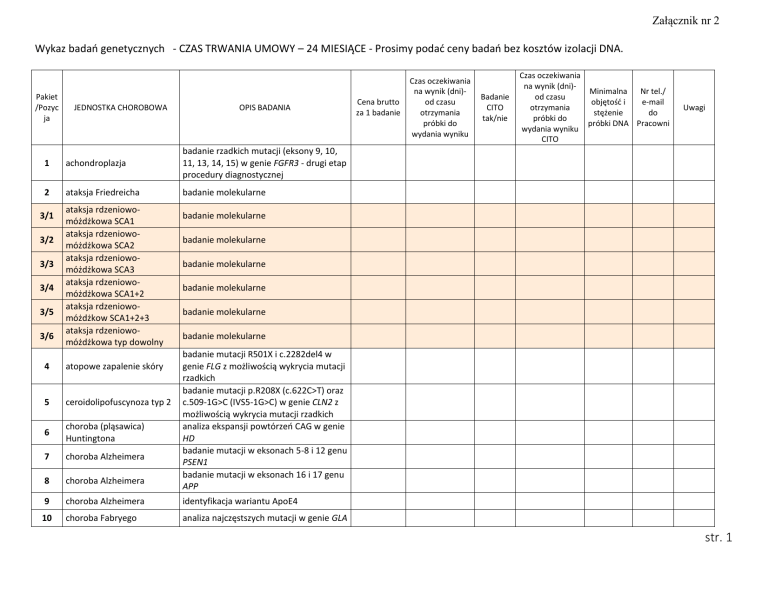
Wykaz badań genetycznych - CZAS TRWANIA UMOWY – 24 MIESIĄCE - Prosimy podać ceny badań bez kosztów izolacji DNA.
Pakiet
/Pozyc
ja
JEDNOSTKA CHOROBOWA
OPIS BADANIA
1
achondroplazja
badanie rzadkich mutacji (eksony 9, 10,
11, 13, 14, 15) w genie FGFR3 - drugi etap
procedury diagnostycznej
2
ataksja Friedreicha
badanie molekularne
3/1
3/2
3/3
3/4
3/5
3/6
ataksja rdzeniowomóżdżkowa SCA1
ataksja rdzeniowomóżdżkowa SCA2
ataksja rdzeniowomóżdżkowa SCA3
ataksja rdzeniowomóżdżkowa SCA1+2
ataksja rdzeniowomóżdżkow SCA1+2+3
ataksja rdzeniowomóżdżkowa typ dowolny
Cena brutto
za 1 badanie
Czas oczekiwania
na wynik (dni)od czasu
otrzymania
próbki do
wydania wyniku
Badanie
CITO
tak/nie
Czas oczekiwania
na wynik (dni)Minimalna
Nr tel./
od czasu
objętość i
e-mail
otrzymania
stężenie
do
próbki do
próbki DNA Pracowni
wydania wyniku
CITO
Uwagi
badanie molekularne
badanie molekularne
badanie molekularne
badanie molekularne
badanie molekularne
badanie molekularne
badanie mutacji R501X i c.2282del4 w
genie FLG z możliwością wykrycia mutacji
rzadkich
badanie mutacji p.R208X (c.622C>T) oraz
c.509-1G>C (IVS5-1G>C) w genie CLN2 z
możliwością wykrycia mutacji rzadkich
analiza ekspansji powtórzeń CAG w genie
HD
badanie mutacji w eksonach 5-8 i 12 genu
PSEN1
badanie mutacji w eksonach 16 i 17 genu
APP
4
atopowe zapalenie skóry
5
ceroidolipofuscynoza typ 2
6
choroba (pląsawica)
Huntingtona
7
choroba Alzheimera
8
choroba Alzheimera
9
choroba Alzheimera
identyfikacja wariantu ApoE4
10
choroba Fabryego
analiza najczęstszych mutacji w genie GLA
str. 1
Załącznik nr 2
11
choroba Gauchera
badanie najczęstszych mutacji (N370S i
L444P) w genie GBA z możliwością
wykrycia mutacji rzadkich
12
choroba Kennedy'ego
(SBMA)
badanie molekularne
13
choroba Krabbego
badanie rozległej delecji IVS10del30kb pierwszy etap procedury diagnostycznej
14
choroba Lebera
badanie najczęstszych mutacji mtDNA
choroba Morquio B
(mukopolisacharydoza typu
IVB)
choroba Niemanna-Picka
typ A i B
badanie mutacji w genie GLB1 (eksony:
2,3,8,12,14,15) - pierwszy etap procedury
diagnostycznej
15
16
17
choroba Wilsona
18
choroba PelizaeusaMerzbachera (PLP)
19
20
choroba Parkinsona o
wczesnym początku
choroba Parkinsona o
wczesnym początku
badanie regionu kodującego genu SMPD1
badanie mutacji w eksonach 6, 7, 9, 10,
11 genu ATP7B
analiza sekwencji kodującej
test MLPA
metoda: MLPA
sekwencjonowanie genu PARK2
21
CMT1A
analiza genu PMP22 (sekwencjonowanie)
22
CMT2A typ aksonalny
analiza genu MFN2 (sekwencjonowanie)
23
deficyt alfa1-antytrypsyny
24
deficyt alfa1-antytrypsyny
25
deficyt alfa1-antytrypsyny
26
deficyt biotynidazy
27
drżenie samoistne
dysplazja ektodermalna
hypohydrotyczna,
sprzężona z X
28
badanie mutacji S i Z w genie AAT (inna
nazwa genu: PI, SERPINA1)
badanie mutacji w regionie kodującym
genu AAT (badanie eksonów 2, 3, 4 i 5)
badanie mutacji w eksonach 2 i 3 genu
AAT
badanie mutacji p.Cys33PhefsX36,
p.Arg538Cys, p.Gln456His, p.Asp444His
oraz innych rzadkich mutacji w eksonach
2 i 4 genu BTD
badanie mutacji S9G w genie DRD3
badanie mutacji najczęstszych w
eksonach 2, 4, 6 i 7 genu EDA
str. 2
Załącznik nr 2
29
30
31
dystonia typ 6 z dyskinezą
dystrofia kończynowoobręczowa typ 2A
(LGMD2A)
dystrofia mięśniowa
obręczowo-kończynowa
(LGMD) typ 1A
sekwencjonowanie genu THAP1
badanie mutacji c.550delA i R490Q w
genie CAPN3
badanie najczęstszych mutacji w genie
TTID
32/1 dystrofia miotoniczna typ 1 badanie molekularne
32/2 dystrofia miotoniczna typ 2 badanie molekularne
32/3
dystrofia miotoniczna typ
1+2
33
fenyloketonuria
34
fenyloketonuria
35
fibrodysplasia ossificans
progressiva
36
fruktozemia
37
galaktozemia
38
gangliozydoza
39/1 gen BRCA1
39/2 gen BRCA2
39/3 geny BRCA1 + BRCA2
badanie molekularne
badanie mutacji R408W, R158Q,
c.1315+1G>A, c.1066-11G>A oraz innych
rzadkich mutacji w eksonach 5 i 12 genu
PAH - pierwszy etap procedury
diagnostycznej
badanie mutacji w eksonach 2, 3, 6, 7, 11
genu PAH - drugi etap procedury
diagnostycznej
badanie najczęstszej mutacji R206H w
eksonie 7 genu ACVR1 [1] - pierwszy etap
diagnostyki
badanie mutacji A150P i A175D w genie
ALDOB z możliwością wykrycia mutacji
rzadkich
badanie eksonów 6, 7, 8 i 9 genu GALT
(badanie obejmuje dwie najczęstsze
mutacje: Q188R i K285N)
badanie mutacji w genie GLB1 (eksony 26, 8, 14 i 15) - pierwszy etap procedury
diagnostycznej
sekwencjonowanie całej sekwencji
kodującej genu
sekwencjonowanie całej sekwencji
kodującej genu
sekwencjonowanie całych sekwencji
kodujących genów
str. 3
Załącznik nr 2
40
gen CFTR
identyfikacja ponad 600 mutacji genu w
tym 16 najczęstszych w Polsce
41
głuchota mitochondrialna
analiza powszechnej mutacji m.1555A>G
w genie MTRNR1 (sekwencjonowanie)
42
hiperbilirubinemia - zespół
Crigler-Najjar, przejściowa
noworodkowa
hiperbilirubinemia,
diagnostyka uzupełniająca
w zespole Gilberta
badanie regionu kodującego genu
UGT1A1 - diagnostyka uzupełniająca
43
hypochondroplazja
44
hypochondroplazja
45
inne choroby " sieroce"
46
inne choroby " sieroce"
47
inne choroby " sieroce"
48
49
MCAD (deficyt
dehydrogenazy acylo-CoA
średnołańcuchowych
kwasów tłuszczowych)
mitochondrialne zespoły
delecyjne zespół Kearnsa i
Sayrea (KSS)
zespół Pearsona (PS)
zespół postępującej
badanie najczęstszych mutacji (c.C1620A i
c.C1620G)w genie FGFR3 - pierwszy etap
procedury diagnostycznej
badanie rzadkich mutacji w genie FGFR3
(fragment eksonu 8 oraz eksony 9, 13, 14,
15)- drugi etap procedury diagnostycznej
badanie genetyczne mające na celu
wykrycie mutacji odpowiedzialnej za
chorobę rzadką lub ultrarzadką – analiza
1 amplikonu metodą PCR i
sekwencjonowania
badanie genetyczne mające na celu
wykrycie mutacji odpowiedzialnej za
chorobę rzadką lub ultrarzadką – analiza
2 amplikonów metodą PCR i
sekwencjonowania
badanie genetyczne mające na celu
wykrycie mutacji odpowiedzialnej za
chorobę rzadką lub ultrarzadką – analiza
3-5 amplikonów metodą PCR i
sekwencjonowania
badanie najczęstszej mutacji Lys 304Glu
w genie ACAMD z możliwością wykrycia
mutacji rzadkich
analiza powszechnej delecji
m.8469_13447del i innych delecji mtDNA
[MLPA / long PCR]
str. 4
Załącznik nr 2
zewnętrznej oftalmoplegii
(PEO)i inne
50
51
52
53
mnoga gruczolakowatość
badanie mutacji w eksonach 10, 11,13,
wewnątrzwydzielnicza typu
14, 15 i 16 genu RET
2 (MEN2A i MEN2B)
mózgowa arteriopatia z
podkorowymi zawałami i
leukoencefalopatią CADASIL
mukopolisacharydoza typ
IIIA
mukopolisacharydoza typ
VI
54
neurofibromatoza typu 2
55
niedosłuch (DFNB1)
56
niedosłuch (DFNB1)
57
niepełnosprawność
intelektualna sprzężona z
chromosomem X (MRX)
badanie mutacji w eksonach 2, 3, 5, 6 i 11
genu NOTCH3 - drugi etap procedury
diagnostycznej
badanie mutacji (np. R74C, R245H,
S298P) w eksonach od 2 do 7 genu SGSH
badanie całego regionu kodującego genu
ARSB
badanie wybranych regionów genu NF2 I etap diagnostyki
badanie mutacji 35delG, 310del14,
IVS1+1G>A oraz mutacji rzadkich w części
kodującej genu GJB2
badanie najczęstszych mutacji genu GJB6
ARX
MRX
analiza wszystkich
eksonów
identyfikacja
najczęstszych mutacji w
eksonie 2
test MLPA (14 genów,
P106)
58
test z mitomycyną C
(w kierunku łamliwości
chromosomów)
59
trombocytopenia
(małopłytkowość)
badanie mutacji Glu167Asp w genie
MASTL oraz c.1-116C>T, c.-118C>T, c.1125T>G, c.1-127A>T, c.1-128G>A, c.1134G>A w genie ANKRD26
60
wielotorbielowatość nerek
- postać dziedziczna
autosomalna recesywna
(ARPKD)
badanie wybranych regionów genu
PKHD1 (eksony 32, 36, 59) - pierwsze
etap diagnostyki
str. 5
Załącznik nr 2
61
wrodzona biegunka
sodowa
wrodzona neuropatia
czuciowa i ruchowa
analiza wybranych regionów genu SPINT2
63/1
zapalenie trzustki (ostre i
przewlekłe)
63/2
zapalenie trzustki (ostre i
przewlekłe)
63/3
zapalenie trzustki (ostre i
przewlekłe)
badanie najczęstszych mutacji (K141N i
K141E) w genie HSPB8
badanie 12 najczęstszych mutacji (m. in.
R122H, R122C, A16V, N29I) w genie
PRSS1 z możliwością wykrycia mutacji
rzadkich
badanie najczęstszych mutacji w genach
PRSS1, SPINK1 i CFTR (Badanie 12
najczęstszych mutacji genu PRSS1,
badanie mutacji N34S w genie SPINK1
oraz badanie mutacji F508del,
dele2,3(21kb) IVS8-T+(TG) oraz mutacji w
eksonach 10 i 11 w genie CFTR
badanie mutacji w genie CTRC (np.
R254W, 738_761del24, W55X)
63/4
zapalenie trzustki (ostre i
przewlekłe)
badanie mutacji w całym regionie
kodującym genu SPINK1
zapalenie trzustki (ostre i
przewlekłe)
zespół Alporta - postać
sprzężona z chromosomem
X
badanie mutacji w eksonach 1, 2 i 4 genu
SPINK1 - diagnostyka uzupełniająca
badanie wybranych regionów genu
COL4A5 (eksony 49, 50, 51) - pierwszy
etap diagnostyki
65
zespół Andersen-Tawila
analiza sekwencji kodującej genu KCNJ2
66
zespół Aperta
67
zespół Beckwitha i
Wiedemanna
68
zespół Blooma
62
63/5
64
analiza sekwencji eksonu 7, w tym
identyfikacja mutacji p.Ser252Trp i
p.Pro253Arg – gen FGFR2
test metylacyjny regionu 11p15 na
obecność delecji, UPD11pat, defektów
piętnowania [MS-MLPA]
analiza UPD11pat [STR]; (3 osoby; 5
markerów)
analiza całego genu CDKN1C (ex. 1-2)
metoda: sekwencjonowanie
badanie 70 mutacji genu BLM najczęściej
występujących w rasie białej - pierwszy
etap procedury diagnostycznej
str. 6
Załącznik nr 2
69
zespół Dravet
metoda: sekwencjonowanie
70
zespół Dravet
metoda: MLPA
71
zespół Freemana-Sheldona
72
zespół Hioba (zespół hiperIgE)
73
zespół hipoplazji lewego
serca
badanie mutacji w genie GJA1
74
zespół Kabuki
analiza wybranych regionów genu MLL2
75
zespół Lowe, zespół ocznomózgowo-nerkowy
badanie mutacji w eksonie 15 genu OCRL
zespół łamliwego
76/1
chromosomu X
badanie najczęstszych mutacji (R672C i
R672H) w genie MYH3
badanie mutacji najczęściej
występujących w populacji polskiej w
genie STAT3
badanie przesiewowe w celu wykrycia
ekspansji powtórzeń (CGG)n w genie
FMR1
badanie premutacji i mutacji dynamicznej
z zastosowaniem hybrydyzacji
genomowej
badanie przesiewowe z określeniem
liczby powtórzeń (CGG) w genie FMR1
badanie rozszerzone zastępujące
hybrydyzację:
AmplideX FMR1 PCR lub FragileX PCR kit
badanie najczęstszej mutacji
c.3816+1G>A w genie COL11A1
badanie mutacji 565delCTGA i R210H
oraz innymch mutacji w eksonie 7 genu
GNAS
76/2
zespół łamliwego
chromosomu X
76/3
zespół łamliwego
chromosomu X
76/4
zespół łamliwego
chromosomu X
77
zespół Marshalla
78
zespół McCune-Albright
79
zespół Mohra-Tranebjærga
analiza regionu kodującego genu
TIMM8A
zespół Mowata-Wilsona
badanie genu ZEB2 (inne nazwy: ZFHX1B;
SIP-1): identyfikacja najczęstszej mutacji
p.Arg695X - pierwszy etap procedury
diagnostycznej
80
str. 7
Załącznik nr 2
81
zespół Mulibrey nanism
badanie najczęstszej mutacji c.493-2A>G
w genie TRIM37
82
zespół Nethertona
badanie wybranych eksonów genu
SPINK5
83
zespół Nijmegen
84/1 zespół Noonan
84/2 zespół Noonan
84/3 zespół Noonan
84/4 zespół Noonan
84/5 zespół Costello
badanie najczęstszej mutacji
c.657_661del5 (p.Lys219AsnfsX15) w
genie NBS1
badanie mutacji w eksonach 2, 3, 4, 7, 8,
9, 12 i 13 genu PTPN11 - pierwszy etap
procedury diagnostycznej
badanie mutacji w genie SOS1 (eksony 4,
5, 7, 8, 9, 11, 12, 13, 14, 15, 17) - drugi
etap procedury diagnostycznej
badanie mutacji w genie SOS1 (eksony 2,
3, 6, 10, 16, 18-24) - trzeci etap
procedury diagnostycznej
badanie mutacji w regionie kodującym
genu KRAS
sekwencjonowanie genu HRAS
analiza sekwencji eksonów 8 i 10
(identyfikacja najczęstszych mutacji) –
gen FGFR2
analiza sekwencji eksonu 7, w tym
identyfikacja mutacji p.Pro252Arg – gen
FGFR1
analiza wybranych regionów genu TCF4 - I
etap diagnostyki
85
zespół Pfeiffera/Crouzona
86
zespół Pfeiffera
87
zespół Pitt-Hopkins
88
zespół RubinsteinaTaybiego
analiza genu CREBBP (inna nazwa CBP)
89
zespół Saethre−Chotzen
analiza sekwencji kodującej genu TWIST1
90
zespół ShwachmanaDiamonda
91
zespół Silvera i Russella
badanie fragmentu regionu kodującego
genu SBDS (eksony 1-4)
test metylacyjny regionu 11p15 [MSMLPA]
analiza UPD7mat [STR] (3 osoby, 4
markery)
str. 8
Załącznik nr 2
92
93
94
95
zespół Simpsona, Golabiego
analiza sekwencji kodującej genu GPC3
i Behmela typu1
zespół Smith-Lemli-Opitz
zespół włosowo-nosowo
palcowy
7,8-dehydrocholesterol
(badanie biochemiczne)
badanie mutacji p.Trp151X,p.Val326Leu,
p.Leu157Pro, p.Arg353Trp, IVS8-1G>C,
p.Arg446Gln)w genie DHCR7 z
możliwością wykrycia mutacji rzadkich
badanie całego regionu kodującego genu
TRPS
str. 9










