Genom człowieka. Typy mutacji genomu i związane z tym
choroby genetyczne. III Rok WL1
dr Katarzyna Wicher
1. Budowa, funkcje i rodzaje DNA
-podstawowe funkcje kwasu deosyrybonukleinowego
-struktura prawoskrętnej helisy DNA-B, rodzaje wiązań stabilizujących strukturę helisy, pojęcie
komplementarności nici, zasada Chargaffa
-konformacje DNA: DNA-A, DNA-B, DNA-Z
-etapy kondensacji chromatyny (nukleosom, nukleofilamenty, solenoid, włókienka wyższego rzędudomeny pętlowe, chromosom metafazowy)
2. Genom-całkowity DNA zawarty w komórce danego organizmu.
Genom człowieka zawiera około 21 000 genów kodujących białka, a odcinki kodujące stanowią tylko
ok. 3% całego genomu.
Budowa fizyczna genomu człowieka
Genom jądrowy
zbudowany z około 3 Gb (3000 Mb), dsDNA
złożony z 24 rożnych liniowych cząsteczek – chromosomów
zakres wielkości cząsteczek (chromosomów) od 55 Mb do 250 Mb
wśród chromosomów wyróżniamy autosomy i heterosomy
Genom mitochondrialny
dziedziczony w linii matczynej
zbudowany z około 16 kb
kolista cząsteczka dsDNA
występuje w komórce w wielu kopiach
obecny we wszystkich mitochondriach (w wielu kopiach)
wykazuje wyższą częstość mutacji niż DNA jądrowy (wolne rodniki tlenowe, brak systemów
naprawczych, inna struktura mtDNA - brak histonów, brak intronów)
charakterystyczny wzór dziedziczenia (rodowód: chora matka ma wszystkie dzieci chore,
chory ojciec nie przekazuje choroby potomstwu)
3. Geny i sekwencje związane z genami (30%)
eksony
introny
pseudogeny
fragmenty genów
sekwencje regulatorowe (blisko i dalekozasięgowe)
4. Budowa genu- eksony, introny oraz sekwencje 5’ oraz 3’ flankujące
.
Dwa przykłady niezwykłej organizacji genów:
Geny nakładające się
W niektórych regionach chromosomów, w których zagęszczenie genów jest duże, a sekwencja jest
bogata w pary GC mamy do czynienia ze zjawiskiem nakładania się genów (ang. overlapping genes).
Zwykle sekwencje aminokwasowe białek kodowanych przez kilka genów zachodzących na siebie nie
są podobne. Przykładem mogą być geny kompleksu HLA w chromosomie pary 16 (lokalizacja
16p21.3).
Geny wewnątrz genów
Jeden gen znajduje się wewnątrz intronu innego genu – stosunkowo powszechna cecha genomów
jądrowych. Gen NF1 (neurofibromina 1) zawiera w obrębie swojej sekwencji intronowej trzy inne,
mniejsze geny: OGMP, EVI2B i EVI2A. Geny te ulegają transkrypcji w kierunku przeciwnym niż gen
NF1.
Pojęcie nici sensownej i antysensownej.
5. Pozagenowy DNA
sekwencje unikatowe
sekwencje powtórzone (repetytywne): rozproszone i tandemowe (zwarte)
Sekwencje powtórzone tandemowe (zwarte)
Satelitarne – większość powtórzeń występuje w regionach centromerowych chromosomu,
odgrywając tam prawdopodobnie funkcję strukturalną; powtórzenia dochodzą do setek tysięcy par
zasad:
- minisatelitarne – fragmenty długości sięgającej 20 kb, z powtarzająca się jednostką
długości około 10-100 bp; występują najczęściej w telomerach lub w pobliżu końców chromosomu;
funkcja nieznana
- mikrosatelitarne – powtórzenia krótkie, zwykle poniżej 150 bp, a jednostka powtórzona to
najczęściej 2 - 4 bp; funkcja nieznana, ale powtórzenia tego typu znalazły zastosowanie jako
marker molekularny (np. ustalanie profilu genetycznego – kryminalistyka, wykluczenie ojcostwa,
diagnostyka chorób genetycznych).
Sekwencje powtórzone rozproszone (transpozony i retrotranspozony)
- Krótkie SINE (ang. short interspersed nuclear element) - krótkie rozproszone
sekwencje jądrowe o długości około 100-400 bp. Najbardziej znana to sekwencja Alu o
wielkości 300 bp, która powtarza się ok. miliona razy i tworzy 10% ludzkiego DNA.
- Długie LINE (ang. long interspersed nuclear element) - długie rozproszone sekwencje
jądrowe; sekwencje o długości około 6 kb. Najbardziej znana to sekwencja LINE-1
powtórzona ok. 800 tys. razy, zajmująca 21% ludzkiego DNA.
6. Mapowanie genomu
Mapowanie polega na opracowaniu szczegółowych map chromosomów, które odzwierciedlają
położenie znanych genów, jak również sekwencji DNA o nieznanej funkcji w genomie.
Mapowanie genomu opiera się na dwóch metodach:
- mapowaniu fizycznym
- mapowaniu genetycznym
Mapowanie fizyczne:
• Mapy fizyczne tworzy się za pomocą sklonowanych odcinków DNA, których funkcja w
organizmie nie musi być znana.
• W odróżnieniu do map genetycznych uwzględniają takie szczegóły molekularne, jak: eksony,
introny, sekwencje regulatorowe itp.
• Jednostką mapowania fizycznego jest para zasad (ang. base pair)- bp.
Mapowanie genetyczne:
• Podstawą mapowania genetycznego jest zjawisko rekombinacji
• Mapy genetyczne sporządza się, analizując sprzężenia na podstawie oceny częstości
rekombinacji pomiędzy genami leżącymi na tym samym chromosomie.
• Im bliżej siebie leża dwa geny, tym mniejsza szansa, że będą one rozdzielone podczas
rekombinacji (crossing-over), dlatego współczynnik rekombinacji może być używany jako
miernik odległości między genami
• Współczynnik rekombinacji wynoszący 1% (1c-o na 100 podziałów), nazywany jest
centymorganem (cM)
7. Sekwencjonowanie genomu człowieka
Cele:
skonstruowanie szczegółowych map fizycznych i genetycznych całego genomu człowieka,
zlokalizowanie wszystkich genów w obrębie genomu człowieka
wypracowanie metod przechowywania i udostępniania uzyskanych danych; ulepszenie metod
sekwencjonowania
uzyskanie wiedzy na temat skutków społecznych, ideologicznych i etycznych nowych
technologii genetycznych
Fakty:
2003 – kompletna sekwencja genomu człowieka
2007 – opublikowano sekwencję genomu Craiga Ventera
2007 - James Watson - pierwszy „osobisty” genom (1 milion dolarów, 2 miesiące,
sekwencja opublikowana: http://jimwatsonsequence.cshl.edu/cgiperl/gbrowse/jwsequence/, za wyjątkiem genu ApoE)
2008 – pierwszy zsekwencjonowany genom osoby pochodzącej z Europy oraz pierwszy
genom kobiety, Holenderki dr Marjolein Kriek (40.000 Euro, 6 miesięcy)
2008 – opublikowano sekwencje genomu Azjaty (Chińczyka Han) oraz Nigeryjczyka
(plemię Yoruba)
11 marca 2009 - Personal Genome Project – „Osobiste Genomy”
- cel - zsekwencjonowanie genomów 100 000 ochotników
„PG-10” – pierwszych 10 ochotników, których dane są ogólnie dostępne
http://www.personalgenomes.org/mission.html
Sekwencjonowanie genomu - przyszłość
The Personal Genome Sequencing Service
10 czerwca 2009 - Illumina - amerykańska firma z obszaru life-science, zajmująca się
sekwencjonowaniem, genotypowaniem i ekspresją genów, zaoferowała możliwość
komercyjnego sekwencjonowania genomu za kwotę mniejszą niż 50 000 dolarów (dane
zawierałyby informacje o polimorfizmach typu SNP oraz CNV)
Dr Jay Flatley (President of Illumina) - przewiduje, że w 2019 roku sekwencjonowanie
genomu będzie rutynową techniką wykonywaną tuż po urodzeniu dziecka, a cena
sekwencjonowania genomu spadnie poniżej 1000 dolarów
8. Mutacje
Mutacją nazywamy utrwaloną zmianę w materiale dziedzicznym. Zmiany w DNA mogą być
różnego typu (w zależności, jakiej części genomu dotyczy). Mutacje mogą być dla organizmu
korzystne, neutralne oraz szkodliwe (przykłady).
9. Typy chorób genetycznych (w zależności od rodzaju oraz obszaru uszkodzenia
materiału genetycznego)
1.
2.
3.
4.
5.
6.
Spowodowane aberracjami chromosomowymi (13,18,21, X, XXY)
Spowodowane mutacją pojedynczego genu (choroby jednogenowe)
Choroby mitochondrialne
Mikrorearanżacje (np. zespoły mikrodelecji)
Zaburzenia piętnowania genomowego (Angleman, Prader-Willi)
Uwarunkowane wieloczynnikowo
10. Aberracje chromosomowe
Mutacje o dużym zasięgu występowania (dostępne do badania technikami mikroskopii świetlnej),
dotyczące dużego obszaru DNA .
Można je podzielić na:
liczbowe (odchylenie od normalnej liczby chromosomów)
strukturalne (zmienia się struktura chromosomów
Liczbowe aberracje chromosomowe
Poliploidia (3n,4n itp.)
- Triploidia (69,XXX,XXY lub XYY)
Aneuploidia (autosomy) (2n+1, 2n-1 itp.)
- Nullisomia (utrata pary chromosomów
homologicznych)
- Monosomia (utrata jednego chromosomu)
- Trisomia (jeden chromosom dodatkowy)
1-3% wszystkich poczęć,
w większości przypadków
poronienia samoistne
letalne przed implantacją
letalne w stadium embrionalnym
zwykle letalne, z wyjątkiem
trisomii chromosomów 13, 18, 21
Aneuploidia (chromosomy płci)
- Dodatkowy chromosom płci
(XXY,XXX,XYY)
- Utrata chromosomu płci (45,X)
normalna długość życia
99% przypadków-poronienia
samoistne, normalna inteligencja,
bezpłodność
Strukturalne aberracje chromosomowe
Zrównoważone:
Translokacje (translokacje wzajemne, fuzje centryczne)
Inwersje
Niezrównoważone:
Duplikacje
Delecje
Chromosomy pierścieniowe
Izochromosomy
Fragmenty centryczne (chromosomy markerowe)
11. Choroby uwarunkowane jednogenowo
Zmiany materiału genetycznego są submikroskopowe, dostępne do badania jedynie metodami biologii
molekularnej
Mutacje dotyczą tylko jednego genu.
(Mukowiscydoza, Hemofilia, Głuchota wrodzona, Rdzeniowy zanik mięśni, Zespół łamliwości
chromosomu X, Dystrofia mięśniowa Duchenne’a, Hemochromatoza, Albinizm, Fenyloketonuria,
Galaktozemia, Zespół Retta, Anemia sierpowata i inne)
Mutacje genowe – etiologia chorób jednogenowych
Mutacje genowe - zmiany normalnej sekwencji DNA organizmu, spowodowane błędami w
replikacji DNA (mutacje spontaniczne) lub działaniem czynników chemicznych i fizycznych
(mutacje indukowane).
Zachodzą w zygocie, płodzie, komórkach somatycznych i rozrodczych w ciągu całego życia. Mutacje
w komórkach somatycznych (mutacje somatyczne) nie są przekazywane potomstwu, w odróżnieniu od
mutacji w komórkach rozrodczych (mutacje germinalne), które mogą zostać potomstwu przekazane.
Mutacje somatyczne odgrywają dużą rolę w rozwoju nowotworów u ludzi. Mutacje w komórkach
rozrodczych mogą być odziedziczone lub powstają de novo w procesie oogenezy lub spermatogenezy.
Mutacje spontaniczne powstają w wyniku:
błędów replikacyjnych
poślizgu replikacyjnego
powstawania struktur trzecio- i czwartorzędowych
Częstość mutacji jest niejednakowa dla różnych miejsc w genie (loci). Mutacje spontaniczne mogą
powstać z większą częstością w charakterystycznych miejscach genu zwanych gorącymi miejscami (z
ang. hot spots).
Mutacje indukowane:
Mutageny chemiczne
Mutageny fizyczne
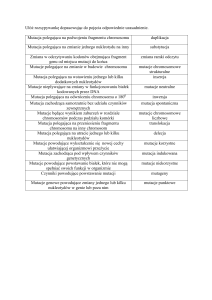
Typy mutacji genowych:
duże zmiany genowe (więcej niż jeden nt)
mutacje punktowe (dotyczące jednego nt)
mutacje transkrypcyjne (regiony regulatorowe)
mutacje splicingowe (GUAG)
mutacje dynamiczne
Duże zmiany genowe:
delecje
duplikacje
insercje
Delecje:
Utrata części sekwencji DNA (alfa-talasemia, rdzeniowy zanik mięśni, rodzinna
hipercholesterolemia, dystrofia mięśniowa Duchenne’a i Beckera, mukowiscydoza, zespół Pradera
Willi’ego).
Delecja małego fragmentu eksonu 44 genu dystrofiny powoduje ciężką formę DMD, natomiast duża
delecja, która zajmuje ponad połowę całego genu, powoduje łagodniejszą chorobę – BMD. Efekt
mutacji zależy więc nie od wielkości delecji, ale od tego czy delecja ta zaburza ramkę odczytu, czy też
nie.
Duplikacje:
Powielenie sekwencji DNA (rodzinna hipercholesterolemia, dystrofia mięśniowa Duchenne’a, zespół
Westa).
Insercje:
Wbudowanie dodatkowej sekwencji DNA (np. hemofilia, mukowiscydoza).
Przykład: W obrębie wstawionego insertu może znajdować się kodon stop, co w konsekwencji
spowoduje syntezę skróconego białka, które okaże się być niefunkcjonalne.
Mutacje transkrypcyjne:
Występują w regionie genu związanym z regulacją procesu transkrypcji, czego efektem jest spadek
produkcji białka (np. polidaktylia - mutacja w rejonie regulatorowym genu SHH).
Mutacje splicingowe
Położone na złączach intron - ekson, zakłócające proces wycinania intronów (np.
fenyloketonuria, beta-talasemia, mukowiscydoza).
Mutacje punktowe
Dotyczą tylko jednego nukleotydu i są najczęstszymi mutacjami w genomie ludzkim.
Typy mutacji punktowych:
insercje - wbudowanie dodatkowego nukleotydu
delecje - wypadnięcie nukleotydu (głuchota wrodzona; 35delG, zespół Marfana)
tranzycje - zamiana puryny na purynę lub pirymidyny na pirymidynę
o (achondroplazja, mukowiscydoza)
transwersje - zamiana puryny na pirymidynę lub odwrotnie (achondroplazja, anemia
sierpowata)
Przykład:
Achondroplazja - substytucje w genie FGFR3 (ang. Fibroblast Growth Factor Receptor 3) w pozycji
nukleotydu 1138:
tranzycja – G>A u 98% pacjentów
transwersja - G>C u 1% pacjentów
80% -mutacja de novo na chromosomie 4 dziedziczonym od ojca
Mutacje dynamiczne:
Pierwszą mutację dynamiczną odkryto w 1991 roku (gen FMR1)
Mutacje dynamiczne stwierdzono tylko u człowieka.
Mutacje dynamiczne wykazują dużą zmienność populacyjną.
Mutacje dynamiczne stanowią podłoże molekularne 14 jednostek chorobowych
mutacje polegające na wzroście liczby powtórzeń (3-, 4-, 12- nukleotydowych)
ilość powtórzeń może wzrastać w kolejnych pokoleniach
-
antycypacja-objawy choroby zaczynają objawiać się coraz wcześniej i w coraz
większym nasileniu w kolejnych pokoleniach
-
premutacja – wzrost ilości powtórzeń poniżej wartości granicznej – nie powoduje
wystąpienia choroby – bezobjawowi nosiciele.
Choroby: choroba Huntingtona (CAG), zespół łamliwego chromosomu X (CGG), ataksje módżkowordzeniowe, rdzeniowo-opuszkowy zanik mięśni, dystrofia miotoniczna.
Zespół łamliwego chromosomu X (FraX)
Mutacja dynamiczna w genie FMR1 w długim ramieniu chromosomu X
Wzrost ilości powtórzeń sekwencji CGG powyżej 200
Osoby zdrowe - do 54 powtórzeń, nosiciele bezobjawowi - do 200 powtórzeń (tzw.
premutacja)
Skutki mutacji genowych:
mutacje missens (zmiany sensu) – zmiana nukleotydu w pierwszej lub drugiej pozycji
kodonu powoduje zmianę kodowanego przez triplet aminokwasu. Zmienia się aminokwas w
białku
mutacje neutralne (ciche, synonimiczne) – nie powodują zmiany kodowanego aminokwasu,
ponieważ zmiana nukleotydu następuje w trzeciej pozycji tripletu lub też mutacja nie dotyczy
sekwencji kodujących lub z nimi związanych. mutacje nonsens (stop) – w wyniku mutacji
(delecji, insercji lub substytucji nukleotydu/ów) powstaje przedwcześnie kodon terminacyjny
„stop”, czego efektem jest białko skrócone i pozbawione funkcji
mutacje zmiany ramki odczytu - delecja lub insercja w rejonie kodującym (nie będąca
wielokrotnością trzech nukleotydów) genu zmienia wszystkie występujące za mutacją kodony;
następuje zmiana sekwencji aminokwasowej – zmienia się kodowane białko
mutacje transkrypcyjne – występują w obszarze regulującym proces transkrypcji czego
efektem jest spadek lub wzrost produkcji białka (np. polidaktylia - mutacja w rejonie
regulatorowym genu SHH)
mutacje splicingowe – zaburzenia w składaniu RNA (np. beta-talasemia).
Skutki mutacji ze względu na funkcję białka:
Utrata funkcji – mutacja znosi lub zmniejsza aktywność białka; większość mutacji tego
typu jest recesywna, ale zdarzają się sytuacje, w których mutacja ma charakter dominujący
(np. zespół Marfana, geny supresorowe nowotworów – BRCA1).
Nabycie funkcji – mutacja nadaje białku nietypową aktywność, często niepożądaną lub
toksyczną, albo aktywność w niewłaściwej tkance czy okresie rozwoju; mutacje tego typu
występują rzadziej i mają najczęściej charakter dominujący (np. protoonkogeny).
Mutacje znoszące
Istnieją takie mutacje, które znoszą działanie innych, wcześniejszych mutacji. Na przykład pierwsza
mutacja powoduje utratę funkcji kodowanego białka, a kolejna znosi tę pierwszą mutację i przywraca
wytwarzanie funkcjonalnego białka lub maskuje jego brak. Obie mutacje mogą zachodzić w tym
samym regionie DNA lub w zupełnie odległych od siebie miejscach.
14. Polimorfizm, a mutacje
Polimorfizm genetyczny oznacza występowanie w populacji dwóch lub więcej form danego genu
— alleli z częstością większą niż oczekiwana, wynikającą z ogólnej częstości mutacji w danej
populacji. Ponieważ częstość mutacji w danej populacji jest trudna do sprecyzowania, przyjęto, że o
polimorfizmie można mówić, gdy najrzadszy wariant alleliczny w danym locus występuje z częstością
większą niż 1%. Polimorfizm, podobnie jak mutacja, jest efektem zmian sekwencji DNA.