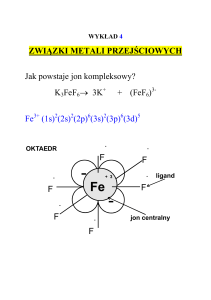
Wykład 3-4
Materiały z wykładów (plik PDF):
http://www.biomodellab.eu/lecture/
Porównanie zakresów modelowania
układów biologicznych
Symulacje mezoskopowe,
jedno ziarno = kilkanaście aminokwasów,
ziarna mogą być w różnych kształtach.
Symulacje gruboziarniste,
np. jedno ziarno = jeden aminokwas
Symulacje pełnoatomowe,
all-atom, united-atom (CH2, CH3 jako united atoms)
Symulacje kwantowe
Elastic Network Model
do badania drgań własnych w białkach
heksokinaza
Szybkie obrazowanie
ruchu dużych domen
białka
Drgania własne (normal modes)
najwolniejsze ruchy obu domen
Zamykanie miejsca aktywnego
Drgania własne heksokinazy
460 aminokwasów, 3350 atomów, ponad 10.000 drgań własnych (3N-6)
Mapowanie pola siłowego pełnoatomowego
(all-atom) na gruboziarniste (coarse-grain)
„Gruboziarnista” cząsteczka
rozpuszczalnika
Krok czasowy obliczeń = 20 - 40 fs
mapowanie 4 to 1 (średnio)
Polar (P), nonpolar (N), apolar (C), charged (Q)
1 - 5 : low - high polarity
d - donor, a - acceptor, da - both, 0 - none
Marrink et al., J. Phys. Chem. B 2007
Gruboziarnista reprezentacja aminokwasów
Porównanie wyników symulacji:
all-atom (50 ns GROMACS ffgmx)
i coarse-grain (4 μs CHARMM martini)
Monticelli et al., J. Chem. Theory and Comput. 2007
Samorzutne tworzenie dwuwarstwy lipidowej
Samoorganizacja błony DPPC
ze wstawieniem białka OmpA
(poryna)
Samoorganizacja błony DPPC i
utworzenie dimeru glikoforyny
Bond & Sansom, JACS 2006
Formowanie się domen z różnych rodzajów lipidów
skala
5 nm
2x2
2x2
0.42 : 0.28 : 0.3
0.28 : 0.42 : 0.3
diC16-PC / diC18:2-PC / cholesterol
Risselada et al. (Marrink group), PNAS 2008
Usuwanie lipidów z krwi przez granule HDL
symulacja gruboziarnista
Struktura
nanodysku
AY Shih et al. (Schulten group), J. Struct. Biol. 2007
Ewolucyjne zachowanie sekwencji i struktury
modelowanie przez homologię
Modelowanie białek przez homologię
Rodzina receptorów GPCR
Struktura receptora GPCR (2AR) z ligandem
Związek między strukturą a funkcją białka
może dotyczyć tylko miejsca aktywnego
Proteazy – przecinają łańcuchy peptydowe
Identyczne ułożenie kluczowych aminokwasów
w miejscu aktywnym: Ser, His, Asp
Mechanizm hydrolizy peptydów
przez chymotrypsynę
Modelowanie rozpoznawania
w układzie lek-receptor
rok
model
autor
1890
klucza i zamka
(lock-and-key)
komplementarność
Emil Fischer
1958
indukowanego dopasowania
(induced fit)
tolerancja
Daniel Koshland
2003
zespołu konformacji
(ensemble of conformations)
różnorodność
Buyong Ma et al.
Wszystkie modele są złe, ale niektóre są użyteczne
(George Box)
Zespół konformacji białkowych
(ensemble of conformations)
Ligand wiąże się z wybraną konformacją białka
Zespół konformacji białkowych
(rozpoznawanie białko-białko)
Uzupełnianie się obydwu metod rozpoznawania
Ubikwityna nawet bez partnerów białkowych przyjmuje te wszystkie konformacje „związane”
Profile energii dla zespołu konformacji
i indukowanego dopasowania
E
Białko może akceptować różne ligandy ale w różnym stopniu
Zmiana profilu (powierzchni) energii białka
w czasie wiązania liganda
Strategie projektowania leków
Ligand-based drug design
nieznana
• Budowanie modelu miejsc aktywnych
liganda (farmakofor)
• Przeszukiwanie baz danych (screening)
• 1D i 3D QSAR
(pseudoreceptory i pola molekularne)
Struktura
celu
molekularnego
• Dopasowanie ligandów do miejsca
aktywnego receptora (dokowanie)
znana
Receptor-based drug design
• Budowa nowych ligandów ab-initio
• Dynamika kompleksu receptor-ligand
Modyfikacje liganda w celu zwiększenia
oddziaływania z celem molekularnym
• Struktura kompleksu nie jest znana
– Poszukiwanie miejsca aktywnego (informacja biochemiczna,
analiza mutacji lub wnęki/rowki na powierzchni receptora)
– Dokowanie znanych ligandów tego białka lub przeszukiwanie
całych bibliotek różnych ligandów (dokowanie w różnych miejscach)
• Struktura kompleksu jest znana
– Analiza istniejących oddziaływań lek-białko
– Maksymalizacja liczby i siły oddziaływań
– Ocena efektów entropowych (zmniejszenie liczby konformacji,
schowanie części hydrofobowych)
Yasara: TransPept, AChE
Analiza oddziaływań
w kompleksie biotyna-streptoawidyna
Silne wiązanie biotyny (witamina B7)
KD 10-14 mol/dm3 (0.01 pM)
Streptoawidyna jest uzyskiwana
z bakterii Streptomyces avidinii.
Znalazła zastosowanie w biotechnologii do
oczyszczania białek (pI 7 więc nie wiąże
się niespecyficznie z innymi białkami).
Biotyna-awidyna nawet 0.001 pM ale dla
awidyny pI 10.
Analiza oddziaływań
w kompleksie biotyna-streptoawidyna
Yasara - analiza
Kataliza enzymatyczna
Model miejsca aktywnego w enzymie:
rowek, zagłębienie lub kanał
Inhibitory współzawodnicze (odwracalne):
Glikol etylenowy (przypadkowe zatrucie)
blokuje dehydrogenazę alkoholową:
Inhibitor wiąże się silniej z enzymem i zatyka go.
Możliwość regulacji siły wiązania.
Usuwane przez nadmiar naturalnego substratu
Sulfonamidy blokują enzymy bakteryjne,
antycholinesterazy - enzym u ssaków acetylochlinesterazę
Inhibitory niewspółzawodnicze, odwracalne
(allosteryczne):
Drugie miejsce aktywne istnieje w większości enzymów
do kontroli ich działania przez komórkę:
6-merkaptopuryna
stosowana w leczeniu białaczki
- inhibituje allosterycznie pierwszy enzym w łańcuchu syntezy puryn
i synteza puryn zostaje zahamowana.
To z kolei blokuje syntezę DNA
Inhibitory niewspółzawodnicze (nieodwracalne)
-OH (Ser)
-SH (Cys)
Gazy bojowe toksyczne dla enzymów ssaków,
penicylina dla enzymów bakterii
Inhibitory będące analogami stanu przejściowego
Idealne w przypadku reakcji enzymatycznych wymagających dwu substratów
Biosynteza dTMP
5-fluorouracyl - stosowany w leczeniu raka piersi i skóry
- wykazuje dużą selektywność do komórek rakowych.
Jest przekształcany w organizmie do fluorowego analogu dUMP
5-fluorouracyl - działanie
Przeciwdziałanie
związaniu kofaktora
dUMP
Reakcja jest niemożliwa gdyż wymaga oderwania F+.
Enzym jest kowalencyjnie związany w stanie przejściowym
- blokada syntezy DNA - replikacja i podział komórek zahamowany
Inhibitor jest wytwarzany dopiero w miejscu reakcji enzymu
i dlatego jest wysoce selektywny
Dimer TS
Tomudex
Receptory komórkowe
neurotransmitery
Funkcje kontrolne i komunikacyjne.
Terapia bólu, depresji, psychoz, choroby Parkisona,
chorób serca, astmy itp
Działanie receptorów
kanały jonowe
Receptor związany z enzymem
Zmiana kształtu przez receptor
działanie antagonisty
przykłady agonistów
działanie silniejsze lub słabsze
+ mniejsze lub większe efekty uboczne
+ inwersyjni agoniści
Czynniki wpływające na aktywność
właściwe położenie
grup wiążących
wielkość i kształt
liganda
Inne typy ligandów receptorów
agonista
allosteryczny
częściowy
agonista
efekt "umbrella"
Antybiotyki
• Blokujące enzymy bakteryjne
• Blokujące DNA
• Blokujące RNA (rybosom bakteryjny)
Elementy aktywne penicylin
Penicylina G sce
WebmedCentral.com
Amoksycylina
Z grupy penicylin, lek pochodny: Augmentin (amoksycylina i kwas klawulanowy – inhibitor -laktamazy)
Glaxo
Amoksycylina – blokuje
enzym transpeptydazę
(kroslinkowanie ściany
komórkowej bakterii z
peptydoglikanów – polimer
peptydowo-sacharydowy)
Schemat półsyntetycznej
produkcji penicylin
Amoksycylina zablokowana
por. miejsc wiążących transpept. i -laktam. sce przez enzym -laktamazę
Pfizer
Inhibitory -laktamazy
Monocykliczne antybiotyki
-laktamowe (odporne na
większość -laktamaz)
Elementy aktywne cefalosporyn
WebmedCentral.com
Cefaklor
z grupy cefalosporyn (grzyb Cephalosporium acremonium)
Porównanie struktur
Amoksycylina
Cefaklor
Etap syntezy cefalosporyn
(podobny do 6-APA w syntezie
penicylin)
Biosynteza penicylin przez
enzym zawierający żelazo
(niewykonalna chemicznie)
Kompleks Ceph R61 z transpeptydazą.
Ceph zastępuje peptydoglikan a Ser62 wiąże się
nieodwracalnie do -laktamu
por. sce
Przykłady cefalosporyn
4 generacje cefalosporyn
Ceftarolina – następna generacja cefalosporyn
Ceftarolina sce + ESP
Izoniazyd
Lek pierwszego rzutu w leczeniu gruźlicy (Mycobacterium tuberculosis)
Izoniazyd, INH, pochodna
kwasu izonikotynowego, prolek
Inne leki tego typu
Budowa ściany komórkowej
mykobakterii:
- Peptydoglikany
- Arabinogalaktany
- Kwas mykolowy
Woskopodobny składnik ściany
komórkowej bakterii tuberculosis
Działanie INH (wewnątrz bakterii)
KatG – katalaza
NADH – dinukleotyd nikotynoadeninowy
Powstaje inhibitor reduktazy zależnej
od NADH (uczestniczy w procesie
produkcji kwasu α-mykolowego)
Inhibicja InhA, mutacja S94A
prowadzi do oporności na INH
1ZID_2.mut.S94A.sce
Budowa DNA
Budowa od 5’ do 3’ końca
Struktura chemiczna DNA
B-DNA
Widełki replikacyjne DNA
źródło: pl.wikipedia.org
Distamycyna
przeciwdziała podziałowi komórek, silny antybiotyk i lek przeciwnowotworowy
Sekwencje kwadrupleksowe występują
w telomerach i miejscach regulatorowych.
Są formowane z jednej, 2 lub 4 nici polinukleotydów.
DNA_quadruplex_distamycin sce
Amikacyna
W poważnych infekcjach przeciwko bakteriom beztlenowym
Struktura molekularna
Struktura krystaliczna ze
związanym rRNA
YASARA SCE:
Związek naturalny
Przy oporności na
streptomycynę
2G5Q_amikacin
Cel leku: bakteryjna mała
jednostka 30S rybosomu –
inhibicja transkrypcji
Działanie rybosomu
50S
30S
Doksycyklina (Vibramycyna)
Antybiotyk tetracyklinowy, inhibitor syntezy białek w rybosomie
Doksycyklina
30S
Tetracykliny > 30.000 ton rocznie.
Dodawane do karmy dla kurcząt i bydła
aby zwiększyć ich masę. Obecnie znanych
ponad 2000 analogów
Wiązanie z miejscem syntezy białka w
podjednostce 30S rybosomu. Przeciwdziała
związaniu tRNA, który dostarcza aminokwasy.
Tetracykliny aktywne przeciwko
leko-opornym bakteriom
1I97_doksyklina sce
Azitromycyna
Antybiotyk makrolidowy (makropierścień zawierający grupę estrową)
Pierwszy odkryty lek
z tej grupy (naturalny
– z bakterii ziemnych)
Bardziej odporna na
środowisko kwaśne
Miejsce wiążące leku w podjednostce
50S
rybosomu.
Blokuje
kanał
wyjściowy dla tworzonego białka
Azitromycyna,
Długożyciowa, jedna dawka dożylna
wystarcza do wyleczenia infekcji
1NWY.sce
Przyczyny oporności bakterii na antybiotyki
• Enzymy niszczące leki (np. -laktamazy)
• Mutacje w bakteryjnych celach molekularnych (np. w rybosomie)
• Zwiększenie ilości białek błonowych eksportujących leki (pompy
wydzielnicze) np. dla leków tetracyklinowych
• Zmiany genetyczne modyfikujące metabolizm (np. bakterie uzyskujące
kwas foliowy od gospodarza)
• Zmniejszenie przepuszczalności ścian komórkowych przez mutacje
(bakterie gruźlicy)
• Tworzenie błon ochronnych (biofilmy) utrudniające wnikanie
antybiotyków i leków przeciwwirusowych.
Oporność powstaje bardzo szybko gdy lek stosowany jest oddzielnie.