Warszawski
Uniwersytet
Medyczny
Wydział
Farmaceutyczny
Oddział
Analityki
Medycznej
Justyna
Krystyna
Ciepły
Nr
albumu
41624
Charakterystyka
lekoopornych
szczepów
wirusa
HIV‐1
izolowanych
w
Polsce
w
2008
roku
Praca
magisterska
wykonana
w
Zakładzie
Mikrobiologii
Farmaceutycznej
Warszawskiego
Uniwersytetu
Medycznego
pod
kierownictwem
naukowym
prof.
dr
hab.
Stefana
Tyskiego
Opiekunowie
pracy:
Dr
n.
farm.
Bohdan
Starościak
(Zakład
Mikrobiologii
Farmaceutycznej
Warszawskiego
Uniwersytetu
Medycznego)
Dr
n.
med.
Janusz
J.
Stańczak
(Pracownia
Diagnostyki
Molekularnej
Wojewódzkiego
Szpitala
Zakaźnego
w
Warszawie)
Wirus
HIV‐1
• Zidentyfikowany
w
1983
roku
przez
L.
Montagnier,
następnie
w
1984
roku
przez
R.
Gallo
• Rodzina
Retroviridae, grupa Len.virus complex • Ma
kształt
kulisty,
średnicę
100
nm
• Genom
to
dwie
cząsteczki
jednoniciowego
+ssRNA
• Geny
gag (białka
strukturalne), pol (białka
enzymatyczne), env (białka
otoczki) oraz
tat,
rev,
nef,
vif,
vpu,
vpr (regulatorowe)
• Enzymy:
1
cząsteczka
proteazy,
1
integrazy i
2
odwrotnej
transkryptazy
Wirus
HIV‐1
• Do
wniknięcia
do
komórki
wymaga
cząsteczki
CD4+
(receptor)
oraz
CCR5
lub
CXCR4
(koreceptory)
• Szczepy
M‐tropowe
korzystające
z
CCR5
• Szczepy
T‐tropowe
korzystające
z
CXCR4
Wirus
HIV‐1
–
główne
grupy
genetyczne
• M
(major)
Grupa
M
to
9
subtypów:
A
–
D,
F
–
H,
J
i
K
• O
(outlier)
• N
(non‐M,
non‐O)
• P
(
wyizolowany
w
2009
roku
od
kobiety
z
Kamerunu
mieszkającej
we
Francji)
• Występują
formy
zrekombinowane
–
CRF
(Circulajng
Recombinant
Forms)
–
48
form
np.
CRF01_AE,
CRF02_AG,
CRF05_DF,
CRF07_BC
Leki
antyretrowirusowe
• Nukleozydowe
(NtRTI)
i
nukleotydowe
(NRTI)
inhibitory
odwrotnej
transkryptazy
• Nienukleozydowe
inhibitory
odwrotnej
transkryptazy
(NNRTI)
• Inhibitory
proteazy
(PI)
• Inhibitory
fuzji
i
wejścia
(EI)
• Inhibitory
integrazy
(II)
• Nowe
preparaty
–
inhibitory
dojrzewania
(III
faza
badań
klinicznych)
Metody
oznaczania
lekooporności
• Fenotypowanie
(w
tym
również
fenotypowanie
wirtualne)
• Genotypowanie
– Hybrydyzacja
– Sekwencjonowanie
Czynniki
wpływające
na
powstanie
lekooporności
wirusa
HIV‐1
• Wysokie
tempo
replikacji
(dziennie
syntetyzowanych
jest
1010
–
1011
nowych
wirionów)
• Niedokładność
wirusowej
odwrotnej
transkryptazy
• 1
–
5x109
wirionów
syntetyzowanych
w
ciągu
24
h
zawiera
mutacje
w
regionach
kodujących
odwrotną
transkryptazę
i
proteazę
• Niewystarczająca
współpraca
pacjenta
z
lekarzem
• Akumulacja
mutantów
w
czasie
zakażenia
Oznaczanie
lekooporności
–
(zalecenia
EACS)
• Pacjenci
nowozdiagnozowani
• Pacjenci
rozpoczynający
leczenie
‐
o
ile
nie
byli
testowani
wcześniej.
Należy
użyć
najwcześniej
pobraną
próbkę
krwi
przed
rozpoczęciem
terapii.
• Pacjenci
z
niepowodzeniem
leczenia
(wirusologicznym)
• Kobiety
ciężarne
przed
rozpoczęciem
terapii
(dla
efektywnej
terapii
i
ochrony
dziecka
przed
zakażeniem)
• Profilaktyka
poekspozycyjna
Cel
pracy
• wykrycie
mutacji
odpowiedzialnych
za
oporność
wirusa
HIV‐1
na
leki
• ocena
częstości
występowania
w
Polsce
wariantów
genetycznych
HIV‐1
o
obniżonej
podatności
na
leki
• wykrycie
przyczyn
niepowodzeń
terapeutycznych
wśród
pacjentów
leczonych,
• umożliwienie
prawidłowego
doboru
następnego
schematu
leczenia
(nowych,
aktywnych
wobec
mutantów
leków)
• ocena
występowania
subtypów
HIV‐1
w
Polsce
• ocena
właściwości/przydatności
metod
sekwencjonowania
ViroSeq
i
Trugene
Przygotowanie
materiału
• Zbadano
285
próbek
–
w
tym
91
od
chorych
leczonych,
194
od
nieleczonych
• Materiałem
wyjściowym
do
badań
było
osocze
• Przed
przystąpieniem
do
sekwencjonowania
materiał
przygotowywano
w
następujących
etapach:
a)
Izolacja
RNA
wirusa
HIV‐1
b)
Odwrotna
transkrypcja
c)
Reakcja
PCR
d)
Oczyszczanie
DNA
na
kolumienkach
e)
Elektroforeza
DNA
w
żelu
agarozowym
f)
PCR
sekwencyjny
g)
Oczyszczanie
DNA
Metody
sekwencjonowania
• ViroSeq
HIV‐1
Genotyping
System
v2.6
(Abbos,
Niemcy)
‐
sekwencjonowany
jest
region
kodujący
proteazę
wirusa
(kodony
1
–
99)
oraz
2/3
regionu
kodującego
odwrotną
transkryptazę
(kodony
1
–
335)
Metody
sekwencjonowania
• Test
Trugene
HIV‐1
Genotyping
Assay
(Siemens,
Niemcy)
‐
określana
jest
sekwencja
regionu
proteazy
wirusa
(kodony
1
–
99)
oraz
odwrotnej
transkryptazy
wirusa
(kodony
40
–
247)
Przydatność
metod
stosowanych
do
oznaczania
lekooporności
wirusa
HIV‐1
Metoda
ViroSeq
Zautomatyzowana
Metoda
Trugene
Półautomatyczna
Wykrywa
67
mutacji
Wykrywa
72
mutacje
Etapy
dodatkowe
Brak
etapów
dodatkowych
w
przygotowaniu
materiału
w
przygotowaniu
materiału
Konieczna
interwencja
eksperta,
nie
wykrywa
insercji
i
delecji
Łatwiej
poszerzyć
oprogramowanie
o
wykrycie
nowych
mutacji
Trudniej
poszerzyć
oprogramowanie
o
wykrycie
nowych
mutacji
Wynik
sekwencjonowania
Pik zielony – adenina (A), czerwony – tymina(T), niebieski – cytozyna (C), czarny – guanina (G)
Subtypy
HIV‐1
zidentyfikowane
w
Polsce
w
2008roku
1,4%
0,4%
3,9%
0,7%
1,8%
91,9%
B
C
D
G
CRF01_AE
CRF02_AG
Wyniki
Najczęściej
wykrywane
mutacje
Leki,
na
które
wystąpiła
największa
oporność
Chorzy
leczeni
Chorzy
nieleczeni
NRTI
i
NtRTI:
M184V
PI:
L10I,
L90M,
L76LV
NNRTI:K103N,
Y181C,
E138A
NRTI
i
NtRTI:
M41L,
T215S
i
M184V
PI:
L10I,
L10IL
NNRTI:
G190A
NNRTI:
newirapina,
delawirdyna,
efawirenz
PI:
nelfinavir,
sakwinawir,
indinawir
NRTI
i
NtRTI:
lamiwudyna,
emtrycytabina
PI:
nelfinawir
NRTI
i
NtRTI:
azydotymidyna,
stawudyna
NNRTI:
newirapina
Wyniki
–
pacjenci
leczeni
• Wśród
91
pacjentów
leczonych
szczepy
lekooporne
HIV‐1
stwierdzono
u
35
(38,5%)
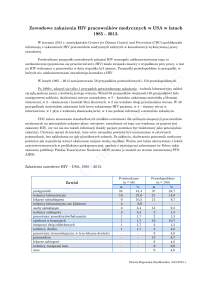
Częstość
występowania
wariantów
opornych
HIV‐1
35%
30%
25%
20%
15%
10%
5%
0%
PI
NRTI
i
NtRTI
Grupy
leków
NNRTI
Wyniki
–
pacjenci
nieleczeni
• Wśród
194
pacjentów
szczepy
oporne
wirusa
HIV‐1
stwierdzono
u
15
(7,7%)
Częstość
występowania
wariantów
opornych
HIV‐1
5,0%
4,5%
4,0%
3,5%
3,0%
2,5%
2,0%
1,5%
1,0%
0,5%
0,0%
PI
NRTI
i
NtRTI
Grupy
leków
NNRTI
Częstość
występowania
wariantów
opornych
HIV‐1
wśród
pacjentów
nieleczonych
w
Polsce
30,0%
25,0%
20,0%
15,0%
28,7%
10,0%
14,1%
5,0%
5,8%
7,7%
3,9%
0,0%
2001‐2002
2002‐2005
2006
2007
2008
Częstość
występowania
wariantów
opornych
HIV‐1
wśród
pacjentów
leczonych
w
Polsce
80,0%
70,0%
60,0%
50,0%
40,0%
75,1%
76,7%
30,0%
52,6%
43,1%
20,0%
38,5%
10,0%
0,0%
2000‐2001
2002‐2005
2006
2007
2008
Podsumowanie
• Chorzy
leczeni
–
częstość
oporności
wirusa
HIV‐1
na
leki
–
38,5%
• Chorzy
nieleczeni
–
częstość
oporności
wirusa
HIV‐1
na
leki
–
7,7%
• Subtypy
HIV‐1
w
Polsce
w
2008
roku:
B(91,9%),
C,
D,
G,
CRF01_AE,
CRF02_AG
• Metoda
Trugene
mniej
złożona
pod
względem
przygotowania
materiału,
metoda
ViroSeq
bardziej
złożona
Dziękuję
za
uwagę