OPIS PROJEKTU
NOWY ALGORYTM IDENTYFIKACJI ZAKAŻEŃ MYCOBACTERIUM KANSASII
1.
AKTUALNY STAN WIEDZY
Termin „prątki niegruźlicze” (NTM, ang. non-tuberculous mycobacteria) lub „prątki inne niż
gruźlicze” (MOTT, ang. Mycobacteria other than tuberculosis) odnosi się do grupy różnych gatunków
prątków niezaliczanych do kompleksu Mycobacterium tuberculosis. Bakterie te występują
powszechnie w środowisku, głównie w glebie i zbiornikach wodnych. Izolowane są także z produktów
spożywczych oraz tkanek zwierzęcych [1]. Mimo, że są wolnożyjącymi saprofitami, mogą
w określonych warunkach wywoływać choroby u ludzi. Zwykle schorzenia wywołane prątkami
niegruźliczymi, tzw. mykobakteriozy rozwijają się u osób z dysfunkcją układu odpornościowego, na
przykład w wyniku leczenia nowotworów, terapii immunosupresyjnych, czy też u pacjentów
z HIV/AIDS. (Mykobakteriozy należą do tzw. chorób wskaźnikowych AIDS). Bardzo rzadko choroby
wywołane przez prątki NTM obserwowane są u osób immunokompetentnych. Mykobakteriozy
występują najczęściej w postaci zapalenia płuc, zapalenia węzłów chłonnych, infekcji skóry i tkanek
miękkich, a także zakażenia uogólnionego (szczególnie u pacjentów chorych na AIDS). Diagnostyka
zakażeń prątkami NTM jest trudna i często niejednoznaczna. Podobnie, trudne i kontrowersyjne jest
leczenie wymagające długotrwałego przyjmowania kombinacji leków, często źle tolerowanych przez
pacjentów [2, 3].
Podczas gdy częstość występowania gruźlicy w krajach rozwiniętych systematycznie maleje,
odsetek zakażeń prątkami NTM niepokojąco wzrasta. Wzrost liczby zachorowań wywołanych przez
prątki NTM wiązany jest w znacznej mierze z szerzącą się na całym świecie epidemią HIV/AIDS [4].
Skuteczne leczenie chorych zakażonych prątkami NTM wymaga szybkich i wiarygodnych metod
identyfikacji czynnika etiologicznego.
Mycobacterium kansasii należy do najczęściej izolowanych gatunków prątków NTM z materiałów
klinicznych i odpowiada za istotną część zakażeń prątkami niegruźliczymi w populacji ludzkiej.
Identyfikacja prątków niegruźliczych, w tym prątków M. kansasii przez długi czas opierała się
jednie na ocenie cech fenotypowych, głównie charakterystyce właściwości biochemicznych. Testy
fenotypowe są czasochłonne (zajmują do 2 miesięcy), a ich wyniki nie zawsze są jednoznaczne. Do
niedawna metodą referencyjną w identyfikacji gatunkowej prątków była analiza kwasów mykolowych
techniką
wysokociśnieniowej
chromatografii
cieczowej
(HPLC, ang.
high-pressure liquid
chromatography) [5]. Technika ta obciążona jest jednak wysokimi kosztami aparaturowymi oraz
koniecznością stosowania toksycznych rozpuszczalników organicznych.
Upowszechnienie metod biologii molekularnej znacznie usprawniło identyfikację prątków NTM
pozwalając choć w niewielkim stopniu przybliżyć epidemiologię wywoływanych przez nie zakażeń.
W ciągu ostatnich dwóch dekad, opracowanych zostało kilka metod identyfikacji opartych na analizie
DNA, które z powodzeniem stosowane są w różnicowaniu prątków M. kansasii od innych gatunków
prątków. W 1991 roku, Huang i wsp. opracowali pierwszą sondę DNA (pMK1-9) specyficzną dla
1
OPIS PROJEKTU
M. kansasii, zarówno szczepów klinicznych, jak i środowiskowych [6]. Jednak w badaniu
przeprowadzonym przez Ross i wsp. wykazano, że spośród 105 szczepów M. kansasii, 20 (19%) nie
dawało sygnału dla sondy pMK1-9. Co ciekawe, wszystkie, poza jednym, szczepy negatywne
w reakcji z sondą pMK1-9 zawierały odmienną sekwencję 16S rDNA, sugerując istnienie różnych
typów genetycznych w obrębie M. kansasii [7]. Analiza biblioteki genomowej szczepów M. kansasii
negatywnych w reakcji z sondą pMK1-9 wykazała obecność repetytywnego DNA, które
scharakteryzowano jako sekwencję insercyjną IS1652. Sekwencję tę wykryto wyłącznie w szczepach
negatywnych dla sondy pMK1-9, przy czym źródłem polimorfizmu miedzy szczepami była liczba
kopii sekwencji IS1652 w chromosomie (od 1 do 11) [8]. Kolejna sonda DNA, opisana przez Yang
i wsp., obecna na plazmidzie p6123, zdolna była do specyficznej hybrydyzacji z DNA wszystkich
badanych szczepów M. kansasii, także tych, które były negatywne w reakcji z sondą pMK1-9 [9].
Obecnie na rynku dostępne są 3 testy używane do identyfikacji M. kansasii (a także innych
gatunków należących do prątków NTM), których zasada opiera się na technologii sond DNA. Są to
test AccuProbe (Gen-Probe, San Diego, USA), wykrywający 16S rRNA, a także test INNO LiPA
MYCOBACTERIA v2 (Innogenetics, Ghent, Belgia) oraz Speed-oligo® Mycobacteria (Vircell, Santa
Fé Granada, Hiszpania), których celem jest region niekodujący ITS (ang. internal transcribed spacer),
między genami kodującymi 16S i 23S rRNA. Wszystkie te testy okazały się skuteczne w identyfikacji
najczęściej występujących gatunków prątków, w tym M. kansasii. (System Accuprobe identyfikuje
5 taksonów Mycobacterium, a ściślej prątki kompleksu M. tuberculosis oraz 4 gatunki prątków NTM:
M. avium, M. intracellulare, M. kansasii i M. gordonae. Z kolei testy INNO LiPA MYCOBACTERIA
i Speed-oligo® Mycobacteria wykrywają odpowiednio 17 i 14 różnych taksonów w oparciu
o kombinację sygnałów pochodzących z hybrydyzacji z 14 lub 6 sondami) [10-13].
Stosunkowo od niedawna dostępny jest nowy kompleksowy system wykorzystujący specyficzne
sondy DNA - GenoType Mycobacterium CM/AS (Hain Lifescience, Nehren, Niemcy). Zasada tego
systemu opiera się na amplifikacji, techniką PCR multiplex, swoistych gatunkowo regionów w obrębie
23S rDNA, a następnie hybrydyzacji produktów amplifikacji z odpowiednio zaprojektowanymi
oligonukleotydami, związanymi na pasku nitrocelulozowym. Dwa najważniejsze testy to test
GenoType Mycobacterium CM (ang. common mycobacteria) oraz GenoType Mycobacterium AS
(ang. additional species), które pozwalają na jednoczesną identyfikację 15 gatunków Mycobacterium
w tym M. tuberculosis, kompleksu M. avium, M. kansasii (CM) i 16 kolejnych mniej powszechnych
patogennych prątków NTM, w tym: M. simiae, M. mucogenicum, M. goodii i M. celatum (AS).
Użyteczność systemu GenoType Mycobacterium, jako szybkiego i skutecznego testu diagnostycznego
potwierdzono w kilku badaniach ewaluacyjnych [14-16].
Alternatywną wobec technik opartych o hybrydyzację DNA metodę identyfikacji kilkunastu
gatunków prątków NTM zaproponowali Plikaytis i wsp. W metodzie tej, amplifikuje się, techniką
PCR, fragment genu kodującego białko szoku cieplnego (hsp65) o długości ok. 1380 pz, a następnie
otrzymany produkt poddaje się analizie restrykcyjnej (PCR-RFLP, ang. PCR-restriction fragment
2
OPIS PROJEKTU
length polymorphism) [17]. Metodę tę zmodyfikowali Telenti i wsp. [18] i w oparciu o nią
zaproponowali algorytm identyfikacji umożliwiający różnicowanie 34, a później nawet 54 gatunków
prątków [19, 20]. W identyfikacji gatunkowej prątków NTM zastosowanie znalazły także inne niż
hsp65 geny metabolizmu podstawowego, m. in. dnaJ [21], rpoB [22] oraz tuf [23] kodujące
odpowiednio: stresowe białko opiekuńcze, podjednostkę β polimerazy RNA oraz czynnik elongacji Tu
(EF-Tu).
Badania przeprowadzone przez Ross i wsp. [7] oraz późniejsze, poświęcone analizie regionu ITS
[24], wykrywaniu sekwencji IS1652 [8], a także analizie PCR-RFLP genu hsp65 [18, 19], wykazały
zróżnicowanie genetyczne w obrębie M. kansasii. Polimorfizm genetyczny tego gatunku potwierdzono
w dwóch kompleksowych badaniach, w których zidentyfikowano 5 różnych typów genetycznych (IV). Picardeau i wsp., badali zarówno szczepy kliniczne (38), jak i środowiskowe (24) stosując analizę
reaktywności DNA z sondami wykrywającymi sekwencje IS1652 oraz powtórzone sekwencje typu
MPTR (ang. major polymorphic tandem repeat; rodzaj powtórzonego DNA, stanowiący serię krótkich
powtórzeń o długości 10 pz, występujący w szczepach M. kansasii, ale też M. tuberculosis
i M. gordonae [25]), a także przy pomocy techniki PFGE (elektroforeza pulsacyjna w zmiennym polu
elektrycznym; ang. pulsed-field gel electrophoresis), AFLP (polimorfizm długości amplifikowanych
fragmentów; ang. amplified fragment length polymorphism) oraz hsp65 PCR-RFLP. Wszystkie
metody, poza IS1652-RFLP, ujawniły wśród szczepów M. kansasii istnienie 5 różnych grup
(klasterów), definiowanych jako typy genetyczne. Podczas gdy metody MPTR-RFLP i hsp65 RFLP
generowały wzory specyficzne dla typów, analiza PFGE i AFLP wykazała dodatkowo zróżnicowanie
wewnątrz poszczególnych genotypów [26]. Tylko dwa typy (II i III) zawierały sekwencję IS1652
(odpowiednio w 1 i 4-6 kopiach) i zgodnie z wcześniejszymi ustaleniami Yang i wsp. [8], oba te typy
były negatywne w reakcji z sondą pMK1-9, dając również negatywny wynik w teście AccuProbe.
Wreszcie, wszystkie badane szczepy reagowały z sondą p6123 opisaną przez Yang i wsp. [9].
Wyniki przedstawione przez Picardeau i wsp. znalazły potwierdzenie w badaniu Alcaide i wsp.,
którzy zastosowali kilka markerów genetycznych w odniesieniu do próby 276 szczepów M. kansasii
pochodzących z 4 krajów europejskich (Szwajcarii, Hiszpanii, Niemiec i Włoch) [27]. Przy użyciu
analizy hsp65 RFLP, techniki PFGE oraz analizy sekwencyjnej regionu ITS, potwierdzono
występowanie 5 typów genetycznych w obrębie M. kansasii. Ponadto, typy II i III dały negatywny
wynik w teście AccuProbe i nie zawierały sekwencji kodującej inteinę genu gyrA [27]. (Obecności
sekwencji kodującej inteinę genu gyrA dowiedziono wcześniej dla typu I, ale nie dla typu II) [28].
Niedawno opisano dwa nowe typy genetyczne M. kansasii (typ VI i VII) [29, 30]. Chociaż
sekwencjonowanie regionu ITS oraz analiza hsp65 PCR-RFLP uważane są za jednakowo skuteczne
w genotypowaniu M. kansasii, okazuję się, że mogą pojawiać się rozbieżności między tymi dwiema
metodami. Iwamoto i Saito opisali 3 szczepy M. kansasii niosące sekwencję ITS specyficzną dla typu
II, i generujące jednocześnie nieopisany wcześniej profil hsp65 PCR-RFLP. Ponadto 4 inne szczepy
3
OPIS PROJEKTU
zawierały sekwencję ITS specyficzną dla typu II, generując przy tym profil hsp65 RFLP
charakterystyczny dla typu I. (Szczepy te oznaczono jako prezentujące tzw. „pośredni” typ I) [31].
W identyfikacji typów genetycznych M. kansasii (I-VI) skuteczną okazała się też metoda oparta
o analizę restrykcyjną amplifikowanego fragmentu genu rpoB o długości 342 pz [22].
W badaniu zróżnicowania genetycznego M. kansasii stosowano wiele markerów molekularnych.
Tylko niewielki wewnątrzgatunkowy polimorfizm wykazano przy użyciu techniki LRF-(ang. large
restriction fragment)-PFGE [26, 27, 32-34] i analizy AFLP [26, 35]. Badania przeprowadzone przez
Picardeau i wsp., pozwoliły wyróżnić 14 różnych wzorów LRF-PFGE wśród 60 szczepów
M. kansasii, w tym 4 wzory obserwowane były wśród 24 szczepów należących do typu I. W tym
samym badaniu, analiza AFLP przy użyciu jednego enzymu restrykcyjnego (PstI) nie ujawniła
zróżnicowania genetycznego wśród szczepów reprezentujących typ I [26]. W innym badaniu, analiza
AFLP z wykorzystaniem enzymu ApaI pozwoliła wyodrębnić wśród 253 szczepów M. kansasii typu I
12 odrębnych klasterów [35]. W badaniach Iinuma i wsp., przeprowadzono LRF-PFGE na próbie 84
szczepów klinicznych M. kansasii z Japonii. Wyróżniono 21 wzorów LRF-PFGE, stosując enzym
VspI oraz 16 wzorów stosując enzym SpeI [32]. Analizę PFGE wykorzystał też Zhang i wsp., którzy
wśród 71 szczepów opisali 28, 32 i 35 różnych wzorów, przy użyciu enzymów, odpowiednio: AseI
(izoschizomer VspI), DraI i XbaI, przy czym różnice między wzorami były niewielkie [33].
Znaczenie typowania genetycznego M. kansasii polega przede wszystkim na różnicowaniu typów
patogennych i środowiskowych (klinicznie obojętnych).
Obecnie diagnostyka zakażeń prątkami NTM, a zwłaszcza tych wywołanych przez M. kansasii
posiada istotne ograniczenia. Po pierwsze, wszystkie dostępne testy identyfikacji są zależne od
hodowli i wymagają uzyskania kultury bakteryjnej (na podłożu płynnym lub stałym). To z kolei
znacząco wydłuża czas i zwiększa koszty analizy. Po drugie, brak obecnie jakichkolwiek
komercyjnych testów pozwalających na identyfikację poznanych dotąd genotypów M. kansasii.
(Zestaw INNO LiPA rozpoznaje typ I i II, nie różnicując między typem III, IV i V; zestaw GenoType
MYCOBACTERIA CM/AS nie różnicuje między typami I-VI). Po trzecie, żadna z dostępnych
obecnie metod nie posiada wystarczającej rozdzielczości dla wiarygodnej oceny polimorfizmu
genetycznego M. kansasii. Po czwarte, brak jest komercyjnych testów do szybkiego wykrywania
lekoopormości w szczepach M. kansasii. Dwa dostępne na rynku testy: GenoType MTBDRplus oraz
MTBDRsl (Hain Lifescience, Nehren, Niemcy) pozwalają na identyfikację oporności na izoniazyd,
ryfampicynę, fluorochinolony, aminoglikozydy/cykliczne peptydy i etambutol poprzez wykrywanie
mutacji związanych z opornością na poszczególne leki jedynie w szczepach M. tuberculosis [36, 37]).
Niezwykle ograniczona wiedza na temat korelacji między genotypem a fenotypem,
molekularnych mechanizmów i determinantów wirulencji i lekooporności prątków M. kansasii
są przyczyną braku wystandaryzowanych procedur terapeutycznych (schematów leczenia)
w przypadku chorób wywołanych przez te patogeny.
4
OPIS PROJEKTU
2.
CHARAKTERYSTYKA PROBLEMU
Obecnie znanych jest ponad 120 gatunków prątków NTM uznawanych za czynniki etiologiczne
chorób człowieka, o zmiennym obrazie klinicznym, przebiegu i rokowaniach. Mycobacterium kansasii
jest jednym z najczęściej izolowanych gatunków prątków niegruźliczych z próbek klinicznych na
całym świecie. Chociaż prewalencja zakażeń prątkami M. kansasii wykazuje duże zróżnicowanie
geograficzne, w większości regionów sytuują się one na pierwszym miejscu jako przyczyna chorób
płuc wywołanych prątkami NTM w populacji HIV-seronegatywnej i na drugim miejscu jako
przyczyna infekcji uogólnionych wśród pacjentów HIV-seropozytywnych [4, 38]. W Polsce liczba
przypadków chorób wywołanych przez prątki NTM znacząco wzrosła w ostatnich latach. Za
większość tych przypadków odpowiada właśnie M. kansasii [39]. (Częstość chorób wywołanych przez
M. kansasii w Polsce nie jest znana, ponieważ nie istnieje obowiązek rejestracji tych przypadków.
Można jedynie szacować ich liczbę pośrednio, tj. w odniesieniu do częstości występowania
przypadków gruźlicy. Tak oszacowana częstość zachorowań wskutek zakażeń M. kansasii w Polsce,
w 2010 roku, wyniosła 2,22 przypadków na 100.000 osób [H. Grubek-Jaworska, doniesienie ustne,
2012]).
Zakażenia
wywołane
przez
M.
kansasii
mają
szczególne
znaczenie
kliniczne
i epidemiologiczne, a to głównie ze względu na utrzymującą się globalnie wzrostową tendencję
w izolacji patogenu z próbek klinicznych, specyfikę leczenia zakażeń (leczenie wymaga
długotrwałego przyjmowania kilku leków równocześnie) oraz skłonność bakterii do rozwoju
lekooporności, w tym lekooporności złożonej.
Wydaje się, że głównym źródłem zakażeń M. kansasii, podobnie jak w przypadku innych prątków
NTM, jest środowisko. Transmisja zakażeń między ludźmi nie została w pełnie potwierdzona. Prątki
M. kansasii rzadko izolowane są z gleby, naturalnych zbiorników wodnych, czy tkanek zwierzęcych.
Często natomiast wykrywane są w wodzie wodociągowej, którą uważa się za główny rezerwuar
patogenu. Jednak związek między naturalnym rezerwuarem M. kansasii a chorobą u ludzi pozostaje
wciąż w dużej mierze niejasny. Co więcej, izolacja prątków M. kansasii od ludzi wcale niekoniecznie
musi oznaczać prawdziwej infekcji, lecz jedynie kolonizację lub kontaminację [1, 4, 6]. Odróżnienie
infekcji od pseudoinfekcji, a tym samym ocena klinicznego znaczenia prątków M. kansasii jest
niezwykle istotna. Wiarygodna diagnoza choroby wywołanej prątkami M. kansasii oparta jest na
wnikliwym badaniu klinicznym i laboratoryjnym, wymagającym syntetycznej oceny danych
symptomatologicznych, radiologicznych i mikrobiologicznych.
Zmienność w obrębie gatunku M. kansasii dowiedziona istnieniem 7 różnych typów genetycznych
ma szereg klinicznie i epidemiologicznie ważnych implikacji. Wyniki przeprowadzonych dotychczas
badań sugerują, że typ I M. kansasii jest najbardziej rozpowszechniony wśród szczepów klinicznych
na całym świecie i rzadko izolowany jest ze środowiska. Udział poszczególnych typów genetycznych
wśród szczepów M. kansasii zbadali po raz pierwszy Picardeau i wsp. [26]. Najczęstszym wśród
wyróżnionych typów genetycznych (I-V) był typ I, który opisywał 25 (39,7%) z 63 badanych
5
OPIS PROJEKTU
szczepów M. kansasii zarówno pochodzenia środowiskowego, jak i klinicznego. Wśród szczepów
klinicznych wykazano obecność wszystkich 5 typów genetycznych, przy czym typ I obejmował 16
(42,1%) spośród 38 takich szczepów. Częstość występowania I typu była znacznie wyższa
w badaniach przeprowadzonych przez Alcaide i wsp. [27]. Opisywał on 109 (66,9%). spośród 163
badanych szczepów klinicznych pochodzących z krajów europejskich. Wyższy odsetek typu I
M. kansasii wśród szczepów klinicznych stwierdzono w 3 kolejnych europejskich badaniach. Taillard
i wsp. wykazali, że 77,9% (60/77) szczepów ze Szwajcarii prezentowało genotyp I [30], podczas gdy
w badaniu przeprowadzonym w Hiszpanii przez Gaafar i wsp., spośród 252 szczepów M. kansasii
tylko dwa należały do II typu genetycznego (pozostałe prezentowały genotyp I) [35]. W innym
badaniu z Hiszpanii wykazano nieobecność II typu genetycznego wśród analizowanych szczepów
klinicznych. Typ I opisywał 97,8% (91/93) szczepów. (Dwa szczepy należały do typu VI) [40].
Analiza szczepów klinicznych ze Stanów Zjednoczonych wykazała, że wszystkie szczepy,
z wyjątkiem trzech, należały do I typu genetycznego. (Dwa szczepy należały do III typu
genetycznego, jeden – do typu II) [33]. Podobne wyniki otrzymali Chimara i wsp. w Brazylii. Wśród
184 szczepów klinicznych, tylko dwa prezentowały inny niż I typ genetyczny (jeden szczep należał do
typu II, jeden – do typu III) [41].
Podsumowując, wydaje się, że szczepy M. kansasii odpowiedzialne za zakażenia u ludzi należą
niemal wyłącznie do typu I i II. Co ciekawe, pacjenci HIV-seropozytywni są szczególnie podatni na
zakażenie typem II M. kansasii [26, 30, 42]. Związek między typem II M. kansasii a HIVseropozytywnością można tłumaczyć niskim potencjałem chorobotwórczym tego genotypu [30].
Epidemiologia i patogeneza chorób związanych z M. kansasii stanowi zaniedbany obszarem
badań. W Polsce literatura poświęcona epidemiologicznym i molekularnym zagadnieniom związanym
z prątkami M. kansasii jest prawie nieobecna.
Jeszcze do niedawana gatunek M. kansasii traktowano jako wysoce klonalny. Pogląd ten
podważyła identyfikacja różnych typów genetycznych (I-VII) w obrębie gatunku. Jednak rzeczywisty
stopień polimorfizmu genetycznego w obrębie M. kansasii jest nieznany. Niezdefiniowana struktura
genetyczna gatunku wynika z niewystarczającego warsztatu obecnie dostępnych metod genetycznych.
Opracowanie nowych metod, charakteryzujących się wysokim potencjałem różnicującym jest
niezbędne dla oceny zmienności genetycznej prątków M. kansasii.
Chociaż w identyfikacji gatunkowej prątków NTM, włączając M. kansasii, stosowanych jest już
kilka różnych metod, żadna nie jest optymalną, w tym sensie, że nie wykrywa jednego genu
(sekwencji), specyficznego wyłącznie dla danego gatunku prątka. Znalezienie takich wysoko
specyficznych gatunkowo markerów genetycznych jest jednym z największych wyzwań współczesnej
diagnostyki mykobakteriologicznej. Istnieje wciąż zapotrzebowanie na nowe, niezależne od hodowli
(tj. stosowane bezpośrednio w materiale klinicznym), wystandaryzowane systemy wykrywania
M. kansasii, które umożliwią wiarygodną i szybką identyfikację patogenu na poziomie zarówno
gatunku, jak i szczepu. (Obecnie taki system nie jest dostępny).
6
OPIS PROJEKTU
Istnieją tylko skąpe i kontrowersyjne doniesienia dotyczące epidemiologicznego i patogennego
znaczenia dotychczas zidentyfikowanych genotypów M. kansasii, a zwłaszcza typów III-VII, które
tylko sporadycznie wykrywane są jako sprawcy zakażeń u ludzi. Izolacja tych typów od pacjentów
z chorobami płuc sugeruje ich rolę jako czynników kolonizujących lub kontaminujących,
pochodzących ze środowiska. Dalsze badania są niezbędne dla wyjaśnienia epidemiologicznego
znaczenia dotąd opisanych i jeszcze nie poznanych genotypów M. kansasii.
Brak najmniejszych danych dotyczących mechanizmów wirulencji i oporności na leki
w szczepach M. kansasii. Nie ustalone są także związki miedzy właściwościami genotypowymi
a prezentowanym profilem fenotypowym.
Ogromne luki w wiedzy na temat M. kansasii powodują, że epidemiologiczne aspekty zakażeń
prątkami M. kansasii, w tym rezerwuar patogenu, zakaźność, drogi transmisji, rozpowszechnienie
w różnych regionach geograficznych pozostają niejasne. To z kolei utrudnia wdrożenie jakichkolwiek
procedur kontrolnych i profilaktycznych.
Podsumowując, z uwagi na fakt, że brak jest obecnie skutecznej metody typowania
genetycznego M. kansasii, pożądanym jest opracowanie nowej techniki o wysokim potencjale
różnicującym. Ponadto, istnieje potrzeba opracowania nowych, szybkich i skutecznych metod
wykrywania lekooporności oraz cech wirulencji prątków M. kansasii, umożliwiających
odróżnienie zakażenia od stanu kolonizacji (lub kontaminacji), przez co pozwalających wdrożyć
szybkie i efektywne leczenie.
3.
CEL PROJEKTU
Celem projektu jest przeprowadzenie wszechstronnej analizy struktury genetycznej szczepów
Mycobacterium kansasii. Przeprowadzona zostanie dokładna charakterystyka gatunku, obejmująca m.
in.
poziom
polimorfizmu
genetycznego
oraz
genetyczne
determinanty
lekooporności,
chorobotwórczości i wirulencji.
W ramach projektu opracowane zostaną nowe markery molekularne o kapitalnym znaczeniu
w badaniach epidemiologicznych (identyfikacja na poziomie gatunku i szczepu) oraz wykrywaniu
lekooporności i wirulencji w szczepach M. kansasii. W oparciu o zdefiniowane markery genetyczne
opracowana zostanie formuła nowych molekularnych testów diagnostycznych.
Ostatecznym celem projektu jest zaproponowanie uniwersalnego algorytmu identyfikacji
uwzględniającego wykrywanie nie tylko gatunku, ale poszczególnych szczepów i typów
genetycznych M. kansasii, a także lekowrażliwości i wirulencji patogenu, składającego się
z zestawu prostych, szybkich i wiarygodnych testów diagnostycznych optymalnie mających
zastosowanie bezpośrednio do próbek klinicznych.
7
OPIS PROJEKTU
4.
EFEKT KOŃCOWY
Projekt pozwoli szczegółowo rozpoznać strukturę genetyczną szczepów M. kansasii oraz podać
możliwie dokładną charakterystykę genetyczną gatunku. Wyniki genotypowania, w połączeniu
z wnikliwą analizą dokumentacji medycznej (danych demograficznych i klinicznych) pomogą
przybliżyć kluczowe cechy zakażeń prątkami M. kansasii. Wyniki badań projektu dostarczą
odpowiedzi na pytania, takie jak: jakie są naturalne rezerwuary patogenu?; jaki jest związek między
rezerwuarami patogenu a chorobą u ludzi?; czy wszystkie szczepy M. kansasii mają podobny
potencjał epidemiologiczny?; czy istnieją specyficzne dla genotypów różnice w fenotypie wyrażające
się odmienną wirulencją, tropizmem narządowym, czy też zdolnością transmisji? Wiedza na ten temat
może mieć ogromne znaczenie dla postępowania z chorymi na mykobakteriozy, jak również dla
opracowania i wdrożenia nowych strategii profilaktycznych.
Poznanie genetycznych podstaw lekooporności i wirulencji M kansasii umożliwi zlokalizowanie
i ocenę nowych celów molekularnych dla terapii zakażeń wywołanych prątkami NTM.
Najważniejszym osiągnięciem projektu będzie zaproponowanie i wdrożenie nowych narzędzi
metodologicznych, które usprawnią molekularną diagnostykę M. kansasii. Szczególne znaczenie
będzie miało opracowanie nowej metody typowania genetycznego, charakteryzującej się wysokim
potencjałem różnicującym, jak również molekularnych metod wykrywania lekooporności
i czynników wirulencji M. kansasii. Na bazie tych metod podana zostanie formuła pod
komercyjnie dostępne, proste, szybkie i wiarygodne testy diagnostyczne o wysokiej czułości
i swoistości. Testy te zostaną włączone do nowego algorytmu identyfikacji zakażeń prątkami
M. kansasii.
Ważnym celem projektu będzie też utworzenie pierwszej bazy wzorów genetycznych
M. kansasii, inicjującej powołania pierwszego, międzynarodowego rejestru takich wzorów.
Ogólnie, wyniki przeprowadzonych badań przyczynią się znacząco do rozwoju diagnostyki
molekularnej M. kansasii i diagnostyki mykobakteriologicznej w ogóle. Wyniki uzyskane w ramach
projektu będą bez wątpienia stanowiły ważny krok na drodze wiodącej do całkowitej eliminacji
zakażeń i chorób wywoływanych przez prątki NTM jako problemu zdrowotnego populacji ludzkiej.
Wyniki otrzymane w ramach przedłożonego projektu pozwolą przygotować szereg publikacji do
czasopism mikrobiologicznych o wysokim wskaźniku „Impact Factor” (m. in. „Journal of Clinical
Microbiology”, „Clinical Microbiology and Infection”, „European Respiratory Journal”).
Wyniki projektu zostaną również przedstawione społeczności naukowej, ustnie i/lub w formie
streszczeń na krajowych i międzynarodowych sympozjach i konferencjach (m. in. Polskiego
Towarzystwa Chorób Płuc, European Respiratory Society, European Congress of Clinical
Microbiology and Infectious Diseases (ECCMID), czy the Annual Congress of the European Society
of Mycobacteriology (ESM)).
8
OPIS PROJEKTU
5.
HARMONOGRAM PRAC
PRZYGOTOWANIE
MATERIAŁU
BADAWCZEGO.
REWITALIZACJA
SZCZEPÓW.
WYWIAD
EPIDEMIOLOGICZNY
Na wstępie zostanie przygotowany materiał badawczy. Etap ten będzie obejmował staranne
przeszukanie całej kolekcji szczepów należącej do Katedry Chorób Wewnętrznych, Pneumonologii
i Alergologii Warszawskiego Uniwersytetu Medycznego, w celu wyboru tylko takich szczepów, które
wcześniej zostały zidentyfikowane jako M. kansasii przy użyciu standardowych procedur
biochemicznych, testu AccuProbe lub wysokociśnieniowej chromatografii cieczowej (HPLC).
Szczepy w liczbie ok. 150-200 będą ożywiane przez posiew na pożywce Löwenstein’a-Jensen’a.
Do badania zostaną również włączone szczepy M. kansasii (w tym szczepy referencyjne)
reprezentujące siedem typów genetycznych (I-VII), zdeponowane w ogólnodostępnych kolekcjach
międzynarodowych. Szczepy będą też pozyskiwane, na zasadach komercyjnych, od różnych ośrodków
badawczych na całym świecie.
Równolegle, przeprowadzona zostanie analiza retrospektywna danych o chorych, od których
izolowano szczepy M. kansasii (zebrany zostanie szczegółowy wywiad demograficzny i kliniczny).
Przeprowadzona zostanie wnikliwa analiza dokumentacji medycznej poszczególnych przypadków.
Istotne znaczenie będą miały dane dotyczące wieku, płci, miejsca zamieszkania, zawodu, nałogów,
dodatkowych
schorzeń,
dane
radiologiczne
i
histopatologiczne,
dane
dotyczące
badań
mikroskopowych (na obecność prątków kwasoopornych), rodzaju materiału klinicznego, liczby
hodowli pozytywnych na obecność prątków, a także czasu hospitalizacji chorych, schematów i czasu
leczenia oraz wyników leczenia.
W celu wstępnego rozróżnienia między infekcją a kolonizacją, wszystkie przypadki zakażeń
M. kansasii zostaną zweryfikowane ściśle w oparciu o kryteria zalecane przez American Thoracic
Society (ATS).
IDENTYFIKACJA GATUNKOWA. WERYFIKACJA PRZYNALEŻNOŚCI WYBRANYCH SZCZEPÓW DO
GATUNKU
Celem tej części projektu będzie potwierdzenie przynależności gatunkowej wszystkich badanych
szczepów przy użyciu najnowszych, dostępnych metod molekularnych. Wcześniej wszystkie szczepy
będą miały uzupełnioną identyfikację przy pomocy konwencjonalnych testów biochemicznych [43],
systemu Accuprobe (Gen-Probe, San Diego, USA) [10], oraz techniki HPLC [5]. Przeprowadzone
zostanie, ściśle wg zaleceń producenta, typowanie testem GenoType Mycobacterium CM/AS (HAIN
Lifescience, Niemcy). Wykonane zostaną analizy PCR-RFLP dla genów hsp65, rpoB i tuf, wg
wcześniej publikowanych protokołów [18, 22, 23]. Pozwoli to na ocenę zdolności rozdzielczej metod
molekularnych w identyfikacji M. kansasii. Ostatecznym celem tej części projektu będzie
przeprowadzenie analizy porównawczej 4 różnych metod stosowanych w identyfikacji M. kansasii, tj.:
metody konwencjonalnej, opartej o kryteria biochemiczne (fenotypowe) (i), techniki HPLC (ii),
9
OPIS PROJEKTU
komercyjnego testu GenoType Mycobacterium, wykorzystującego zasadę hybrydyzacji DNA (iii) oraz
analizy PCR-RFLP trzech różnych genetycznych loci (hsp65, rpoB i tuf) (iv). Podobne badanie
porównawcze nigdy wcześniej nie było przeprowadzone.
Dla celów molekularnej identyfikacji gatunkowej, a także do dalszych etapów genotypowania
wykonana zostanie izolacja genomowego DNA prątków [44].
TYPOWANIE GENETYCZNE M. KANSASII. STRUKTURA GENETYCZA M. KANSASII
Ta część projektu będzie miała na celu ocenę zróżnicowania genetycznego szczepów M. kansasii.
W pierwszym etapie, wyniki analizy hsp65 PCR-RFLP wykorzystane zostaną do identyfikacji typów
genetycznych M. kansasii. Umożliwi to określenie występowania i rozpowszechnienia 7 dotychczas
opisanych typów genetycznych M. kansasii (a być może ujawni istnienie nowych genotypów). Jako,
że wyniki pochodzące z analizy hsp65 PCR-RFLP nie zawsze korespondują z wynikami analiz
sekwencyjnych regionu ITS, zastosowana zostanie także ta metoda badawcza (analiza sekwencyjna
regionu ITS). Dodatkowo, wyniki otrzymane przy pomocy obu metod zostaną porównane z wynikami
uzyskanymi z analizy rpoB PCR-RFLP. Wreszcie, aby lepiej poznać ewolucyjne powiązania między
typami genetycznymi M. kansasii przeprowadzona zostanie analiza sekwencyjna genów rpoB i hsp65.
Znaczenie analizy sekwencyjnej genu rpoB w typowaniu genetycznym M. kansasii nie zostało w pełni
wyświetlone [45].
Drugim krokiem będzie ocena przydatności dwóch metod stosowanych dla oceny zróżnicowania
wewnątrzgatunkowego M. kansasii tj. analizy PFGE i AFLP. Obecnie, metody te jako jedyne
pozwalają różnicować w obrębie poszczególnych typów genetycznych [35, 33].
Wyniki uzyskane w dwóch wyżej wymienionych etapach zostaną skonfrontowane z wynikami
innych autorów, co pozwoli ocenić, w perspektywie globalnej, heterogenność gatunku M. kansasii,
a także zdefiniować potencjalne ewolucyjne relacje między poszczególnymi genotypami.
Ważnym etapem tej części projektu będzie poszukiwanie nowych molekularnych narzędzi,
zdolnych z większą rozdzielczością wykrywać polimorfizm genetyczny w obrębie M. kansasii. W tym
celu wykonanych zostanie szereg analiz bioinformatycznych, w tym szczegółowa analiza in silico
całego genomu M. kansasii (na bazie zdeponowanej sekwencji szczepu referencyjnego M. kansasii
ATCC 12478). Poszukiwane będą charakterystyczne sekwencje genomowe, takie jak np. sekwencje
repetytywne, mogące mieć zastosowanie jako markery genetyczne w identyfikacji (sub-)gatunkowej
M. kansasii. Jednym z poszukiwanych rodzajów powtórzonego DNA będą sekwencje typu VNTR
(ang. variable number of tandem repeats), a w szczególności sekwencje typu MIRU (ang.
mycobacterial interspersed repetitive units), które z powodzeniem wykorzystane zostały
w genotypowaniu kilkunastu gatunków prątków NTM [46-48]. Typowanie oparte o sekwencje typu
VNTR jest odmianą typowania MLVA (ang. multiple-locus variable-number tandem repeat analysis)
i polega na amplifikacji techniką PCR specyficznego locus zawierającego tandemowo ułożone
sekwencje powtórzone, a następnie rozdziale elektroforetycznym produktów amplifikacji i określeniu
10
OPIS PROJEKTU
ich wielkości. Wielkość produktu zależy od liczby powtórzeń jednostki rdzeniowej w danym locus.
Wzory genetyczne generowane w typowaniu MLVA stanowią kody numeryczne, odzwierciedlające
liczbę powtórzeń motywu sekwencyjnego w każdym z analizowanych loci. Jest to szczególnie
wygodne dla porównywania wyników między ośrodkami, a także w analizach filogenetycznych.
Identyfikacja genetycznych loci mogących mieć zastosowanie w typowaniu VNTR będzie
wymagała
szczegółowej
analizy bioinformatycznej
na
bazie
sekwencji
genomu
szczepu
referencyjnego ATCC 12478. Poszukiwane będą przede wszystkim sekwencje homologiczne wobec
sekwencji typu MIRU występujących w szczepach M. tuberculosis, przy użyciu programów BLAST
(http://blast.ncbi.nlm.nih.gov/Blast.cgi) oraz Finder (http://tandem.bu.edu/trf/trf.html). W przypadku
wykrycia loci VNTR, zaprojektowane zostaną specyficzne startery ukierunkowane na sekwencje
flankujące region VNTR umożliwiając jego amplifikację i dalszą analizę.
Innym podejściem wykorzystywanym do genotypowania szczepów M. kansasii będzie użycie
techniki MST (ang. multispacer sequence typing). Opiera się ona na analizie sekwencji regionów
międzygenowych bakterii. Technikę MST uprzednio stosowano w genotypowania tylko kilku
gatunków prątków, w tym M. tuberculosis [49] i prątków kompleksu M. avium [50].
W celu wytypowania regionów przydatnych do genotypowania MST, sekwencja genomu szczepu
referencyjnego ATCC 12478 ponownie będzie poddana analizie bioinformatycznej przy użyciu
oprogramowania EMBOSS (http://www.emboss.sourceforge.net). Po wytypowaniu sekwencji
międzygenowych zaprojektowane zostaną odpowiednie pary starterów. Produkty PCR będą następnie
sekwencjonowane
i
analizowane
przy
użyciu
oprogramowania
CLUSTAL
W
(http://bioinfo.hku.hk/services/analyseq/cgi-bin/clustalw_in.pl).
W celu określenia wewnątrzgatunkowej zmienności genetycznej M. kansasii wykonane zostaną
analizy PCR-RFLP oraz sekwencyjne innych niż dotąd badane genów metabolizmu podstawowego.
Etap ten zakłada przeszukanie genomu M. kansasii w celu znalezienia homologów genów
M. tuberculosis takich, jak np. katG – kodujący enzym katalazę-peroksydazę, warunkujący wrażliwość
prątków na izoniazyd. (M. kansasii jest naturalnie oporny na izoniazyd. Sekwencjonowanie homologu
katG w M. kansasii, może przybliżyć molekularne podstawy oporności na ten lek) [43]).
Wyniki otrzymane przy użyciu różnych metod typowania genetycznego zostaną porównane
i poddane analizie skupień, tak aby dokładnie określić pokrewieństwo klonalne między szczepami
M. kansasii.
Potencjał różnicujący każdej metody genotypowania będzie obliczany przy użyciu tzw. Hunter
Gaston Index [51]. W celu określenia pokrewieństwa genetycznego szczepów M. kansasii, na
podstawie otrzymanych wzorów genetycznych, wykonana zostanie analiza skupień (a także
konstrukcja drzew filogenetycznych) przy użyciu oprogramowania GelCompar (ver. 4.0; Applied
Maths., Kortrijk, Belgia).
Ogólnie, prezentowana część projektu pozwoli uzyskać szczegółowy wgląd w strukturę
genetyczną szczepów M. kansasii.
11
OPIS PROJEKTU
BADANIE WRAŻLIWOŚCI NA LEKI I IDENTYFIKACJA GENÓW WIRULENCJI
Badanie wrażliwości na leki (DST ang. drug susceptibility testing) jest integralnym elementem
postępowania diagnostycznego przy zakażeniach prątkami M. kansasii. Obecnie nieznany jest związek
między określonym typem genetycznym M. kansasii a opornością na leki. Ważne jest także określenie
czynników ryzyka rozwoju lekooporności w szczepach M. kansasii. Celem tej części projektu będzie
próba wyjaśnienia tych zagadnień. Ustalone zostaną profile lekowrażliwości dla wszystkich badanych
szczepów M. kansasii. Będą one analizowane w kontekście wzorów genetycznych oznaczonych dla
poszczególnych szczepów. Jak dotąd nie przeprowadzono jeszcze podobnego badania.
Profile lekowrażliwości oznaczone zostaną metodą mikrorozcieńczeń, zgodnie ze standardowym
protokołem Clinical and Laboratory Standards Institute (CLSI) [52]. Testowane będą leki tzw.
pierwszego rzutu: streptomycyna (SM), izoniazyd (INH), etambutol (EMB), ryfampicyna (RMP),
a także tzw. drugiego rzutu: ryfabutyna (RFB), amikacyna (AMK), ciprofloksacyna (CFX),
klofazymina (CFZ), klarytromycyna (CLR), etionamid (ETH), cykloseryna (CS). Stężenia graniczne
stosowane do klasyfikowania szczepów jako wrażliwe lub oporne na dany lek będą zgodne
z wytycznymi CLSI [52]. Do określenia minimalnego stężenia hamującego (MIC, ang. minimal
inhibitory concentration) leków stosowane będą, zgodnie z instrukcją producenta, paski E-test (AB
Biodisk, Solna, Szwecja).
Czynniki ryzyka rozwoju lekooporności zostaną zdefiniowane poprzez porównanie przypadków
zakażeń prątkami M. kansasii opornymi i wrażliwymi na poszczególne leki.
Zbadane zostaną także determinanty wirulencji M. kansasii. Motywem do podjęcia takich badań
jest fakt, że w literaturze światowej brak jakichkolwiek szerszych danych na temat patogenezy chorób
wywołanych prątkami M. kansasii. Ponadto, jako, że wirulencja patogenu znacząco wpływa na jego
epidemiologię, poznanie determinantów zjadliwości prątków M. kansasii pozwoli lepiej zrozumieć
epidemiologię wywoływanych przez nie zakażeń.
W celu identyfikacji genów wirulencji M. kansasii, na podstawie analizy dostępnej literatury oraz
dostępnych baz danych utworzony zostanie wykaz genów kodujących potencjalne czynniki wirulencji.
Wybrane zostaną geny, kodujące zarówno dobrze zdefiniowane, jako i hipotetyczne czynniki
wirulencji dla M. tuberculosis, jak np. katG, hspX, plcABC, i fbpA [53], czy też geny MAV dla
prątków M. avium [54]). Sekwencje tych genów będą poszukiwane w genomie M. kansasii (ATCCC
12478). Obecność danego genu zostanie potwierdzona jeśli jego białkowy produkt wykaże ponad 50%
homologii z typowanym białkiem i jeśli długość białkowego produktu przekroczy 80% długości białka
typowanego. Zidentyfikowane tak geny będą następnie amplifikowane i analizowane sekwencyjnie.
Strategia PCR-MLST (ang. multilocus sequence typing) zostanie wykorzystana do różnicowania
wewnątrzgatunkowego prątków. Ponadto, z uwagi na fakt, że obecność genu nie jest tożsama
z ekspresją białka, oznaczony zostanie poziom transkrypcji genu przy użyciu techniki Real-Time
(RT)-PCR. Wyniki uzyskane na tym etapie zostaną porównane z wynikami z wcześniejszych etapów.
12
OPIS PROJEKTU
INTEGRACJA DANYCH. ZAPORPONOWANIE METODOLOGICZNEGO ALGORYTMU GENOTYPOWANIA
M. KANSASII
Celem ostatniej części projektu będzie integracja danych molekularnych (pochodzących z badań
nad zróżnicowaniem genetycznym szczepów M. kansasii) oraz społeczno-demograficznych
i klinicznych (pochodzących z przeglądu dokumentacji medycznej chorych) w całościowej analizie
epidemiologicznej zakażeń M. kansasii. Szczególnie ważne będzie porównanie wyników uzyskanych
dla szczepów klinicznych i środowiskowych.
Podjęta zostanie próba odpowiedzi na pytania dotyczące rezerwuaru M. kansasii, związku między
nim a chorobą u ludzi, związku między cechami genotypowymi a określonym profilem fenotypowym,
wyrażającym się odmienną wirulencją, tropizmem narządowym, czy też zdolnością transmisji, a także
możliwości transmisji zakażeń między ludźmi oraz różnic w potencjale epidemiologicznym między
różnymi genotypami (szczepami) M. kansasii.
Wyniki wszystkich badań wykonanych w trakcie realizacji projektu będą poddawane licznym
analizom porównawczym i statystycznym. Wyniki będą też zestawiane z danymi obecnymi
w literaturze przedmiotu. Wnioski wyprowadzone z badań ujętych w projekcie zostaną
skonfrontowane z istniejącą wiedzą i formułowanymi dotąd hipotezami na temat epidemiologii
M. kansasii.
Na podstawie kompleksowej analizy zebranych danych, zaproponowany zostanie nowy
algorytm identyfikacji, typowania genetycznego oraz wykrywania lekooporności i wirulencji
w szczepach M. kansasii.
13
OPIS PROJEKTU
SCHEMAT ILUSTRUJĄCY KLUCZOWE ETAPY PROJEKTU
14
OPIS PROJEKTU
PLANOWANE POSTĘPY W REALIZACJI PROJEKTU
I.
KAMIENIE MILOWE
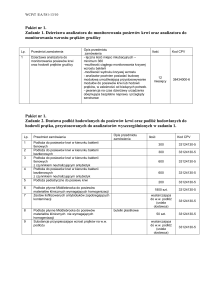
Kamień milowy
Planowany
czas
rozpoczęcia
Planowany
czas
zakończenia
Planowany czas
punktu
krytycznego
Planowany czas
punktu
ostatecznego
1.
Zebranie materiału badawczego
1. miesiąc
6 miesiąc
2. miesiąc
6. miesiąc
2.
Identyfikacja gatunkowa
7. miesiąc
12 miesiąc
9. miesiąc
12. miesiąc
3.
Genotypowanie M. kansasii;
S
Struktura genetyczna M. kansasii
13. miesiąc
30 miesiąc
23. miesiąc
28. miesiąc
4.
Lekowrażliwość i identyfikacja
genów wirulencji
21. miesiąc
32 miesiąc
28. miesiąc
32. miesiąc
33. miesiąc
36 miesiąc
34. miesiąc
36. miesiąc
5.
II.
Integracja danych. Propozycja
nowego algorytmu identyfikacji
M. kansasii
WSKAŹNIKI PRODUKTU
Nazwa wskaźnika
Jednostka miary
wskaźnika
Planowana wartość bazowa
mierzona przed rozpoczęciem
realizacji projektu
Planowana wartość
docelowa wskaźnika
Nowe testy diagnostyczne:
typowanie genetyczne;
2. oznaczanie lekooporności;
3. oznaczanie cech wirulencji
1
0
1
Nowy algorytm identyfikacji zakażeń
M. kansasii
1
0
1
Określenie molekularnych celów dla
nowych strategii terapeutycznych
1
0
1
1.
III.
WSKAŹNIKI REZULTATU
Nazwa wskaźnika
Jednostka miary
wskaźnika
Planowana wartość bazowa
mierzona przed rozpoczęciem
realizacji projektu
Planowana wartość
docelowa wskaźnika
Identyfikacja oraz wybór markerów
genetycznych mających zastosowanie
w różnicowaniu wewnątrzgatunkowym,
wykrywaniu lekooporności i wirulencji
1
0
1
Określenie profilów lekooporności dla
szczepów M. kansasii
1
0
1
Komercjalizacja opracowanych narzędzi
metodologicznych
1
0
1
Upowszechnienie wyników badań przez
udział w konferencjach (referaty/plakaty)
i publikacje naukowe
1
0
1
15
OPIS PROJEKTU
6.
CEL EKONOMICZNY I ZNACZENIE PRAKTYCZNE PROJEKTU
Celem ekonomicznym projektu jest
opracowanie
nowych testów
diagnostycznych,
pozwalających na szybką i wiarygodną identyfikację zakażeń M. kansasii. Szczególną wartość
będą miały opracowania – w formie komercyjnie dostępnych testów – metod typowania genetycznego
szczepów M. kansasii, charakteryzujących się wysokim potencjałem różnicującym, a także technik
wykrywania lekooporności i cech (poziomu) wirulencji w szczepach M. kansasii. (Obecnie metody
takie nie są komercyjnie dostępne). Na podstawie opracowanych testów diagnostycznych,
zaproponowany zostanie nowy, uniwersalny algorytm molekularnej identyfikacji zakażeń
M. kansasii, który w istotny sposób ułatwi i przyspieszy postawienie właściwej diagnozy i dokonanie
wyboru najodpowiedniejszej terapii, prowadząc do wyleczenia w jak najszybszym czasie
z wykluczeniem lub minimalizując działania niepożądane.
Wyniki badań przeprowadzonych w ramach projektu będą miały istotne znaczenie dla
praktyki klinicznej, tj. postępowania z chorymi na mykobakteriozy wywołane zakażeniem
M. kansasii,
czy
też
opracowania
i
wdrożenia
nowych
strategii
terapeutycznych
i profilaktycznych. Metody te zostaną włączone jako integralny element światowego programu walki
na rzecz eliminacji tego patogenu.
Wyniki uzyskane w trakcie realizacji projektu w istotny sposób poszerzą wiedzę na temat
epidemiologii zakażeń wywoływanych przez prątki M. kansasii. W świetle wyjątkowo ubogiej
literatury poświęconej temu problemowi, otrzymane wyniki pozwolą po raz pierwszy szczegółowo
opisać cechy epidemiologiczne zakażeń M. kansasii, włączając źródła i dynamikę transmisji zakażeń,
stopień osobniczej podatności na zakażenia, czy w końcu poziom wirulencji i inwazyjności szczepów
M. kansasii. Wyniki molekularnych badań epidemiologicznych, wykonanych w ramach projektu, będą
miały
bezpośrednie
przełożenie
na
praktykę
kliniczną
(leczenie
chorych)
oraz
nadzór
epidemiologiczny (profilaktyka medyczna). Z kolei opracowanie nowej metody wykrywania zakażeń
M. kansasii przyczyni się do rozwoju diagnostyki bakteriologicznej. W końcu wyniki otrzymane
w toku badań przewidzianych w projekcie będą miały znaczenie dla lepszego poznania filogenetyki
i ewolucji NTM.
Prezentowany projekt posiada wysoki potencjał aplikacyjny i komercjalizacyjny. Przy transferze
wyników prac do gospodarki planujemy skorzystać z oferty Uniwersyteckiego Ośrodka Transferu
Technologii (UOTT), który wspiera przedsięwzięcia mające na celu praktyczne wykorzystanie
wyników pracy naukowej. W ramach działań przygotowujących wyniki badań do wdrożenia
planowane jest zgłoszenie do ochrony patentowej opracowanych metod lub objęcie ich licencją „know
how” oraz ustalenie prawnych zasad przejmowania projektu przez producenta.
Działania w zakresie szczegółowego badania rynku dla przyszłego produktu, sporządzenia
niezbędnej do wdrożenia dokumentacji technicznej, certyfikacji, określenia i zlecenia badań
jakościowych, opracowania właściwej technologii wytwarzania i dokumentacji produkcyjnej będą
16
OPIS PROJEKTU
leżały po stronie producenta. Potencjalnymi odbiorcami wyników projektu są firmy farmaceutyczne
i biotechnologiczne zajmujące się produkcją komercyjnych testów do identyfikacji drobnoustrojów
patogennych czy też oferujące usługi w zakresie molekularnej diagnostyki mikrobiologicznej,
a w konsekwencji szpitale i inne jednostki medyczne i ochrony zdrowia, a także ośrodki naukowe.
PIŚMIENNICTWO
1.
Falkinham, J. O. 2002. Nontuberculous mycobacteria in the environment. Clin. Chest Med. 23:529-551.
2.
Heifets, L. 2004. Mycobacterial infections caused by nontuberculous mycobacteria. Semin. Respir. Crit. Care Med.
25:283-295.
3.
Griffith, D. E., T. Aksamit, B. A. Brown-Elliott, A. Catanzaro, C. Daley, F. Gordin, S. M. Holland, R.
Horsburgh, G. Huitt, M. F. Iademarco, M. Iseman, K. Olivier, S. Ruoss, C. F. von Reyn, R. J. Wallace Jr. K.
Winthrop; ATS Mycobacterial Diseases Subcommittee; American Thoracic Society; Infectious Disease
Society of America. 2007. An official ATS/IDSA statement: diagnosis, treatment, and prevention of
nontuberculous mycobacterial diseases. Am. J. Respir. Crit. Care Med. 175:367-416.
4.
Falkinham, J. O. 1996. Epidemiology of infection by nontuberculous mycobacteria. Clin. Microbiol. Rev. 9:177215.
5.
Butler, W. R., and J. O. Kilburn. 1988. Identification of major slowly growing pathogenic mycobacteria and
Mycobacterium gordonae by high-performance liquid chromatography of their mycolic acids. J. Clin. Microbiol.
30:2402-2407.
6.
Huang, Z. H., B. C. Ross, and B. Dwyer. 1991. Identification of Mycobacterium kansasii by DNA hybridization.
J. Clin. Microbiol. 29:2125-2129.
7.
Ross, B. C., K. Jackson, M. Yang, A. Sievers, and B. Dwyer. 1992. Identification of a genetically distinct
subspecies of Mycobacterium kansasii. J. Clin. Microbiol. 30:2930-2933.
8.
Yang, M., B. C. Ross, and B. Dwyer. 1993. Identification of an insertion sequence-like element in a subspecies of
Mycobacterium kansasii. J. Clin. Microbiol. 31:2074-2079.
9.
Yang, M., B. C., Ross, and B. Dwyer. 1993. Isolation of a DNA probe for identification of Mycobacterium
kansasii, including the genetic subgroup. J. Clin. Microbiol. 31:2769-2772.
10. Tortoli, E., M. T. Simonetti, and F. Lavinia. 1996. Evaluation of reformulated chemiluminescent DNA probe
(AccuProbe) for culture identification of Mycobacterium kansasii. J. Clin. Microbiol. 34:2838-2840.
11. Tortoli, E., A. Nanetti, C. Piersimoni, P. Cichero, C. Farina, G. Mucignat, C. Scarparo, L. Bartolini, R.
Valentini, D. Nista, G. Gesu, C. P. Tosi, M. Crovatto, and G. Brusarosco. 2001. Performance assessment of
new multiplex probe assay for identification of mycobacteria. J. Clin. Microbiol. 39:1079-1084.
12. Quezel-Guerraz, N. M., Arriaza, M. M., Avila, J. A., Sánchez-Yebra Romera, W. E., Martínez-Lirola, M. J.;
Indal-TB Group. Evaluation of the Speed-oligo® Mycobacteria assay for identification of Mycobacterium spp.
from fresh liquid and solid cultures of human clinical samples. Diagn. Microbiol. Infect. Dis., 2010, 68:123-131.
13. Hofmann-Thiel, S., Turaev, L., Alnour, T., Drath, L., Müllerova, M., Hoffmann, H. Multi-centre evaluation of
the speed-oligo Mycobacteria assay for differentiation of Mycobacterium spp. in clinical isolates. BMC Infect. Dis.
2011, 11:353.
14. Lee, A. S., P. Jelfs, V. Sintchenko, and G. L. Gilbert. 2009. Identification of non-tuberculous mycobacteria:
utility of the GenoType Mycobacterium CM/AS assay compared with HPLC and 16S rRNA gene sequencing. J.
Med. Microbiol. 58:900-904.
15. Richter, E., S. Rüsch-Gerdes, and D. Hillemann. 2006. Evaluation of the GenoType Mycobacterium Assay for
Identification of mycobacterial species from cultures. J. Clin. Microbiol. 44:1769-1775.
17
OPIS PROJEKTU
16. Russo, C., E. Tortoli, and D. Menichella. 2006. Evaluation of the new GenoType Mycobacterium assad for
identification of mycobacterial species. J. Clin. Microbiol. 44:334-339.
17. Plikaytis, B. B., B. D. Plikaytis, A. Mitchell, W. Yakrus, R. Butler, C. L. Woodley, V. A. Silcox, and T. M.
Shinnick. 1992. Differentiation of slowly growing Mycobacterium species, including Mycobacterium tuberculosis,
by gene amplification and restriction fragment length polymorphism analysis. J. Clin. Microbiol. 30:1815-1822.
18. Telenti, A., F. Marchesi, M. Balz, F. Bally, E. C. Böttger, and T. Bodmer. 1993. Rapid identification of
mycobacteria to the species level by polymerase chain reaction and restriction enzyme analysis. J. Clin. Microbiol.
31:175-178.
19. Devallois, A., K. S. Goh, and N. Rastogi. 1997. Rapid identification of mycobacteria to species level by PCRrestriction fragment length polymorphism analysis of the hsp65 gene and proposition of an algorithm to
differentiate 34 mycobacterial species. J. Clin. Microbiol. 35:2969-2973.
20. Brunello, F., M. Ligozzi, E. Cristelli, S. Bonora, E. Tortoli, and R. Fontana. 2001. Identification of 54
mycobacterial species by PCR-restriction fragment length polymorphism analysis of the hsp65 gene. J. Clin.
Microbiol. 39:2799-2806.
21. Takewaki, S., K. Okuzumi, I. Manabe, M. Tanimura, K. Miyamura, K. Nakahara, Y. Mazaki, A. Ohkubo,
and R. Nagai. 1994. Nucleotide sequence comparison of the mycobacterial dnaJ gene and PCR-restriction
fragment length polymorphism analysis for identification of mycobacterial species. Int. J. Syst. Bacteriol. 44:159166.
22. Kim, B.-J., K.-H. Lee, B.-N. Park, S.-J. Kim, G.-H. Bal, S.-J. Kim, and Y.-H. Kook. 2001. Differentiation of
mycobacterial species by PCR-restriction analysis of DNA (343 base pairs) of the RNA polymerase gene (rpoB). J.
Clin. Microbiol. 39:2102-2109.
23. Shin, J.-H., E.-J. Cho, J.-Y. Lee, J.-Y. Yu, and Y.-H. Kang. 2009. Novel diagnostic algorithm using tuf gene
amplification and restriction fragment length polymorphism is promising tool for identification of nontuberculous
mycobacteria. J. Microbiol. Biotechnol. 19:323-330.
24. Abed, Y., C. Bollet, and P. de Micco. 1995. Demonstration of Mycobacterium kansasii species heterogeneity by
the amplification of the 16S-23S spacer region. J. Med. Microbiol. 42:112-114.
25. Hermans, P. W., D. van Soolingen, and J. D.A. van Embden. 1992. Characterization of a major polymorphic
tandem repeat in Mycobacterium tuberculosis and its potential use in the epidemiology of Mycobacterium kansasii
and Mycobacterium gordonae. J. Bacteriol. 174:4157-4165.
26. Picardeau, M., G. Prod’hom, L. Raskine, M. P. LePennec, and V. Vincent. 1997. Genotypic characterization of
five subspecies of Mycobacterium kansasii. J. Clin. Microbiol. 35:25-32.
27. Alcaide, F., I. Richter, C. Bernasconi, B. Springer, C. Hagenau, R. Schulze-Röbbecke, E. Tortoli, R. Martín,
E. C. Böttger, and A. Telenti. 1997. Heterogeneity and clonality among isolates of Mycobacterium kansasii:
implications for epidemiological and pathological studies. J. Clin. Microbiol. 35:1959-1964.
28. Sander, P., F. Alcaide, I. Richter, K. Frischkorn, E. Tortoli, B. Springer, A. Telenti, and E. C. Böttger. 1998.
Inteins in mycobacterial GyrA are a taxonomic character. Microbiology 144:589-591.
29. Richter, E., S. Niemann, S. Rüsch-Gerdes, and S. Hoffner. 1999. Identification of Mycobacterium kansasii by
using a DNA probe (AccuProbe) and molecular techniques. J. Clin. Microbiol. 37:964-970.
30. Taillard, C., G. Greub, R. Weber, G. E. Pfyffer, T. Bodmer, S. Zimmerli, R. Frei, S. Bassetti, P. Rohner, J.
C. Piffaretti, E. Bernasconi, J. Bille, A. Telenti, and G. Prod’hom. 2003. Clinical implications of
Mycobacterium kansasii species heterogeneity: Swiss National Survey. J. Clin. Microbiol. 41:1240-1244.
31. Iwamoto, T., and H. Saito. 2005. comparative study of two typing methods hsp65 PRA and ITS sequencing
revealed a possible evolutionary link between Mycobacterium kansasii type I and II isolates. FEMS Microbiol.
Lett. 254:129-133.
18
OPIS PROJEKTU
32. Iinuma, Y., S. Ichiyama, Y. Hasegawa, K. Shimokata, s. Kawahara, and T. Matsushima. 1997. Largerestriction-fragment analysis of Mycobacterium kansasii genomic DNA and its application in molecular typing. J.
Clin. Microbiol. 35:596-599.
33. Zhang, Y., L. B. Mann, R. W. Wilson, B. A. Brwon-Elliott, V. Vincent, Y. Iinuma, and R. J. Wallace Jr.
2004. Molecular analysis of Mycobacterium kansasii isolates from the United States. J. Clin. Microbiol. 42:119125.
34. Wu, T.-S., H.-S. Leu, C.-H. Chiu, M.-H. Lee, P.-C. Chiang, T.-L. Wu, J.-H. Chia, L.-H. Su, A.-J. Kuo, and
H.-C. Lai. 2009. Clinical manifestations, antibiotic susceptibility and molecular analysis of Mycobacterium
kansasii isolates from a university hospital in Taiwan. J. Antimicrob. Chemother. 64:511-514.
35. Gaafar, A., M. J. Unzaga, R. Cisterna, F. E. Clavo, E. Urra, R. Ayarza, and G. Martín. 2003. Evaluation of a
modified single-enzyme amplified-fragment length polymorphism technique for fingerprinting and differentiating
of Mycobacterium kansasii type I isolates. J. Clin. Microbiol. 41:3846-3850.
36. Hillemann, D., Weizenegger, M., Kubica, T., Richter, E., Niemann, S. 2005. Use of the genotype MTBDR
assay for rapid detection of rifampin and isoniazid resistance in Mycobacterium tuberculosis complex isolates. J.
Clin. Microbiol. 43:3699-703.
37. Brossier, F., Veziris, N., Aubry, A., Jarlier, V., Sougakoff, W. 2010. Detection by GenoType MTBDRsl test of
complex mechanisms of resistance to second-line drugs and ethambutol in multidrug-resistant Mycobacterium
tuberculosis complex isolates. J. Clin. Microbiol. 48:1683-1689.
38. Bloch, K. C., L. Zwerling, M. J. Pletcher, J. A. Hahn, J. L. Gerberding, S. M. Ostroff, D. J. Vugia, and A. L.
Reingold. 1998. Incidence and clinical implications of isolation of Mycobacterium kansasii: results of a 5-year,
population-based study. Ann. Intern. Med. 129:698-704.
39. Walkiewicz, R., A. Safianowska, H. Grubek-Jaworska, M. Nowacka-Mazurek, R. Renke, T. Przybyłowski,
and R. Chazan. 2004. Frequency of mycobacterioses among the patients with positive cultures of nontuberculous
mycobacteria (NTM) – 5 year study. Eur. J. Resp. J. Suppl.48:190.
40. Santin, M., F. Alcaide, M. A. Benitez, A. Salazar, C. Ardanuy, D. Podzamczer, G. Rufi, J. Dorca, R. Martin,
and F. Gudiol. 2004. Incidence and molecular typing of Mycobacterium kansasii in a defined geographical area in
Catalonia, Spain. Epidemiol. Infect. 132:425-432.
41. Chimara, E., C. M. Giampaglia, M. Conceição Martins, M. A. De Silva Telles, S. Y. Ueki, and L. Ferrazoli.
2004. Molecular characterization of Mycobacterium kansasii isolates in the state of São Paulo between 1995-1998.
Mem. Inst. Oswaldo Cruz 99:739-743.
42. Tortoli, E., M. T. Simonetti, C. Lacchini, V. Penati, and P. Urbano. 1994. Tentative evidence of AIDSassociated biotype of Mycobacterium kansasii. J. Clin. Microbiol. 32:1779-1782.
43. Kent, P. T., and G. P. Kubica. 1985. Public health mycobacteriology. A guide for the level III laboratory. Centers
for Disease Control, Atlanta.
44. van Embden, J. D. A., M. D. Cave, J. T. Crawford, J. W. Dale, K. D. Eisenach, B. Gicquel, et al. 1993. Strain
identification of Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized
methodology. J Clin. Microbiol. 31:406-409.
45. Adékambi, T., P. Colson, and M. Drancourt. 2003. rpoB-based identification of nonpigmented and latepigmenting rapidly growing mycobacteria. J. Clin. Microbiol. 41:5699-5708.
46. Dauchy, F.-A., S. Dégrange, A. Charron, M. Dupon, Y. Xin, C. Bébéar, and J. Maugein. 2010. Variablenumber tandem-repeat markers for typing Mycobacterium intracellulare strains isolated in humans. BMC
Microbiol. 10:93.
47. Overduin, P., L. Schouls, P. Roholl, A. van der Zanden, N. Mahmmod, A. Herrewegh, and D. van Soolingen.
2004. Use of multilocus variable-number tandem-repeat analysis for typing Mycobacterium avium subsp.
paratuberculosis. J. Clin. Microbiol. 42:5022-5028.
19
OPIS PROJEKTU
48. Hilty, M., M. Käser, J. Zinsstag, T. Stinear, and G. Pluschke. 2007. Analysis of the Mycobacterium ulcerans
genome sequence reveals new loci for variable number tandem repeats (VNTR) typing. Microbiology 153:14831487.
49. Djelouadji, Z., C. Arnold, S. Gharbia, D. Raoult, and M. Drancourt. 2008. Multispacer sequence typing for
Mycobacterium tuberculosis genotyping. PLoS One. 3:2433.
50. Cayrou, C., C. Turenne, M. A. Behr, and M. Drancourt. 2010. Genotyping of Mycobacterium avium complex
organisms using multispacer sequence typing. Microbiology. 156:687-694.
51. Hunter, P. R., and M. A. Gaston. 1988. Numerical index of the discriminatory ability of typing systems: an
application of Simpson's index of diversity. J. Clin. Microbiol. 26:2465–2466.
52. National Committee for Clinical Laboratory Standards. 2003. Susceptibility Testing of Mycobacteria,
Nocardiae, and Other Aerobic Actinomycetes: Approved Standard M24-A. NCCLS, Wayne, USA.
53. Smith, I. 2003. Mycobacterium tuberculosis pathogenesis and molecular determinants of virulence. Clin.
Microbiol. Rev. 16:463-496.
54. Li, Y.-J., L. Danelishvili, D. Wagner, M. Petrofsky, and L. E. Bermudez. 2010. Identification of virulence
determinants of Mycobacterium avium that impact on the ability to resist host killing mechanisms. J. Med.
Microbiol. 59:8-16.
20