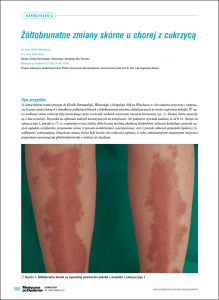
Nr wniosku: 191700, nr raportu: 13928. Kierownik (z rap.): dr Izabela Barbara Klich
Izolowany niedobór IgA (ang. Selective IgA Deficiency, SIgAD) jest najczęstszym, pierwotnym niedoborem
odporności związanym z zaburzeniem produkcji przeciwciał. Projekt badawczy pt. „Zastosowanie
innowacyjnych metod w poszukiwaniu związku podłoża genetycznego i wykładników dysregulacji
immunologicznej zaangażowanych w rozwój wrodzonego niedoboru IgA w patogenezie cukrzycy typu 1”
miał na celu poszerzenie wiedzy na temat roli podłoża genetycznego wraz z ekspresją określonych
wykładników immunologicznych w występowaniu niedoboru IgA u dzieci z cukrzycą typu 1. Dotychczas
przeprowadzone badania nie pozwoliły na jednoznaczne określenie genetycznego defektu leżącego u
podłoża tych chorób. Być może współistnienie wspólnego podłoża genetycznego wraz z charakterystycznym
panelem wykładników immunologicznych ma istotne znaczenie w obrazie klinicznym choroby, terapii czy
rokowaniu. Projekt zakładał stworzenie bazy danych pacjentów z cukrzycą i niedoborem IgA spowodowaną
mutacjami genów TNFRSF13B i ICOS zarówno punktowymi (sekwencjonowanie DNA), jak i
mikrodelecjami (metoda MPLA). Poszukiwanie mutacji w genach kandydatach przeprowadzono u 60 osób z
populacji polskiej u których zdiagnozowano współwystępowanie cukrzycy typu 1 z niedoborem IgAD. U
jednej z pacjentek stwierdzono, nie opisaną do tej pory zmianę z nukleotydu G na A w intronie 1 genu ICOS.
U badanych osób nie stwierdzono obecności mikrodelelcji. Dotychczas nie prowadzono na świecie badań
polimorfizmów genów TNFRSF13B i ICOS wśród pacjentów u których niedobór IgA współwystępował z
cukrzycą typu 1. Dlatego kolejnym celem pracy było badanie polimorfizmów jako czynników
modyfikujących przebieg kliniczny chorób. Zgenotypowano 12 polimorfizmów zlokalizowanych w genie
ICOS oraz 9 polimorfizmów w genie TNFRSF12B. Analiza nie wykazała zależności pomiędzy wariantami
polimorficznymi a przebiegiem klinicznym choroby. Analiza alleli HLA klasy II wykazała, że częstość
występowania allela DRB1*04 u osób z IgAD i cukrzycą typu 1 była wyższa wynosiła 25% w porównaniu z
kontrolą 11,8% (pc=0,01, OR(95%CI)=2,6(1,23-5,7)). Częstość allela DRB1*04 była jednak wyższa w
grupie osób z cukrzycą typu 1 niż u osób u których współwystępował niedobór IgA: 41,5% vs. 25%
(pc=0,01, OR(95%CI)=0,47(0,25-0,86)). Związek z cukrzycą typu 1 wykazywał również allel DRB1*03.
Jednak nie występuje on w grupie osób z cukrzyca z taką częstością jak allel DRB1*04, odpowiednio: 29% i
11% (pc<0,0001; OR(95%CI)=3,2(2,1-4,7)). W przypadku grupy badanej u której zdiagnozowano cukrzycę
typu 1 wraz niedoborem IgA, allel DRB1*03 występował znacznie częściej niż w grupie osób tylko z
cukrzycą typu 1:44,2% vs 29% (pc=0,024, OR(95%CI)=1,95(1,08-3,52)). Kolejne trzy allele genu DRB1:
DRB1*07, DRB1*11 i DRB1*1501 występowały z kolei częściej w grupie kontrolnej, niż w grupach osób
chorych, wykazując w ten sposób efekt protekcyjny. W przypadku genu DQA1 częstość allela DQA1*0301
była znacząco wyższa w grupie osób chorych zarówno tylko z cukrzycą typu 1 jak i ze współwystępującym
IgAD: 39,6% vs 11,2%; pc<0,0001; OR(95%CI)=5,2(3,5-7,6); 31,8% vs 11,2%; pc=0,0008; OR(95%CI)=
3,64 (1,7-7,77). Jednak obecność alleli DQA1*0101, DQA1*0102, DQA1*0201 nie wykazała związku z
cukrzycą typu 1. Istotnie częściej występowały one u osób zdrowych. Dwa spośród alleli genu DQB1
wykazywały związek z cukrzycą typu 1. Allele DQB1*0201 i DQB1*0302 obserwowano częściej wśród
chorych w porównaniu z kontrolą; odpowiednio: 36,5% vs 24,1%; pc=0,0003, OR(95%CI)=1,8(1,3-2,4) i
26,6% vs 6,7%; pc<0,0001, OR(95%CI)=5,(3,1-7,9) oraz 44,06% vs 24,1%; pc=0,0031,
OR(95%CI)=2,5(1,36-4,58) i 27,96% vs 6,7%; pc=0,0002 OR(95%CI)=5,8(2,27-14,82). Brak związku z
cukrzycą typu 1 stwierdzono dla alleli DQB1*0301, DQB1*0602 i *0603, które występowały częściej wśród
osób z grupy kontrolnej niż w grupach badanych. Nieprawidłowa regulacja cytokin bądź obecność
czynników działających antagonistycznie może stanowić jedną z cech determinujących upośledzoną syntezę
IgA. Stwierdzono istotną zależność pomiędzy polimorfizmem w genie ICOS (rs200852713) a stężeniem
cytokin: IL-1beta, IL-1ra, IL-5, IL-6, IL-7, IL-8, IL-9, G-CSF, INF-, MCP-1. Oczekiwane efekty
wdrożenia wyników projektu obejmowały ustalenie obiektywnych i wydajnych diagnostycznie markerów
defektów genetycznych TNFRSF13B, ICOS, co mogłoby znaleźć zastosowanie w diagnostyce i terapii
pacjentów ze zdiagnozowaną cukrzycą typu 1 i towarzyszącym jej niedoborem IgA oraz przyspieszyć i
obniżyć koszty badań genetycznych nad ustaleniem terapii celowanej. Zastosowanie praktyczne markerów
białkowych pozwoliłoby na porównywanie pacjentów o zróżnicowanym fenotypie klinicznym. Wyzwaniem
dla wykonawców projektu był nie tylko postęp naukowy, ale również możliwość zaoferowania rezultatów
tego postępu pacjentom. Realizacja niniejszego projektu może mieć istotne implikacje naukowe i praktyczne,
które pozwolą na stworzenie zaleceń postępowania diagnostycznego u dzieci z podejrzeniem niedoboru IgAi
cukrzycy typu 1. Badania w tym Projekcie nad udziałem czynników genetycznych w etiopatogenezie tych
chorób mogą dać możliwość opracowania modelu poradnictwa genetycznego dla pacjentów z tą wadą i ich
rodzin. Istniejący stan wiedzy w zakresie tematu badań jaki również oryginalny wkład wniesie rozwiązanie
postawionego problemu do dorobku danej dyscypliny naukowej w świecie i w Polsce.