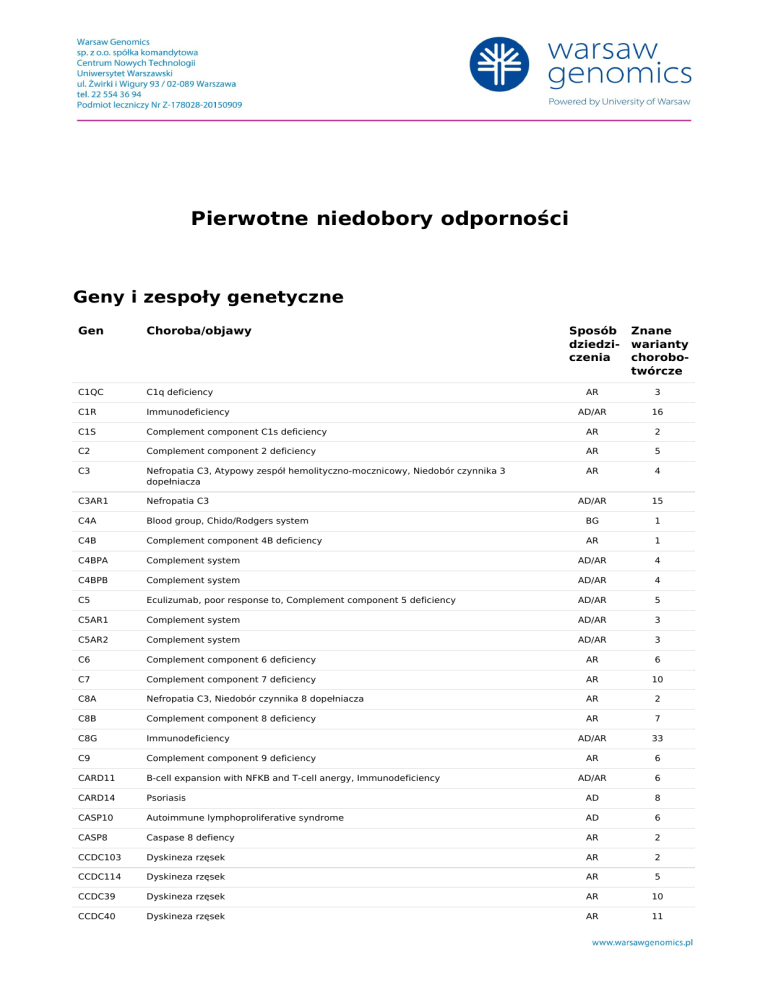
Pierwotne niedobory odporności
Geny i zespoły genetyczne
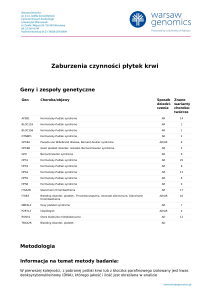
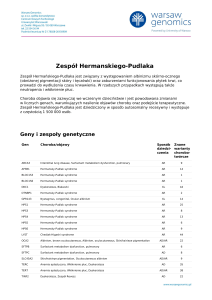
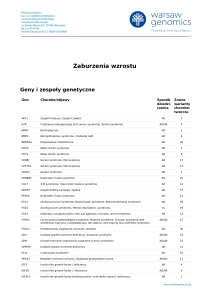
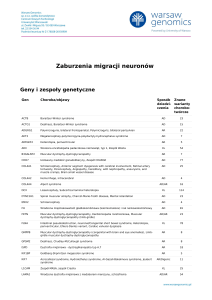
Gen
Choroba/objawy
C1QC
C1q deficiency
C1R
Immunodeficiency
C1S
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
AR
3
AD/AR
16
Complement component C1s deficiency
AR
2
C2
Complement component 2 deficiency
AR
5
C3
Nefropatia C3, Atypowy zespół hemolityczno-mocznicowy, Niedobór czynnika 3
dopełniacza
AR
4
C3AR1
Nefropatia C3
AD/AR
15
C4A
Blood group, Chido/Rodgers system
BG
1
C4B
Complement component 4B deficiency
AR
1
C4BPA
Complement system
AD/AR
4
C4BPB
Complement system
AD/AR
4
C5
Eculizumab, poor response to, Complement component 5 deficiency
AD/AR
5
C5AR1
Complement system
AD/AR
3
C5AR2
Complement system
AD/AR
3
C6
Complement component 6 deficiency
AR
6
C7
Complement component 7 deficiency
AR
10
C8A
Nefropatia C3, Niedobór czynnika 8 dopełniacza
AR
2
C8B
Complement component 8 deficiency
AR
7
C8G
Immunodeficiency
AD/AR
33
C9
Complement component 9 deficiency
AR
6
CARD11
B-cell expansion with NFKB and T-cell anergy, Immunodeficiency
AD/AR
6
CARD14
Psoriasis
AD
8
CASP10
Autoimmune lymphoproliferative syndrome
AD
6
CASP8
Caspase 8 defiency
AR
2
CCDC103
Dyskineza rzęsek
AR
2
CCDC114
Dyskineza rzęsek
AR
5
CCDC39
Dyskineza rzęsek
AR
10
CCDC40
Dyskineza rzęsek
AR
11
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
CCDC65
Dyskineza rzęsek
AR
2
CCNO
Dyskineza rzęsek
AR
7
CD19
Immunodeficiency, common variable
AR
2
CD247
Immunodeficiency
AR
7
CD27
Lymphoproliferative syndrome
AR
2
CD3D
Immunodeficiency
AR
3
CD3E
Immunodeficiency
AR
3
CD3G
Immunodeficiency
AR
3
CD40
Immunodeficiency with Hyper-IgM
AR
4
CD40LG
Immunodeficiency, with hyper-IgM
XL
19
CD46
Nefropatia C3, Atypowy zespół hemolityczno-mocznicowy
AD/AR
7
CD55
Blood group, Cromer system
BG
1
CD59
CD59 deficiency
AR
3
CD8A
CD8 deficiency
AR
1
CD93
Complement system
AD/AR
8
CECR1
Polyarteritis nodosa, ADA2 deficiency
AR
11
CFB
Nefropatia C3, Atypowy zespół hemolityczno-mocznicowy, Niedobór czynnika B
dopełniacza
AD/AR
3
CFD
Complement factor D deficiency
AR
2
CFH
Nefropatia C3, Atypowy zespół hemolityczno-mocznicowy, Niedobór czynnika H
dopełniacza
AD/AR
14
CFHR1
Nefropatia C3, Atypowy zespół hemolityczno-mocznicowy
AD/AR/Dig
enic
6
CFHR3
Nefropatia C3, Atypowy zespół hemolityczno-mocznicowy
AD/AR/Dig
enic
6
CFI
Nefropatia C3, Atypowy zespół hemolityczno-mocznicowy, Niedobór czynnika I
dopełniacza
AD/AR
5
CFP
Properdin deficiency
XL
5
CIITA
Bare lymphocyte syndrome
AR
6
CLU
Complement system
AD/AR
42
COLEC11
3MC syndrome
AR
7
CORO1A
Immunodeficiency
AR
22
CR1
Nefropatia C3
BG
1
CR2
Common variable immunodeficiency
AR
2
CRP
Complement system
AD/AR
4
CSF2RA
Surfactant metabolism dysfunction, pulmonary
XL
2
CTC1
Cerebroretinal microangiopathy with calcifications and cysts
AR
13
CTLA4
Pierwotne niedobory odporności
AD
6
CTSC
Periodontitis, juvenile, Haim-Munk syndrome, Papillon-Lefevre syndrome
AR
15
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
CYBA
Chronic granulomatous disease
AR
11
CYBB
Chronic granulomatous disease, Immunodeficiency
XL
29
DCLRE1C
Omenn syndrome, Severe combined immunodeficiency with sensitivity to ionizing
radiation
AR
15
DDX58
Singleton-Merten syndrome
AD
2
DGKE
Nefropatia C3, Atypowy zespół hemolityczno-mocznicowy, Zespół nerczycowy
AR
10
DKC1
Dyskeratoza, Białaczki
XL
44
DNAAF1
Dyskineza rzęsek
AR
7
DNAAF2
Dyskineza rzęsek
AR
5
DNAAF3
Pierwotna dyskineza rzęsek
AD/AR
3
DNAAF5
Dyskineza rzęsek
AR
1
DNAH11
Dyskineza rzęsek
AR
8
DNAH5
Dyskineza rzęsek
AR
25
DNAI1
Dyskineza rzęsek
AR
6
DNAI2
Dyskineza rzęsek
AR
5
DNAL1
Dyskineza rzęsek
AR
2
DNMT3B
Immunodeficiency-centromeric instability-facial anomalies syndrome
AR
12
DOCK2
Immunodeficiency
AR
5
DOCK8
Hyper-IgE recurrent infection syndrome
AR
23
DRC1
Pierwotna dyskineza rzęsek
AD/AR
3
DYX1C1
Dyskineza rzęsek
AR
4
ELANE
Neutropenia, Zespół mielodysplastyczny, Białaczki
AD
14
FAS
Autoimmune lymphoproliferative syndrome
AD/AR
20
FCN1
Complement system
AD/AR
26
FCN2
Complement system
AD/AR
26
FCN3
Immunodeficiency due to Ficolin 3 deficiency
AR
1
FERMT3
Leukocyte adhesion deficiency
AR
8
FOXP3
Poliendokrynopatia i endokrynopatia, Cukrzyca typu I
XL
15
G6PC3
Neutropenia, severe congenital, Dursun syndrome
AR
11
G6PD
Glucose-6-phosphate dehydrogenase deficiency
XL
34
GATA2
Zespół mielodysplastyczny, Neutropenia z monocytopenią, Ostra białaczka szpikowa,
Niedobory odporności
AD
18
HAX1
Neutropenia, severe congenital
AR
8
HYDIN
Pierwotna dyskineza rzęsek
AD/AR
3
IFIH1
Singleton-Merten syndrome
AD
9
IFNGR1
Immunodeficiency
AD/AR
15
IFNGR2
Immunodeficiency
AR
4
IGHM
Agammaglobulinemia
AR
5
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
IGLL1
Agammaglobulinemia
AR
2
IKBKG
Incontinentia pigmenti, Ectodermal, dysplasia, anhidrotic, lymphedema and
immunodeficiency, Immunodeficiency, Invasive pneumococcal disease, recurrent, isolated
XL
30
IL10RA
Inflammatory bowel disease
AR
4
IL10RB
Inflammatory bowel disease
AR
2
IL12RB1
Immunodeficiency
AR
8
IL1RN
Osteomyelitis, sterile multifocal, with periostitis and pustulosis
AR
5
IL2RA
Interleukin 2 receptor, alpha, deficiency
AR
5
IL2RG
Combined immunodeficiency
XL
19
IL36RN
Pustular psoriasis, generalized
AR
8
IL7
Interleukin 7 deficiency, Generalized verrucosis, HPV susceptibility
IL7R
Severe combined immunodeficiency, , T-cell negative, B-cell positive, NK cell positive
AR
17
ISG15
Immunodeficiency, with basal ganglia calcification
AR
3
ITGB2
Leukocyte adhesion deficiency
AR
28
ITK
Lymphoproliferative syndrome
AR
3
JAGN1
Neutropenia, severe congenital
AR
8
JAK3
Severe combined immunodeficiency, , T cell-negative, B cell-positive, natural killer cellnegative
AR
12
LCK
Immunodeficiency
AR
2
LIG4
Severe combined immunodeficiency with sensitivity to ionizing radiation, LIG4 syndrome
AR
5
LPIN2
Majeed syndrome
AR
5
LRBA
Common variable immunodeficiency
AR
10
LRRC6
Dyskineza rzęsek
AR
8
LYST
Chediak-Higashi syndrome
AR
44
MAGT1
Niedobory odporności, Hipomagnezemia
XL
4
MALT1
Immunodeficiency
AR
3
MASP1
3MC syndrome
AR
6
MASP2
MASP2 deficiency
AR
1
MAT2A
Complement system
AD/AR
2
MEFV
Familial Mediterranean fever
AD/AR
19
MRE11A
Rak piersi
AD
25
MVK
Mevalonic aciduria, Hyper-IgD syndrome
AR
20
NBN
Rak piersi, Zespół Nijmegen
AD/AR
45
NCF1
Chronic granulomatous disease
AR
14
NCF2
Chronic granulomatous disease
AR
11
NCF4
Granulomatous disease
AR
3
NFKB1
Common variable immunodeficiency
AD
3
NFKB2
Common variable immunodeficiency
AD
7
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
NFKBIA
Ectodermal dysplasia, anhidrotic, with T-cell immunodeficiency
AD
3
NHEJ1
Severe combined immunodeficiency with microcephaly, growth retardation, and
sensitivity to ionizing radiation
AR
7
NHP2
Dyskeratosis congenita
AR
3
NLRP12
Familial cold autoinflammatory syndrome
AD
3
NLRP3
Neonatal onset multisystem inflammatory disease (NOMID), Familial cold
autoinflammatory syndrome, Muckle-Wells syndrome, Chronic infantile neurologic
cutaneous articular (CINCA) syndrome
AD
10
NME8
Dyskineza rzęsek
AR
2
NOD2
Choroba Leśniowskiego i Crohna, zespół Blau, sarkoidoza
MG
18
NOP10
Dyskeratosis congenita
AR
1
NRAS
Zespół Noonan
AD
16
OFD1
Simpson-Golabi-Behmel syndrome, Retinitis pigmentosa, Orofaciodigital syndrome,
Joubert syndrome
XL
122
ORAI1
Immunodeficiency
AR
7
PIGA
Multiple congenital anomalies-hypotonia-seizures syndrome
XL
18
PIK3CD
Immunodeficiency
AD
4
PIK3R1
Agammaglobulinemia
AR
13
PLCG2
Familial cold autoinflammatory syndrome 3 (PLAID), Autoinflammation, antibody
deficiency, and immune dysregulation syndrome (APLAID)
AD
4
PMS2
Zespół Lyncha
AD/AR
123
PNP
Purine nucleoside phosphorylase deficiency
AR
10
PRF1
Chłoniak nieziarniczy, Anemia aplastyczna, Limfohistiocytoza
AR
16
PRKDC
Immunodeficiency
AR
5
PSMB8
Nakajo-Nishimura syndrome, Chronic atypical neutrophilic dermatosis with lipodystrophy
and elevated temperature syndrome, Autoinflammation, lipodystrophy, and dermatosis
syndrome, Joint contractures, muscular atrophy, microcytic anemia, and panniculitisinduced lipodystrophy syndrome
AR
4
PSTPIP1
Pyogenic sterile arthritis, pyoderma gangrenosum, and acne
AD
2
PTPRC
Severe combined immunodeficiency, , T-cell negative, B-cell positive, NK cell positive
AR
4
PTX3
Complement system
AD/AR
9
RAB27A
Griscelli syndrome, Elejalde syndrome
AR
10
RAG1
Omenn syndrome, Alpha/beta T-cell lymphopenia with gamma/delta T-cell expansion,
severe cytomegalovirus infection, and autoimmunity, T cell-negative, B cell-negative,
natural killer cell-positive severe combined immunodeficiency, Combined cellular and
humoral immune defects with granulomas
AR
29
RAG2
Omenn syndrome, Combined cellular and humoral immune defects with granulomas
AR
18
RFX5
Bare lymphocyte syndrome
AR
5
RFXANK
MHC class II deficiency
AR
5
RFXAP
Bare lymphocyte syndrome
AR
5
RHOH
T-cell immunodeficiency with epidermodysplasia verruciformis
AD/AR
3
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
RMRP
Cartilage-hair hypoplasia, Metaphyseal dysplasia without hypotrichosis, Anauxetic
dysplasia
AR
22
RNASEH2A
Aicardi-Goutières syndrome
AR
12
RNASEH2B
Aicardi-Goutières syndrome
AR
4
RNASEH2C
Aicardi-Goutières syndrome
AR
4
RPGR
Zwyrodnienie barwnikowe siatkówki, dystrofia pręcikowo-czopkowa
XL
38
RSPH1
Dyskineza rzęsek
AR
9
RSPH4A
Dyskineza rzęsek
AR
8
RSPH9
Dyskineza rzęsek
AR
2
RTEL1
Pulmonary fibrosis and/or bone marrow failure, Dyskeratosis congenita
AD/AR
26
SAMHD1
Aicardi-Goutières syndrome
AR
23
SBDS
Anemia aplastyczna, Zespół Shwachmana-Diamonda
AD/AR
12
SERPING1
Angioedema
AD/AR
14
SH2D1A
Lymphoproliferative syndrome
XL
14
SLC37A4
Glycogen storage disease
AR
26
SMARCAL1
Schimke immunoosseous dysplasia
AR
9
SP110
Hepatic venoocclusive disease with immunodeficiency
AR
7
SPAG1
Pierwotna dyskineza rzęsek
AD/AR
8
SPINK5
Netherton syndrome
AR
3
STAT1
Immunodeficiency
AD/AR
27
STAT2
Immunodeficiency
AR
2
STAT3
Hyper-IgE recurrent infection syndrome, Autoimmune disease, multisystem, infantile
onset
AD
26
STAT4
Behçet disease, Juvenile rheumatoid factor-negative polyarthritis, Oligoarticular juvenile
arthritis, Pediatric systemic lupus erythematosus
AD/AR
12
STAT5B
Growth hormone insensitivity with immunodeficiency
AR
5
STIM1
Stormorken syndrome, Immunodeficiency
AD/AR
10
STK4
T-cell immunodeficiency syndrome, recurrent infections, autoimmunity,
AR
3
STXBP2
Hemophagocytic lymphohistiocytosis, familial
AR
7
TAP1
Bare lymphocyte syndrome
AR
2
TAP2
Bare lymphocyte syndrome
AR
2
TAPBP
Bare lymphocyte syndrome
AR
1
TBX1
Conotruncal anomaly face syndrome
AD
6
TCIRG1
Osteopetrosis
AR
9
TERC
Anemia aplastyczna, Włóknienie płuc, Dyskeratoza
AD
35
TERT
Anemia aplastyczna, Włóknienie płuc, Dyskeratoza
AD/AR
38
THBD
Nefropatia C3, Atypowy zespół hemolityczno-mocznicowy, Trombofilia związana
z niedoborami trombomoduliny
AD
5
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
TINF2
Dyskeratoza, Zespół Revesz
AD
22
TMEM173
STING-associated vasculopathy, infantile-onsent (SAVI)
AD
3
TNFRSF13B
Common variable immunodeficiency, Immunoglobulin A deficiency
AD/AR
7
TNFRSF1A
Periodic fever (TNF receptor-associated periodic syndrome)
AD
13
TNFRSF4
Immunodeficiency
AR
1
TRAC
T-cell receptor-alpha/beta deficiency
AR
1
TREX1
Vasculopathy, retinal, with cerebral leukodystrophy, Chilblain lupus, Aicardi-Goutières
syndrome
AD/AR
22
TYK2
Immunodeficiency
AR
6
UNC119
Immunodeficiency
AR
2
USB1
Poikiloderma with neutropenia
AR
4
VSIG4
Complement system
AD/AR
74
VTN
Complement system
AD/AR
1
WAS
Neutropenia, severe congenital, Thrombocytopenia, Wiskott-Aldrich syndrome
XL
28
WRAP53
Dyskeratosis congenita
AR
5
XIAP
Lymphoproliferative syndrome
XL
4
ZAP70
Selective T-cell defect
AR
10
ZMYND10
Dyskineza rzęsek
AR
6
Metodologia
Informacja na temat metody badania:
W pierwszej kolejności, z pobranej próbki krwi lub z bloczka parafinowego izolowany jest kwas
deoksyrybonukleinowy (DNA), którego jakość i ilość jest określana w analizie
spektrofotometrycznej i fluorymetrycznej. Po mechanicznej lub enzymatycznej fragmentacji,
DNA jest wykorzystywany do stworzenia biblioteki, umożliwiającej oznaczenie, a następnie
zsekwencjonowanie i analizę genów, które zostały wybrane w ramach zleconego panelu.
Otrzymana biblioteka jest sekwencjonowana na sekwenatorze nowej generacji. Otrzymane
wyniki zostają następnie poddane analizie bioinformatycznej i interpretacji klinicznej. Warianty
genetyczne są identyfikowane z wykorzystaniem Burrows-Wheeler Aligner. Test umożliwia
wykrycie 100% substytucji i 95% małych insercji i delecji.
Informacja na temat klasyfikacji wariantów:
W raporcie z badania przedstawiana jest informacja na temat wariantów zaklasyfikowanych
jako warianty „potencjalnie patogenne” i „patogenne”, z uwagi na ich potencjalne znaczenie
kliniczne. Zidentyfikowane warianty są klasyfikowane do następujących kategorii:
Wariant patogenny: znaleziona zmiana w sekwencji genu ma bezpośredni związek
z powstawaniem choroby. Równocześnie, niektóre zmiany patogenne mogą nie mieć pełnej
penetracji, tj. pojedyncza zmiana może być niewystarczająca do wywołania pełnoobjawowej
choroby.
Wariant potencjalnie patogenny: znaleziona zmiana w sekwencji genu jest z dużym
prawdopodobieństwem związana z powstawaniem choroby, jednakże udowodnienie tego
związku nie jest możliwe w oparciu o aktualnie dostępne dane naukowe. Potwierdzenie
patogenności wariantu wymaga dodatkowych badań i dowodów; nie można wykluczyć, że
dalsze badania wykażą, że znaleziona zmiana ma niewielkie lub żadne znaczenie kliniczne.
Wariant o nieznanej patogenności: w oparciu o aktualnie dostępne dane naukowe nie ma
możliwości określenia znaczenia znalezionej zmiany.
Wariant potencjalnie łagodny: znaleziona zmiana w sekwencji genu najprawdopodobniej nie
ma związku z powstawaniem choroby, jednakże w oparciu o aktualnie dostępne dane naukowe
nie ma możliwości potwierdzenia łagodności zmiany. Potwierdzenie klinicznego znaczenia
wariantu wymaga dodatkowych badań i dowodów; nie można wykluczyć, że dalsze badania
wykażą, że znaleziona zmiana ma znaczenie kliniczne i prowadzi do rozwinięcia choroby
Wariant łagodny: znaleziona zmiana nie ma związku z powstawaniem choroby
Zidentyfikowane warianty genetyczne klasyfikowane są w oparciu o wytyczne opracowane
przez American College of Medical Genetics and Genomics i American Association for Molecular
Pathology (S. Richards, Genet Med. 2015 May;17(5):405-24). W klasyfikacji wariantów brane są
pod uwagę następujące kryteria:
wcześniejsza identyfikacja wariantu u osób obciążonych chorobą
wpływ wariantu na powstawanie funkcjonalnego produktu genu:
określony w analizach bioinformatycznych
potwierdzony w badaniach in vitro/in vivo
lokalizacja wariantu (ekson/intron, domena funkcjonalna)
zmiana de novo/dziedziczna
częstość występowania wariantu w populacji ogólnej (każdy wariant występujący
z częstością >5% zgodnie z Exome Sequencing Project, 1000 Genomes Project lub
Exome Aggregation Consortium jest klasyfikowany jako zmiana łagodna)
częstość występowania wariantu w populacji ogólnej w stosunku do populacji osób
chorych
Ostateczna klasyfikacja wariantów prowadzona jest w oparciu o sumę wymienionych kryteriów.
Przeszukiwane bazy danych obejmują: 1000GP, ClinVar, ConsensusPathDB, Exome Aggregation
Consortium, Exome Variant Server, FATHMM, GO (Gene Ontology), GTEx (Genotype-Tissue
Expression), GWAS (Genome Wide Association Study), HGMD, KEGG, MetaLR, MetaSVM,
MutationAssessor, MutationTaster, OMIM, PolyPhen-2, PROVEAN, SIFT, SnpEff, dbNSFP, UniProt,
VEP (Variant Effect Predictor).
Ograniczenia badania:
Wszystkie technologie sekwencjonowania mają swoje ograniczenia. Zlecane badanie jest
wykonywane z wykorzystaniem sekwencjonowania nowej generacji (NGS) i ma na celu
zbadanie regionów kodujących i splicingowych zleconych genów. Chociaż stosowane techniki
sekwencjonowania oraz późniejsze analizy bioinformatyczne są ukierunkowane na ograniczenie
znaczenia sekwencji pseudogenów, to jednak obecność wysoce homologicznych sekwencji
genowych może nadal sporadycznie zakłócać zdolność identyfikacji patogennych alleli, jak
i delecji/duplikacji. Sekwencjonowanie Sangera jest metodą wykorzystywaną do potwierdzania
wariantów, które uzyskały niższe parametry jakości. Analizy delecji/duplikacji wskazują na
zmiany ilościowe DNA obejmujące minimum jeden ekson i zawsze wymagają potwierdzenia
innymi metodami (qPCR lub MLPA). Wykonane analizy nie są przeznaczone do wykrywania
pewnych typów zmian genomowych, jak translokacje, inwersje, mutacje dynamiczne (np.
zwiększenie ilości powtórzeń trzynukleotydowych), zmian w regionach regulatorowych czy
intronowych. Jeśli raportowane jest zwiększenie liczby powtórzeń dwu- czy trzynukleotydowych,
to trzeba założyć, że dokładna liczba powtórzeń nie jest precyzyjna. Przeprowadzane badanie
nie jest przeznaczone do wykrywania mozaikowatości somatycznych, a analizy mutacji
somatycznych powinny być prowadzone w kontekście sekwencji DNA germinalnego.
Nie ma możliwości wykluczenia obecności mutacji w genach i rejonach innych niż objęte
wykonywanym badaniem, a także zmian liczby kopii genu. Raport z badania zawiera informację
na temat zmian w sekwencji genów zidentyfikowanych w oparciu o porównanie z aktualnymi
sekwencjami referencyjnymi zdeponowanymi w bazach danych NCBI Nucleotide i Ensembl.
Testy są opracowywane w Warsaw Genomics do celów klinicznych. Wszystkie otrzymywane
wyniki badań są interpretowane i analizowane przez ekspertów naukowych i medycznych
Warsaw Genomics.
Jak zlecić badanie
Informacja na temat metody badania:
Badanie można zlecić bezpośrednio na stronie internetowej Warsaw Genomics, poprzez
zaznaczenie wybranego testu. Zalecamy jednak, by przed każdym badaniem skonsultować się
z lekarzem, który pomoże w wybraniu odpowiedniego testu diagnostycznego, wyjaśni
możliwości i ograniczenia testów genetycznych a także przedstawi możliwe wyniki
i konsekwencje przeprowadzenia badania.
Czas realizacji badania: od 4 do 10 tygodni. Będziemy informować o kolejnych etapach analizy.
KONSULTACJA LEKARSKA
REJESTRACJA
Wybranie właściwego testu
genetycznego lub
indywidualnego zestawu
genów
Wypełnienie formularza
zlecenia testu.
Formularz wypełnia pacjent
lub wybrany przez niego
lekarz.
OPŁACENIE TESTU
Po wykonaniu testu
POBRANIE KRWI
Łącznie należy pobrać 4ml
krwi do jednej probówki
z EDTA (takiej jak na
morfologie). Pobraną krew
można przechowywać
w lodówce (w temp. +4st C)
do 7 dni
WYNIK BADANIA
PRZESŁANIE PRÓBKI
KRWI NA NASZ ADRES
Próbkę można dostarczyć
osobiście lub kurierem
(w temperaturze pokojowej)
w ciągu 48 godzin.
Szczegółowa instrukcja
pakowania próbki
i zamówienia kuriera jest
dostępna tutaj. Do próbki
dołączamy wydrukowany
i podpisany formularz
zlecenia testu.
KONSULTACJA LEKARSKA
zostanie przekazany osobie
zlecającej test – pacjentowi
lub wybranemu przez niego
lekarzowi
Jak przekazać materiał do badania?
Badanie genetyczne z krwi:
1. Krew należy pobrać do jednej probówki z EDTA (nie pobierać do probówek na skrzep,
ani na heparynę litową). Pobranie krwi może nastąpić w dowolnej godzinie, pacjent nie
musi być na czczo:
osoba dorosła - ok. 4 ml krwi żylnej do izolacji DNA (pobraną krew należy
dokładnie wymieszać z antykoagulantem i przechowywać w temperaturze 4°C)
dzieci – ok. 4 ml (minimalna ilość 2 ml) krwi żylnej do izolacji DNA (pobraną
krew należy dokładnie wymieszać z antykoagulantem i przechowywać
w temperaturze 4°C)
niemowlę – ok. 1,5 - 2 ml krwi żylnej do izolacji DNA (pobraną krew należy
dokładnie wymieszać z antykoagulantem i przechowywać w temperaturze 4°C)
2. Probówkę należy opisać imieniem i nazwiskiem i zabezpieczyć (można zakleić taśmą
klejącą).
3. Zabezpieczoną probówkę wraz z wypełnionym i podpisanym formularzem zlecenia
badania należy zapakować i wysłać zgodnie z instrukcją dostępną pod adresem:
https://badamygeny.pl/BADAMY_GENY/docs/instrukcja-wysylki-probki-krwi.pdf
Badanie genetyczne z bloczka parafinowego (profilowanie
nowotworu):
1. Należy pobrać łącznie 4 ml krwi do jednej probówki z EDTA (nie pobierać do probówek
na skrzep, ani na heparynę litową). Pobranie krwi może nastąpić w dowolnej godzinie,
pacjent nie musi być na czczo. Pobraną krew należy dokładnie wymieszać
z antykoagulantem i przechowywać w temperaturze 4°C.
2. Probówkę należy opisać imieniem i nazwiskiem i zabezpieczyć (można zakleić taśmą
klejącą).
3. Uzyskanie tkanki nowotworowej do badania w postaci:
bloczka parafinowego zawierającego wycinek nowotworu wraz z uzyskanym
z bloczka preparatem histopatologicznym (szkiełkiem) umożliwiającym
zlokalizowanie fragmentu tkanki nowotworowej,
albo wycinka tkanki nowotworowej z bloczka parafinowego o wymiarach min.
4x4x1mm, zawierającego wyłącznie tkankę nowotworową.
4. Próbkę należy opisać imieniem i nazwiskiem i zabezpieczyć (można zakleić taśmą
klejącą).
5. Zabezpieczony materiał wraz z wypełnionym i podpisanym formularzem zlecenia
badania należy zapakować i wysłać zgodnie z instrukcją dostępną pod adresem:
https://badamygeny.pl/BADAMY_GENY/docs/instrukcja-wysylki-probki-krwi.pdf
Powered by TCPDF (www.tcpdf.org)