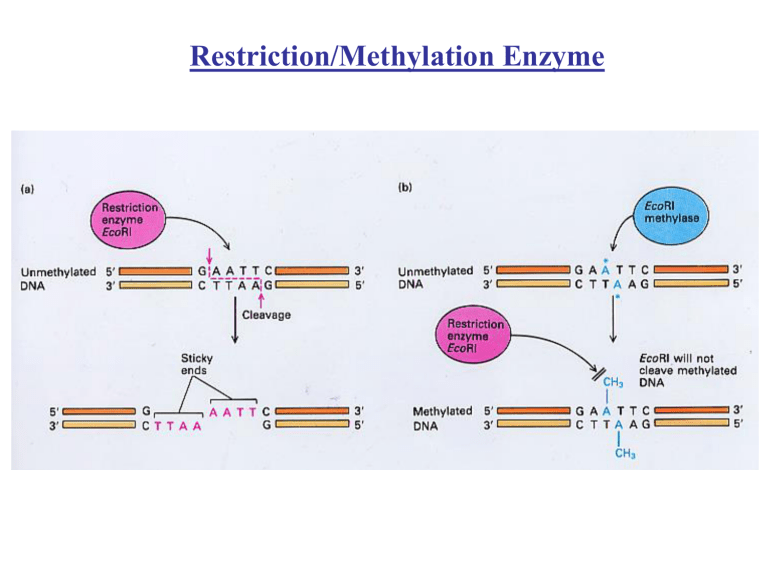
Restriction/Methylation Enzyme
Eco RI Restriction Enzyme
Single stranded “nick”
• First restriction enzyme from Escherichia coli, so Eco R1
Restriction Enzyme Recognition Sites
Restriction sites are general palindromic:
5’-GGATCC-3’
Bam H1 site:
3’-CCTAGG-5’
Restriction Enzyme Recognition Sites
BglII
5’ A-G-A-T-C-T
T-C-T-A-G-A 5’
Sau3A
BamHI
5’ G-A-T-C
C-T-A-G 5’
All these sticky ends
are compatible
5’ G-G-A-T-C-C
C-C-T-A-G-G 5’
Isoschizomers: In certain cases, two or more different enzymes may
recognize identical sites. (e.g. MboI also cleaves at GATC, and so is an
isochizomer of Sau3A.)
Frequency of cutting of recognition enzymes
Sau 3A (GATC) cuts (¼)(¼)(¼)(¼) = once every 256 base pairs
(assuming G/C = A/T, which is often does not)
BamH1 (GGATCC) cuts (¼)(¼)(¼)(¼)(¼)(¼) = once every ~4Kb
HindII (GTPyPuAC) cuts (¼)(¼)(½)(½)(¼)(¼) = once every ~1Kb
“Sticky” ends
5’ overhang (EcoRI)
5’-GAATTC-3’
3’-CTTAAG-5’
5’-G-OH
3’-CTTAA-PO4
3’ overhang (PstI)
5’-CTGCAG-3’
3’-GACGTC-5’
5’-CTGCA-OH
3’-G-PO4
+
PO4-AATTC-3’
HO-G-5’
+
PO4-G-3’
HO-ACGTC-5’
+
PO4-GGG-3’
HO-CCC-5’
“Blunt” ends
5’ overhang (SmaI)
5’-CCCGGG-3’
3’-GGGCCC-5’
5’-CCC-OH
3’-GGG-PO4
Ligation of compatible sticky ends
Human DNA cleaved with EcoRI
5’-C-G-G-T-A-C-T-A-G-OH
3’-G-C-C-A-T-G-A-T-C-T-T-A-A-PO4
Corn DNA cleaved with EcoRI
+
PO4-A-A-T-T-C-A-G-C-T-A-C-G-3’
HO-G-T-C-G-A-T-G-C-5’
Complementary base pairing
5’-A-C-G-G-T-A-C-T-A-G A-A-T-T-C-A-G-C-T-A-C-G-3’
3’-T-G-C-C-A-T-G-A-T-C-T-T-A-A G-T-C-G-A-T-G-C-5’
+ DNA Ligase, + rATP
5’-A-C-G-G-T-A-C-T-A-G-A-A-T-T-C-A-G-C-T-A-C-G-3’
3’-T-G-C-C-A-T-G-A-T-C-T-T-A-A-G-T-C-G-A-T-G-C-5’
recombinant DNA molecule
Agarose Gel Electrophoresis
_
DNA is negatively
charged from the
phosphate backbone
Agarose mesh
+
Visualize DNA with ethidium
bromide – fluoresces orange
ONLY when bound to DNA
•
•
•
•
•
Enzymy restrykcyjne izolowane z różnych gatunków
bakterii podzielono na trzy klasy (I, II i III)
w zależności od:
Liczby i organizacji wchodzących w ich skład
podjednostek
Wymagań dotyczących kofaktorów
Mechanizmu enzymatycznego
Specyficzności rozpoznawanej sekwencji
Regulacji ekspresji genów kodujących enzymy
Podział ten jest stale modyfikowany, uaktualniany w
związku z odkrywaniem nowych systemów RM
Typ I RM
•
•
•
•
•
•
Jest najbardziej skomplikowanym systemem, złożonym z
trzech podjednostek strukturalno-funkcjonalnych:
Podjednostka S – rozpoznaje sekwencję DNA
Podjednostka M – modyfikuje DNA
Podjednostka R – aktywność restrykcyjna
Podjednostki S i M tworzą m6A-metylazę DNA o stechiometrii
M2S1, która rozpoznaje i modyfikuje DNA w obrębie określonej
sekwencji
Kompleks 3 podjednostek R2M2S1 jest enzymem restrykcyjnym
(wymaga ATP) gdy napotka niezmodyfikowany DNA
Cięcie następuje w różnych niezdefiniowanych odległościach
od miejsca rozpoznania, zwykle kilkaset do kilku tysięcy par
zasad
Typ II RM
• W zdefiniowanych warunkach posiadają wysoką
specyficzność rozpoznawanej sekwencji
• Dają powtarzalne produkty trawienia
endonukleolitycznego
• Nie wymagają ATP i S-adenozylo-L-metioniny, a jedynie
jonów Mg
• Aktywności metylazy i endonukleazy rozdziolone są
między dwa odrębne białka kodowane przez różne geny
• Rozpoznają krótkie najczęściej palindromiczne
sekwencje 4-8 pz i trawią DNA w obrębie sekwencji
rozpoznania lub w pewnej ściśle określonej odległości
od niej
• Wyróżnia się podtypy lub klasy w obrębie rodziny II
Klasy typu II RM
• II S (monomery w roztworze; rozpoznają asymetryczną
sekwencję; cięcie w zdefiniowanej odległości od
sekwencji rozpoznawanej, 1-20 pz; np. FokI –
GGATGN9/13
• II E (rozpoznawane dwie sekwencje: w efektorze
allosterycznym i właściwej sekwencji ciętej, np. NaeI –
GCG/CGC)
• II F (homotetramer, rozpoznaje dwie sekwencje,
trawienie jednoczesne obu miejsc, np. NgoMIV –
G/CCGGC
• II T (heterodimer, rozpoznawana sekwencja
asymetryczna, sekwencja palindromiczna, np. Bpu10I –
CC/TNAGG
Klasy typu II RM c.d.
• II G (aktywność R i M w jednym łańcuchu
polipeptydowym, cięcie poza sekwencją
rozpoznania, stymulacja przez SAM, np. Eco57I
- CTGAAGN14/16)
• II B (trawienie po obu stronach rozpoznawanej
sekwencji, aktywność R i M w jednym Łańcuchu
polipeptydowym, np. BcgI –
NN/N10CGAN6TGCN10/NN)
• II M (rozpoznawana sekwencja zmetylowana,
np. DpnI – GmA/TC)
Typ III RM
• Zbudowany z 2 podjednostek: M –
modyfikującej i R – restrykcyjnej,
występujących w stechiometrii R2M2
• Enzymy rozpoznają 5-6 pz, nie
wykazujące symetrii wewnętrznej i trawią
w odległości około 25 pz od miejsca
rozpoznania
• Wymagają do aktywności ATP i Sadenozylo-L-metioniny
Enzymy użyteczne w analizie
restrykcyjnej DNA. Podział na grupy.
• enzymy należące do grupy "regularnych 6t-ek", np.:
EcoRI, BamHI, BglII, PstI, HindIII
• enzymy należące do grupy rozpoznających kilka
specyficzności, np.:
HincII - GT[PyPu]AC, AccI, AvaII, AflIII
• enzymy należące do grupy rozpoznających nieciągłe
sekwencje palindromowe, np.:
BglI (GCCNNNN'NGGC), BstXI (CGANNNNN'NTGG)
• enzymy rozpoznające specyficzną sekwencję, lecz
przecinające DNA poza nią ("shiftery", klasa lI S), np.:
FokI
GGATGNNNNNNNNN(9)’
CCTACNNNNNNNNNNNNN(13)’
MboII
GAAGANNNNNNNN(8)’
CTTCTNNNNNNN(7)’
Grupy enzymów pozostawiających
komplementarne końce
Istnieją grupy enzymów rozpoznające
odmienne sekwencje lecz pozostawiające po
trawieniu komplementarne końce DNA. Są one
dostępne komercyjnie i dają duże możliwości w
opracowaniu strategii wieloetapowego
klonowania.
Grupy enzymów pozostawiających
komplementarne końce
Najpopularniejsze pary enzymów
pozostawiających komplementarne końce
to:
• BamHI
G’GATC’C
• BglII
A’GATC’T
• BstYI (XhoII)
G/A’GATC’C/T
• BclI
T’GATC’A
• Sau3AI
‘GATC’
Grupy enzymów pozostawiających
komplementarne końce
•
•
•
•
XbaI
NheI
SpeI
AvrII
T’CTAG’A
G’CTAG’C
A’CTAG’T
C’CTAG’G
•
•
•
•
SalI
PaeR7I
XhoI
AvaI
G’TCGA’C
C’TCGA’G
C’TCGA’G
C’C/TCGG/A’G
Izoschizomery
• to enzymy pochodzące z różnych
organizmów bakteryjnych, ale
rozpoznające taką samą sekwencję i
przecinające ją identycznie. Np.:
SphI
GCATG’C
BbuI
GCATG’C
Neoschizomery
• rozpoznają taką samą sekwencję DNA lecz przecinają ją w inny
sposób. Uniemożliwia to łączenie (ligację) końców, więc
podczas planowania ligacji nie można tego przeoczyć! Np.:
Acc65I
G’GTACC
KpnI
GGTAC’C
SmaI
XmaI
CCC’GGG
C’CCGGG
BbeI
EheI
KasI
NarI
GGCGC’C
GGC’GCC
G’GCGCC
GG’CGCC
Problem dam/dcm metylacji DNA w
komórkach E. coli
Jeżeli sekwencje GATC lub CC A/T GG są
częścią sekwencji rozpoznawanej, bądź enzym
rozpoznaje i przecina taką sekwencję bezpośrednio,
to fakt ten ma swoje konsekwencje, jeśli DNA jest
otrzymywane w komórkach E. coli dam+ dcm+. W
związku z metylacją adeniny w sekwencji GATC
(dam) i wewnętrznej cytozyny w sekwencji CC A/T
GG (dcm) w dzikich szczepach E. coli, należy brać
pod uwagę wrażliwość danego enzymu
restrykcyjnego na tego typu metylację substratu.
Jeżeli zatem sekwencje GATC lub CC A/T GG
nakładają się na sekwencje rozpoznawane, należy
wiedzieć, że nie każde istniejące na danym DNA
miejsce restrykcyjne dla odpowiedniego enzymu
będzie cięte.
Problem dam/dcm metylacji DNA w
komórkach E. coli
• Lista niektórych enzymów, których aktywność
jest zależna od zmetylowanego DNA w
sekwencji dam (podkreślono). Enzymy te nie
trawią takich sekwencji:
BclI
TGAMTCA
ClaI
gAMTCGAT
DpnII, MboI
GAMTC
HphI
GGTGAMtc
MboII
GAAGAMtc
NruI
TCGCGAMtc
XbaI
TCTAGAMtc
Problem dam/dcm metylacji DNA w
komórkach E. coli
• Lista enzymów dam niezależnych
(niewrażliwych):
BamHI
GGAMTCC
BglII
AGAMTCT
BstYI
(A/G)GAMTC(C/T)
PvuI
CGAMTCG
Sau3AI
GAMTC
Problem dam/dcm metylacji DNA w
komórkach E. coli
• Lista enzymów dcm zależnych wrażliwych (sekwencje dcm podkreślono):
AvaII
GG(A/T)CCM(a/t)gg
BalI
TGGCCMAgg
EcoRII
CCM(A/T)GG
Sau96I
GGNCCM(a/t)gg
StuI
AGGCCMTgg
Problem dam/dcm metylacji DNA w
komórkach E. coli
• Lista enzymów niewrażliwvch na dcm
metylację:
BglI
GCCM(A/T)GGNNGGC
BstNI
CCM(A/T)GG
HaeIII
GGCCM(a/t)gg
KpnI
GGTACCM(a/t)gg
NarI
GGCGCCM(a/t)gg
Warunki trawienia a niespecyficzna
aktywność (ang. star activity)
• Jeżeli przeprowadza się trawienie w warunkach
znacznie odbiegających od optymalnych dla danego
enzymu, to często zdarza się, że obserwujemy
niespecyficzne cięcia.
• Dzieje się tak, ponieważ w tych warunkach enzym
rozpoznaje sekwencje różniące się od sekwencji
specyficznej (kanonicznej), np. o jedną zasadę.
• W przypadku EcoRI, dla którego sekwencją
kanoniczną jest GAATTC takie zmienione przecinane
sekwencje to np. CAATTC, GAATTG, GTATTC itp.
Warunki trawienia a niespecyficzna
aktywność (ang. star activity)
•
•
•
•
•
•
Do czynników mogących wywołać rozluźnioną
specyficzność enzymu zalicza się:
1. stężenie glicerolu powyżej 5%
2. obecność DMSO, etanolu, glikolu
etylenowego
3. zbyt niską siłę jonową mieszaniny reakcyjnej
4. zbyt wysokie pH
5. obecność innych niż Mg2+ jonów
dwuwartościowych metali (np.: Mn, Cu, Zn, Fe,
Co, Ca)
6. zbyt wysokie stężenie enzymu w próbce.
Tworzenie nowych miejsc restrykcyjnych
przez łączenie końców DNA
•
•
•
•
Nowe miejsca restrykcyjne w zrekombinowanym DNA mogą
powstawać w miejscu ligacji naturalnych końców DNA bądź
po wypełnieniu ich fragmentem Klenowa polimerazy DNA I:
powstanie nowego miejsca przypadkowo:
SspI/ClaI (Klenow) - AAT/CGAT
odtworzone ClaI (ATCGAT)
ClaI (Klenow)/SspI - ATCG/ATT
odtworzone ClaI
EcoRV/ClaI (Klenow) - GAT/CGAT
odtworzone ClaI dam-zależne (gATCGAT)
EcoRI (Klenow)/PvuII - GAATT/CTG
odtworzone EcoRI (GAATTC)
Tworzenie nowych miejsc restrykcyjnych
przez łączenie końców DNA
powstanie nowego miejsca w sposób zamierzony:
• (np. w sytuacji konieczności zlikwidowania starego miejsca
restrykcyjnego i wykreowanie nowego [uzupełnianie
fragmentem Klenowa polimerazy DNA I]:
• EcoRI/EcoRI - GAATT/AATTC
XmnI (GAANNNNTTC)
• HindIII/HindIII - AAGCT/AGCTT
NheI (GCTAGC)
• TaqI/TaqI - TCG/CGA
NruI (TCGCGA)
Lista enzymów modyfikujących
niezbędnych do klonowania
• zestaw enzymów restrykcyjnych
• fragment Klenowa polimerazy DNA I - tworzenie tępych
końców przez wypełnianie cofniętych końców 3'
• polimeraza DNA T4 - tworzenie tępych końców przez
usuwanie jednoniciowych końców 3' lub wypełnianie
cofniętych końców 3'
• kinaza polinukleotydowa T4 - fosforylacja końców 5'
• ligaza DNA T4 - łączenie końców DNA
• nukleaza mung bean - tworzenie tępych końców przez
usuwanie jednoniciowych lepkich końców
• RNazaA - degradacja RNA
• alkaliczna fosfataza - usuwanie grup fosforanowych z
końców 5'
What is a Cloning Vehicle or Vector?
• A vector is a DNA molecule in which a
foreign piece of DNA can be inserted
without loss of the vector’s ability to self
replicate.
• A vector may be a plasmid, cosmid,
artificial yeast chromosome, or virus.
Essential Features of
Plasmids
• Most vectors currently in use carry a
replicon from pMB1 or Col E1
• Both pMB1 and ColE1 are large
plasmids obtained from clinical isolates
of E. coli
• These maintain 15-20 copies in a cell
FOR MORE INFO...
Hershfield, V., H. W. Boyer, C. Yanofsky, M. A. Lovett, and D. R.
Helinski. 1974. Plasmid ColE1 as a molecular vehicle for cloning and
amplification of DNA. Proc. Natl. Acad. Sci. 71:3455
Essential Features of
Plasmids
• Plasmids such as pMB1 and ColE1
(multicopy plasmids) replicate in a
“relaxed” fashion
– Replication functions are not
encoded on the plasmid
– Uses functions from the host genes
• DNA polymerases I and III, DNAdependent RNA polymerase, etc.
Importance of “relaxed”
replication
• Plasmids can function in the absence
of ongoing protein synthesis = absence
of host replication
• If you inhibit protein synthesis with
antibiotics (chloramphenicol,
spectinomycin)…………
FOR MORE INFO...
Staudenbauer, W. L. 1978. Structure and replication of the
colicin E1 plasmid. Curr. Top. Microbiol. Immunol. 83:93.
Importance of “relaxed”
replication
• Host chromosome replication is
prevented
• Plasmids carrying the pMB1 or ColE1
replicon will continue to replicate
• Two to three thousand copies per cell
• Why is this important?
FOR MORE INFO...
Clewell, D. B. 1972. Nature of Col E1 plasmid replication in
Escherichia coli in the presence of chloramphenicol. J.
Bacteriol. 110:667.
pMB1 and ColE1 replicons
Direction of DNA replication
RNAse H
Processing
RNA II
-500
RNA I
-300
-100
ori 100
300
500
rop gene
Rop protein (63 amino acids)
pMB1 and ColE1 Replicons
• So what will happen if we alter RNA I or
rop?
– Decrease negative regulation of RNA II
– More RNA II available
– More plasmid replication
• Example
– pUC plasmids have a single mutation
(G->A) one nucleotide upstream of the
initiation of RNA I.
– pUC plasmids have 500-700 copies per
cell
Replicons Also Control
Plasmid Compatibility
• Plasmid compatibility – the ability of
two different plasmids to co-exist in
the same host
• Plasmids that utilize the same
replication system cannot co-exist in
the same bacterial cell
• Plasmids carrying the same replicon
belong to the same incompatibility
group
Over 30 Incompatibility
Groups are Known
Plasmid
Replicon
Copy Number
pBR 322 and pMB1
its derivatives
pUC vectors pMB1
15-20
pACYC and
p15A
its derivatives
pSC101 and
pSC101
its derivatives
ColE1
ColE1
10-12
500-700
~5
15-20
Plasmid Safety
• Some naturally-occurring plasmids can
be transferred to new hosts by
conjugation
• Conjugation requires three elements
– A trans-acting mobilization gene (mob)
– A cis-acting element (bom)
– A specific site that is nicked by mob (nic)
Plasmid Safety
• Some of the older plasmids such as
pBR322 are missing mob
• Some of the newer vectors such as
pUC are missing the nic/bom and
cannot be mobilized
In the 1970’s and 1980’s
• The first cloning vectors such as
pSC101 had limited functionality
• The next trend was to develop smaller
plasmids
• Advantages
– Increased efficiency of transformation
– Easier to restriction map
– Higher copy numbers
Plasmid vectors
• Circular DNA molecules that replicate independently of E.
coli chromosome.
• Are present in various copy per cell - Some are very high
copy (can be > 100 per cell); Others are low copy (1-25 per
cell).
• Three key features of plasmid vectors:
1) Origin of replication (e. g. ColE1, very high copy 500
copies per cell).
2) Antibiotic resistance (or other selectable marker).
3) Multiple cloning site (often embedded in a LacZ reporter
for ease of selecting inserts)
Desirable features of plasmids used in cloning DNA
1. Small size (<15 kb) so that it is easier to do transformation
2. Choice of restriction sites into which to insert a fragment
3. One to several selectable markers for identifying which bacteria have
“picked up” the desired plasmid with fragment
Plasmids - Vectors for Cloning DNA Fragments
• Plasmid - small (1 kb to 500 kb) extra-genomic, circular
piece of DNA maintained by bacteria and replicated
(passed on) during cell division
•
•
•
•
•
•
•
R plasmids - carry resistance to antibiotics
F plasmids - carry genes that allow transfer to other bacteria
high copy number plasmids - 10 to 100 copies in a single bacterium
low copy number plasmids - 1 to 4 copies in a single bacterium
incompatible plasmids - cannot exist together in the same bacterium
narrow-host-range plasmids - small number of species can replicate
broad-host-range plasmids - many species can replicate
Copyright © The McGraw-Hill Companies, Inc. Permission required for reproduction or display.
The First cloning
experiment involving
a recombinant DNA
assembled in vitro
Boyer and Cohen
Example 1: pBR322 as a vector used to clone fragments
•
•
•
•
•
•
•
4,361 bp
Ampr gene (w/ PstI site)
Tetr gene (w/ BamHI, , HindIII, and Sal I sites)
EcoRI site not within any coding region
funtions only in E. coli
high copy number
easily transferred to other E. coli
pBR322 restriction map
Copyright © The McGraw-Hill Companies, Inc. Permission required for reproduction or display.
Cloning foreign DNA using
the PstI site of pBR322
Copyright © The McGraw-Hill Companies, Inc. Permission required for reproduction or display.
Copyright © The McGraw-Hill Companies, Inc. Permission required for reproduction or display.
Joining of vector to insert
a) Mechanism of DNA ligase
Ligase reacts with AMP donor either ATP or NAD
Activated ligase
Activated ligase donates the AMP to the free 5'–phosphate
creating a high energy diphosphate group
With energy provided by cleavage of the bond between
phosphate groups, a new phosphodiester bond is formed
Joining of vector to insert
b) Alkaline phosphatase action
Generic rDNA Protocol
• prepare foreign DNA
• prepare vector
• ligate foreign DNA and vector
introduce rDNA into host
– heat-shock
– electroporation
Transformation
• incubate ligation mixture
with ‘competent cells’
– cells pretreated to enhance
DNA uptake
• treat according to method
– 40-41o for 1-2 minutes
– brief pulse of high voltage
Bacterial Transformation with a Plasmid
chromosome
E. Coli cell
Amps
Ampr
Permeablize membrane
with Ca2+ and heat shock
E. Coli cell
Ampr + plasmid
Select for growth in the presence of ampicillin
Screening bacteria by replica
plating
Transformation of E. coli and Selection of Desired Clones
• grow E. coli to log phase, optimal for competent cells
• CaCl2 exposure, maintained at -70° C, then heat shocked at 42 ° C
• Transformation frequency = transforms / all competent cells
• Transformation efficiency = transforms / amount of construct DNA
E. coli host cell characteristics:
• No native restriction endonucleases
• Generally unable to exchange DNA with other E. coli (RecA-)
• Do not encode for the endA1 endonuclease
Next Major Advance:
Polylinkers
• Polylinker is a tandem array of
restriction endonuclease sites in a very
short expanse of DNA
• For example, pUC18 polylinker
– Sites for 13 RE’s
– Region spans the equivalent of 20 amino
acids or 60 nucleotides
The Polylinker Advantage
•
•
•
•
Unique sites (usually)
Insert excision facilitated
Restriction endonuclease mapping and
Subcloning made easier
Example 2: pUC19 as a vector used to clone fragments
•
•
•
•
•
2,686 bp
Ampr gene
Regulatable segment of lacZ gene for b-galactosidase
EcoRI site not within any coding region
Multiple restriction sites for inserting fragments
Useful Plasmid
Features
• Relaxed Replication
• Selectable Markers
• Streamlined
• Polylinker or MCS
• Identification of
Recombinants
• most derived from
pUC or pBR322
Multiple Cloning Site:
|SacI| |ScII|
|XbaI||SpeI||BamH||SmaI||PstI||EcRI||EcRV||HIII||ClaI|
|SalI||XhoI|
|KpnI|
GAGCTCCACCGCGGTGGCGGCCGCTCTAGAACTAGTGGATCCCCCGGGCTGCAGGAATTCGATATCAAGCTTATCGATACCGTCGACCTCGAGGGGGGGCCCGGTACC
CTCGAGGTGGCGCCACCGCCGGCGAGATCTTGATCACCTAGGGGGCCCGACGTCCTTAAGCTATAGTTCGAATAGCTATGGCAGCTGGAGCTCCCCCCCGGGCCATGG
a-complementation – relies on modular structure of bgalactosidase
- basic idea is often used with cloning vectors – called insertional inactivation
a-complementation
LacZ+ - blue colony
LacZ- - while colony
-of you interrupt the lacZ
gene, the colony is white
Brock Biology of
Microorganisms,
vol. 9, Chapter 10
Polylinker from pUC118
EcoR1 SacI KpnI SmaI BamHI XbaI SalI PstI SphI HindIII
XmaI
AccI
HincII
Digest with BamHI
Add:
BamHI digested target DNA
Ligated rDNA
EcoR1 SacI KpnI SmaI BamHI
XmaI
BamHI XbaI SalI PstI SphI HindIII
AccI
HincII
Major Advance: Blue/White
Screening
• E. coli normally produces bgalactosidase
• Production is under control by the lac
operon
Z = beta galactosidase, Y = lactose permease. A = thiogalactoside
transactylase, lacI = repressor, Pi = promoter for the lac repressor,
P and O = promoter and operator
The lac operon with lactose
present
The lac operon when lactose
is absent
Major Advance: Blue/White
Screening
• Host E. coli strains have a deletion in
the N-terminus region of the lacZ gene
• These cloning vectors have the P, O
and first 58 amino acids of the lacZ
gene (alpha peptide)
• Intact, the vector in an alpha mutant
strain (ex. JM109) produces betagalactosidase
Identifying Recombinants
• based on interruption of a gene
• eg., lacZ gene = b-galactosidase
• intact b-galactosidase produces
blue color in presence of X-gal
• a-complementation or bluewhite screening
How does acomplementation
work?
It all comes down to bgalactosidase
Certain strains supply the
b-Gal fragment
When a is supplied in
trans, this allows b-Gal to
function
Current Protocols in
Molecular Biology
Which cells have the pUC19-fragment constructs?
• IPTG (isopropyl-B-D-tiogactopyranoside) is an inducer of the lac
operon regulation
• Plate the transforms onto ampicillin, IPTG and X-gal plates
• If no fragment inserted, transform will express b-galactosidase, and
it will convert X-gal into a blue product.
LB/ampicllin/X-Gal/IPTG plate showing colonies of the
E. coli JM109 competent cells for the white- blue selection
Major Advance: Blue/White
Screening
• When insert DNA in
cloned into the
polylinker separating
the start codon and
the alpha peptide
sequence in the
vector, the peptide is
not made
Hence, bacterial colonies containing vector plus
insert DNA are white
Major Advance: Blue/White
Screening
• Inducer ISOPROPYL-ß-DTHIOGALACTOPYRANOSIDE
(IPTG)
• Colorimetric indicator 5-Bromo4-chloro-3-indoxyl-beta-Dgalactopyranoside (X-gal),
colonies turn blue
The Major Limitation of
Cloning in Plasmids
• Upper limit for clone DNA size is 10 kb
• Requires the preparation of
“competent” host cells
• If the E. coli genome contains 4,639,221
base pairs, how many plasmid clones
are needed to clone the entire genome?
The Major Limitation of
Cloning in Plasmids
• Inefficient from generating genomic
libraries as overlapping regions needed
to place in proper sequence
• Preference for smaller clones to be
transformed
• Lose large genetic regions
Ligation Reaction
• mix foreign and vector DNA in
presence of DNA ligase
• optimal ratios of vector to insert generally
1.5-2:1
• intermolecular base-pairing can occur
between compatible overhangs
Intramolecular vs. Intermolecular
IV. Kinases and Phosphatases
•
add or remove phosphate groups and the 5’ ends of
DNA or RNA.
Kinase +ATP
PO4-GATC…
HO-GATC…
Phosphatase
A
(P)-(P)-(P)-
- the enzyme is not sequence-specific
O
Removal of 5’-PO4 Prevents
Vector Self Ligation
TERMINI
Identical
Overhangs
Blunt-end
Different
Overhangs
CLONING
REQUIREMENTS
Phosphatase treatment of
linear plasmid improves
efficiency.
High concentrations of DNA
and ligase needed.
Phosphatase treatment.
Purification of double-cut
plasmid increases
efficiency.
COMMENTS
Restriction sites at junctions preserved.
Both orientations of insert DNA possible.
Tandem copies of insert possible.
Restriction sites at junctions often
eliminated. Tandem copies of insert DNA
possible. Both orientations possible.
Restriction sites at junctions preserved.
Background of non-recombinants is low.
One possible orientation of insert. Tandem
copies unlikely.
Purification of Plasmids
Takes advantage of distinct topological state of plasmids.
- plasmids will be covalently closed, negatively wound circles when E. coli is
lysed.
- chormosomal DNA will be sheared into linear, non-topologically constrained
fragments (because so big).
This difference can be exploited to allow
purification of plasmids:
- difference in binding ethidium bromide,
leading to different densities (CsCl banding,
right).
- Different rater of re-associate of two strands
following denaturation by boiling or alkaline
treatment
Plasmid conformation affects
gel mobility
Figures 4.1 & 4.2
Provides extremely tight regulation and extremely
strong expression
Invitrogen website
The alkaline phosphatase from E. coli was cloned downstream of PBAD
Controlled expression systems- many different varieties
araC-PBAD cassette is now marketed by Invitrogen as a
controlled expression system
1. Tight repression in the absence of arabinose (and
presence of glucose)
2. Strong activation with addition of arabinose
Multipartite
repression loop
Class I transcription
activator
Schleif, 1996, Chapter 83, Escherichia coli and Salmonella, ASM Press
Gene fusion systems
– monitor the activity
of a gene by fusing it
to another
Current favorites are the
autofluorescent proteins
HeLa cells expressing
gfp and rfp
Clontech website
Phages as Vectors
• Have a natural advantage over
plasmids: they infect cells much
more efficiently than plasmids
transform cells, so the yield of
clones with phage vectors is usually
higher
Cloning in Charon 4
λ DNA
At least 12 kb
Max. 20 kb
Replacement vector
Copyright © The McGraw-Hill Companies, Inc. Permission required for reproduction or display.
Selection of positive
genomic clones by
Plaque hybridization
Cosmids
• Cos (cohesive ends), mids=plasmid
• Accommodate 40-50 kb
• Recombinant cosmids are packaged
into phage particles. These, cannot
replicate as phages but they are
infectious so they carry their
recombinant DNA into bacterial cells
M13 Phage Vectors
• Single-stranded DNA
• Site-directed
mutagenesis
• DNA sequencing
Copyright © The McGraw-Hill Companies, Inc. Permission required for reproduction or display.
Obtaining single-stranded DNA
by cloning in M13 phage
Phagemids
• Characteristics of both phages and
plasmids
• e.g. pBluescript (pBS)
pBluescript
A widely used plasmid
cloning vector
origin of
replication
ampicillin
resistance
gene
MCS
MCS, Multiple Cloning Site
cDNA cloning
• cDNA is a DNA copy of the mRNA
• cDNA library is a set of clones
representing as many as possible of
the mRNA in a given cell type at a given
time
Copyright © The McGraw-Hill Companies, Inc. Permission required for reproduction or display.
Nick translation
Fig. 4.14
Making a cDNA Library
Copyright © The McGraw-Hill Companies, Inc. Permission required for reproduction or display.
Using RT-PCR in cDNA
Cloning
Copyright © The McGraw-Hill Companies, Inc. Permission required for reproduction or display.
Using RT-PCR to clone a single
cDNA whose sequence is known
What kind of vector should be used
to ligate a cDNA?
• A plasmid or phagemid vector such
as pUC or pBS can be used; if so,
positive clones are usually identified
by colony hybridization with a
labeled DNA probe
• Or a λ vector, such as λgt11, as
vector → places the cloned cDNA
under the control of lac promoter so
that transcription and translation can
occur
• One can use an antibody to screen
Rapid Amplification of cDNA
Ends
(RACE)
Copyright © The McGraw-Hill Companies, Inc. Permission required for reproduction or display.
5' RACE to fill in the
5' end of a cDNA
One major issue is the size of the insert DNA
The larger the fragment, the more of the source genome can be
represented on one plasmid
For a gene library, we can easily calculate the number of individual
recombinant molecules required to represent an entire genome
N = [ln (1-P)]/[ln (1-f)]
P – probability of complete coverage
N – number of individual clones required
f – proportion of genome in average
fragment
For the human genome and a
standard plasmid
P – 99% or 0.99 confidence
f–
5 kb/4,400,000 kb = 1.14 x 10-6
N = [ln .01]/[ln 0.99999886]
= -4.6/-1.14 x 10-7
= 40,350,877 individuals
Plasmid vectors
stable inserts of 5 kb
Bacteriophage vectors can acommodate larger inserts
larger inserts than plasmids – introduced by phage infection of cells
20 – 35 kb inserts
For the human genome and a
standard plasmid
P – 99% or 0.99 confidence
f – 35 kb/4,400,000 kb = 7.95 x 106
N = [ln .01]/[ln 0.99999205]
= -4.6/-7.95 x 10-6
= 578, 616 individuals
A big improvement!
Yeast artificial chromsomes (YACs) and
bacterial artificial chromsomes (BACs)
Can accomodate from 300 – 500 kb of DNA – great for
large genomes
For the human genome and a
standard plasmid
P – 99% or 0.99 confidence
f – 500 kb/4,400,000 kb = 1.14 x 10-4
N = [ln .01]/[ln 0.999886]
= -4.6/-1.14 x 10-4
= 40,350 individuals
A even bigger improvement!
Whats a YAC?
Yeast artificial
chromsome
self-replicating
vector that can be
maintained in yeast
Can accommodate
large insert
fragments
Reeves et al., 1992, Methods Enzymol. 216:584-603
What are BACs?
bacterial artificial
chromsomes
- very low copy number
vectors that can accomodate
huge inserts
Shizuya et al, 1992, PNAS 89:9794-8797
Derived from the F plasmid of E. coli
- very stably maintained
- 1-2 copies per cell (strict copy
number contro)
