Niepełnosprawność intelektualna sprzężona
z chromosomem X
Do tej grupy chorób zaliczamy wszelkie niepełnosprawności intelektualne, które są
dziedziczone w sposób sprzężony z chromosomem X. Mężczyźni chorują zazwyczaj znacznie
częściej niż kobiety - niepełnosprawność intelektualna związana z chromosomem X odpowiada
za około 16% przypadków wrodzonych zaburzeń intelektualnych u mężczyzn.
Istnieje wiele zespołów dziedziczonych z chromosomem X, w których niepełnosprawność
intelektualna jest objawem współistniejącym, takich jak zespół Coffina-Lowry'ego, zespół
MASA, czy zespół Lescha-Nyhana.
W niniejszym teście, dzięki nowoczesnej technologii sekwencjonowania genomowego,
badamy pełne sekwencje 94 genów, odpowiedzialnych za niepełnosprawność intelektualną
sprzężoną z chromosomem X.
Z uwagi na specyfikę zastosowanej metody, niniejsze badanie nie wykryje zaburzeń
związanych z zespołem łamliwego chromosomu X.
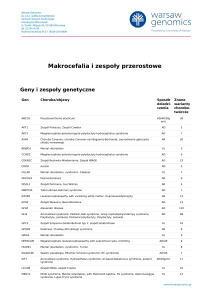
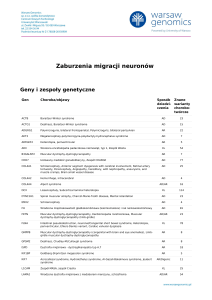
Geny i zespoły genetyczne
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
ABCD1
Adrenoleukodystrofia; Choroba Addisona i stwardnienie guzowate; choroba SiemerlingaCreutzfeldta; choroba Schildera; Melanodermia
XL
55
ACSL4
Mental retardation
XL
4
AFF2
Premature ovarian failure
XL
3
AGTR2
Mental retardation
XL
95
AP1S2
Mental retardation, syndromic, Fried (Pettigrew syndrome)
XL
6
ARHGEF6
Mental retardation
XL
2
ARHGEF9
Wczesna encefalopatia padaczkowa niemowląt, typ 8
XL
2
ARX
Wczesna encefalopatia padaczkowa niemowląt, typ 1, Zespół Westa
XL
54
ATP6AP2
Mental retardation, syndromic, Hedera, Parkinsonism with spasticity
XL
2
ATP7A
Menkes disease
XL
101
ATRX
Alfa talasemia z upośledzeniem umysłowym
XL
38
BCOR
Microphthalmia, syndromic, Oculofaciocardiodental syndrome
XL
19
BRWD3
Mental retardation
XL
5
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
CASK
Mental retardation and microcephaly with pontine and cerebellar hypoplasia, FG
syndrome, Mental retardation
XL
33
CDKL5
Wczesna encefalopatia padaczkowa niemowląt, typ 2
XL
207
CUL4B
Mental retardation, syndromic, Cabezas
XL
7
DCX
Lissencephaly, Subcortical laminal heterotopia
XL
114
DKC1
Dyskeratoza, Białaczki
XL
44
DLG3
Mental retardation
XL
8
ELK1
Niepełnosprawność intelektualna sprzężona z chromosomem X
XL
90
FANCB
Anemia Fanconiego
XL
7
FGD1
Aarskog-Scott syndrome, Mental retardation, syndromic
XL
19
FLNA
Intestinal pseudoobstruction, neuronal/Congenital short bowel syndrome, Heterotopia,
periventricular, Ehlers-Danlos variant, Cardiac valvular dysplasia
XL
78
FTSJ1
Mental retardation
XL
5
GDI1
Mental retardation
XL
4
GK
Glycerol kinase deficiency
XL
8
GPC3
Zespół Simpsona-Golabi-Behmel typ 1; zespół Golabi-Rosen
XL
16
GRIA3
Mental retardation
XL
9
HCCS
Linear skin defects with multiple congenital anomalies 1 (MIDAS syndrome)
XL
6
HPRT1
Lesch-Nyhan syndrome, Kelley-Seegmiller syndrome
XL
60
HSD17B10
17-beta-hydroxysteroid dehydrogenase X deficiency, Mental retardation, syndromic
XL
6
HUWE1
Mental retardation, syndromic, Turner
XL
8
IDS
Mucopolysaccharidosis
XL
65
IGBP1
Corpus callosum, agenesis of, with mental retardation, ocular coloboma and micrognathia
XL
1
IL1RAPL1
Mental retardation
XL
9
IQSEC2
Mental retardation
XL
10
KDM5C
Mental retardation, syndromic, Claes-Jensen
XL
14
KIAA2022
Mental retardation
XL
6
KLF8
Niepełnosprawność intelektualna sprzężona z chromosomem X
XL
85
L1CAM
Zespół MASA, zespół Crasha
XL
25
LAMP2
Choroba Danona
XL
46
MAGT1
Niedobory odporności, Hipomagnezemia
XL
4
MAOA
Brunner syndrome
XL
5
MBTPS2
Keratosis follicularis spinulosa decalvans, IFAP syndrome, Palmoplantar keratoderma,
mutilating, with periorificial keratotic plaques
XL
8
MECP2
Zespół Angelman-like
XL
425
MED12
Ohdo syndrome, Mental retardation, with Marfanoid habitus, FG syndrome, Opitz-Kaveggia
syndrome, Lujan-Fryns syndrome
XL
12
MID1
Opitz GBBB syndrome
XL
19
MTM1
Myopathy, centronuclear
XL
144
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
NDP
Exudative vitreoretinopathy, Norrie disease
XL
23
NDUFA1
Mitochondrial complex I deficiency
XL
3
NHS
Nance-Horan syndrome, Cataract
XL
18
NLGN3
Autism, Asperger syndrome
XL
75
NLGN4X
Autism, Asperger syndrome, Mental retardation
XL
4
NSDHL
Congenital hemidysplasia with ichthyosiform erythroderma and limb defects (CHILD
syndrome), CK syndrome
XL
16
NXF5
Familial heart block and focal segmental glomerulosclerosis
XL
95
OCRL
Zespół oczno-mózgowo-nerkowy; zespół Lowe'a
XL
26
OFD1
Simpson-Golabi-Behmel syndrome, Retinitis pigmentosa, Orofaciodigital syndrome,
Joubert syndrome
XL
122
OPHN1
Mental retardation, with cerebellar hypoplasia and distinctive facial appearance
XL
12
OTC
Ornithine transcarbamylase deficiency
XL
325
PAK3
Mental retardation
XL
6
PCDH19
Wczesna encefalopatia padaczkowa niemowląt typ 9; Zespół Juberga-Hellmana
XL
57
PDHA1
Leigh syndrome, Pyruvate dehydrogenase E1-alpha deficiency
XL
36
PGK1
Phosphoglycerate kinase 1 deficiency
XL
14
PHF6
Zespół Borjesona-Forssmana-Lehmana
XL
15
PHF8
Mental retardation syndrome, Siderius
XL
9
PLP1
Choroba Pelizaeusa-Merzbachera, leukodystrofia
XL
32
PORCN
Focal dermal hypoplasia
XL
6
PQBP1
Renpenning syndrome
XL
6
PRPS1
Deafness, Phosphoribosylpyrophosphate synthetase I superactivity, Arts syndrome
XL
21
RAB39B
Waisman parkinsonism-mental retardation syndrome, Waisman parkinsonism-mental
retardation syndrome, Mental retardation
XL
4
RPL10
Autism
XL
123
RPS6KA3
Coffin-Lowry syndrome, Mental retardation
XL
29
SHROOM4
Stocco dos Santos mental retardation syndrome
XL
2
SLC16A2
Zespół Allana-Herndona-Dudleya
XL
27
SLC6A8
Creatine deficiency syndrome
XL
14
SLC9A6
Niepełnosprawność intelektualna, sprzężona z chromosomem X
XL
19
SMC1A
Cornelia de Lange syndrome
XL
36
SMS
Mental retardation, Snyder-Robinson
XL
9
SOX3
Panhypopituitarism
XL
3
SRPX2
Rolandic epilepsy, mental retardation, and speech dyspraxia
XL
1
SYN1
Epilepsy, with variable learning disabilities and behavior disorders
XL
6
SYP
Mental retardation
XL
4
TIMM8A
Zespół Mohra-Tranebjaerga
XL
11
Gen
Choroba/objawy
Sposób Znane
dziedzi- warianty
czenia
chorobotwórcze
TSPAN7
Mental retardation
XL
4
UBE2A
Mental retardation, syndromic, Nascimento
XL
3
UPF3B
Mental retardation, syndromic
XL
5
ZCCHC12
Intellectual disability
XL
96
ZDHHC15
Mental retardation
XL
77
ZDHHC9
Mental retardation, syndromic, Raymond
XL
4
ZNF41
Mental retardation
XL
91
ZNF674
Mental retardation
XL
91
ZNF711
Mental retardation
XL
2
ZNF81
Mental retardation
XL
3
Metodologia
Informacja na temat metody badania:
W pierwszej kolejności, z pobranej próbki krwi lub z bloczka parafinowego izolowany jest kwas
deoksyrybonukleinowy (DNA), którego jakość i ilość jest określana w analizie
spektrofotometrycznej i fluorymetrycznej. Po mechanicznej lub enzymatycznej fragmentacji,
DNA jest wykorzystywany do stworzenia biblioteki, umożliwiającej oznaczenie, a następnie
zsekwencjonowanie i analizę genów, które zostały wybrane w ramach zleconego panelu.
Otrzymana biblioteka jest sekwencjonowana na sekwenatorze nowej generacji. Otrzymane
wyniki zostają następnie poddane analizie bioinformatycznej i interpretacji klinicznej. Warianty
genetyczne są identyfikowane z wykorzystaniem Burrows-Wheeler Aligner. Test umożliwia
wykrycie 100% substytucji i 95% małych insercji i delecji.
Informacja na temat klasyfikacji wariantów:
W raporcie z badania przedstawiana jest informacja na temat wariantów zaklasyfikowanych
jako warianty „potencjalnie patogenne” i „patogenne”, z uwagi na ich potencjalne znaczenie
kliniczne. Zidentyfikowane warianty są klasyfikowane do następujących kategorii:
Wariant patogenny: znaleziona zmiana w sekwencji genu ma bezpośredni związek
z powstawaniem choroby. Równocześnie, niektóre zmiany patogenne mogą nie mieć pełnej
penetracji, tj. pojedyncza zmiana może być niewystarczająca do wywołania pełnoobjawowej
choroby.
Wariant potencjalnie patogenny: znaleziona zmiana w sekwencji genu jest z dużym
prawdopodobieństwem związana z powstawaniem choroby, jednakże udowodnienie tego
związku nie jest możliwe w oparciu o aktualnie dostępne dane naukowe. Potwierdzenie
patogenności wariantu wymaga dodatkowych badań i dowodów; nie można wykluczyć, że
dalsze badania wykażą, że znaleziona zmiana ma niewielkie lub żadne znaczenie kliniczne.
Wariant o nieznanej patogenności: w oparciu o aktualnie dostępne dane naukowe nie ma
możliwości określenia znaczenia znalezionej zmiany.
Wariant potencjalnie łagodny: znaleziona zmiana w sekwencji genu najprawdopodobniej nie
ma związku z powstawaniem choroby, jednakże w oparciu o aktualnie dostępne dane naukowe
nie ma możliwości potwierdzenia łagodności zmiany. Potwierdzenie klinicznego znaczenia
wariantu wymaga dodatkowych badań i dowodów; nie można wykluczyć, że dalsze badania
wykażą, że znaleziona zmiana ma znaczenie kliniczne i prowadzi do rozwinięcia choroby
Wariant łagodny: znaleziona zmiana nie ma związku z powstawaniem choroby
Zidentyfikowane warianty genetyczne klasyfikowane są w oparciu o wytyczne opracowane
przez American College of Medical Genetics and Genomics i American Association for Molecular
Pathology (S. Richards, Genet Med. 2015 May;17(5):405-24). W klasyfikacji wariantów brane są
pod uwagę następujące kryteria:
wcześniejsza identyfikacja wariantu u osób obciążonych chorobą
wpływ wariantu na powstawanie funkcjonalnego produktu genu:
określony w analizach bioinformatycznych
potwierdzony w badaniach in vitro/in vivo
lokalizacja wariantu (ekson/intron, domena funkcjonalna)
zmiana de novo/dziedziczna
częstość występowania wariantu w populacji ogólnej (każdy wariant występujący
z częstością >5% zgodnie z Exome Sequencing Project, 1000 Genomes Project lub
Exome Aggregation Consortium jest klasyfikowany jako zmiana łagodna)
częstość występowania wariantu w populacji ogólnej w stosunku do populacji osób
chorych
Ostateczna klasyfikacja wariantów prowadzona jest w oparciu o sumę wymienionych kryteriów.
Przeszukiwane bazy danych obejmują: 1000GP, ClinVar, ConsensusPathDB, Exome Aggregation
Consortium, Exome Variant Server, FATHMM, GO (Gene Ontology), GTEx (Genotype-Tissue
Expression), GWAS (Genome Wide Association Study), HGMD, KEGG, MetaLR, MetaSVM,
MutationAssessor, MutationTaster, OMIM, PolyPhen-2, PROVEAN, SIFT, SnpEff, dbNSFP, UniProt,
VEP (Variant Effect Predictor).
Ograniczenia badania:
Wszystkie technologie sekwencjonowania mają swoje ograniczenia. Zlecane badanie jest
wykonywane z wykorzystaniem sekwencjonowania nowej generacji (NGS) i ma na celu
zbadanie regionów kodujących i splicingowych zleconych genów. Chociaż stosowane techniki
sekwencjonowania oraz późniejsze analizy bioinformatyczne są ukierunkowane na ograniczenie
znaczenia sekwencji pseudogenów, to jednak obecność wysoce homologicznych sekwencji
genowych może nadal sporadycznie zakłócać zdolność identyfikacji patogennych alleli, jak
i delecji/duplikacji. Sekwencjonowanie Sangera jest metodą wykorzystywaną do potwierdzania
wariantów, które uzyskały niższe parametry jakości. Analizy delecji/duplikacji wskazują na
zmiany ilościowe DNA obejmujące minimum jeden ekson i zawsze wymagają potwierdzenia
innymi metodami (qPCR lub MLPA). Wykonane analizy nie są przeznaczone do wykrywania
pewnych typów zmian genomowych, jak translokacje, inwersje, mutacje dynamiczne (np.
zwiększenie ilości powtórzeń trzynukleotydowych), zmian w regionach regulatorowych czy
intronowych. Jeśli raportowane jest zwiększenie liczby powtórzeń dwu- czy trzynukleotydowych,
to trzeba założyć, że dokładna liczba powtórzeń nie jest precyzyjna. Przeprowadzane badanie
nie jest przeznaczone do wykrywania mozaikowatości somatycznych, a analizy mutacji
somatycznych powinny być prowadzone w kontekście sekwencji DNA germinalnego.
Nie ma możliwości wykluczenia obecności mutacji w genach i rejonach innych niż objęte
wykonywanym badaniem, a także zmian liczby kopii genu. Raport z badania zawiera informację
na temat zmian w sekwencji genów zidentyfikowanych w oparciu o porównanie z aktualnymi
sekwencjami referencyjnymi zdeponowanymi w bazach danych NCBI Nucleotide i Ensembl.
Testy są opracowywane w Warsaw Genomics do celów klinicznych. Wszystkie otrzymywane
wyniki badań są interpretowane i analizowane przez ekspertów naukowych i medycznych
Warsaw Genomics.
Jak zlecić badanie
Informacja na temat metody badania:
Badanie można zlecić bezpośrednio na stronie internetowej Warsaw Genomics, poprzez
zaznaczenie wybranego testu. Zalecamy jednak, by przed każdym badaniem skonsultować się
z lekarzem, który pomoże w wybraniu odpowiedniego testu diagnostycznego, wyjaśni
możliwości i ograniczenia testów genetycznych a także przedstawi możliwe wyniki
i konsekwencje przeprowadzenia badania.
Czas realizacji badania: od 4 do 10 tygodni. Będziemy informować o kolejnych etapach analizy.
KONSULTACJA LEKARSKA
REJESTRACJA
Wybranie właściwego testu
genetycznego lub
indywidualnego zestawu
genów
Wypełnienie formularza
zlecenia testu.
Formularz wypełnia pacjent
lub wybrany przez niego
lekarz.
POBRANIE KRWI
Łącznie należy pobrać 4ml
krwi do jednej probówki
z EDTA (takiej jak na
morfologie). Pobraną krew
można przechowywać
w lodówce (w temp. +4st C)
do 7 dni
PRZESŁANIE PRÓBKI
KRWI NA NASZ ADRES
Próbkę można dostarczyć
osobiście lub kurierem
(w temperaturze pokojowej)
w ciągu 48 godzin.
Szczegółowa instrukcja
pakowania próbki
i zamówienia kuriera jest
dostępna tutaj. Do próbki
dołączamy wydrukowany
i podpisany formularz
zlecenia testu.
OPŁACENIE TESTU
Po wykonaniu testu
WYNIK BADANIA
KONSULTACJA LEKARSKA
zostanie przekazany osobie
zlecającej test – pacjentowi
lub wybranemu przez niego
lekarzowi
Jak przekazać materiał do badania?
Badanie genetyczne z krwi:
1. Krew należy pobrać do jednej probówki z EDTA (nie pobierać do probówek na skrzep,
ani na heparynę litową). Pobranie krwi może nastąpić w dowolnej godzinie, pacjent nie
musi być na czczo:
osoba dorosła - ok. 4 ml krwi żylnej do izolacji DNA (pobraną krew należy
dokładnie wymieszać z antykoagulantem i przechowywać w temperaturze 4°C)
dzieci – ok. 4 ml (minimalna ilość 2 ml) krwi żylnej do izolacji DNA (pobraną
krew należy dokładnie wymieszać z antykoagulantem i przechowywać
w temperaturze 4°C)
niemowlę – ok. 1,5 - 2 ml krwi żylnej do izolacji DNA (pobraną krew należy
dokładnie wymieszać z antykoagulantem i przechowywać w temperaturze 4°C)
2. Probówkę należy opisać imieniem i nazwiskiem i zabezpieczyć (można zakleić taśmą
klejącą).
3. Zabezpieczoną probówkę wraz z wypełnionym i podpisanym formularzem zlecenia
badania należy zapakować i wysłać zgodnie z instrukcją dostępną pod adresem:
https://badamygeny.pl/BADAMY_GENY/docs/instrukcja-wysylki-probki-krwi.pdf
Badanie genetyczne z bloczka parafinowego (profilowanie
nowotworu):
1. Należy pobrać łącznie 4 ml krwi do jednej probówki z EDTA (nie pobierać do probówek
na skrzep, ani na heparynę litową). Pobranie krwi może nastąpić w dowolnej godzinie,
pacjent nie musi być na czczo. Pobraną krew należy dokładnie wymieszać
z antykoagulantem i przechowywać w temperaturze 4°C.
2. Probówkę należy opisać imieniem i nazwiskiem i zabezpieczyć (można zakleić taśmą
klejącą).
3. Uzyskanie tkanki nowotworowej do badania w postaci:
bloczka parafinowego zawierającego wycinek nowotworu wraz z uzyskanym
z bloczka preparatem histopatologicznym (szkiełkiem) umożliwiającym
zlokalizowanie fragmentu tkanki nowotworowej,
albo wycinka tkanki nowotworowej z bloczka parafinowego o wymiarach min.
4x4x1mm, zawierającego wyłącznie tkankę nowotworową.
4. Próbkę należy opisać imieniem i nazwiskiem i zabezpieczyć (można zakleić taśmą
klejącą).
5. Zabezpieczony materiał wraz z wypełnionym i podpisanym formularzem zlecenia
badania należy zapakować i wysłać zgodnie z instrukcją dostępną pod adresem:
https://badamygeny.pl/BADAMY_GENY/docs/instrukcja-wysylki-probki-krwi.pdf
Powered by TCPDF (www.tcpdf.org)