Acta Haematologica Polonica 2009, 40, Nr 1, str. 55–66
PRACA POGLĄDOWA – Review Article
IDA FRANIAK-PIETRYGA1, MAREK MIROWSKI1, EWA LECH-MARAŃDA2,
TADEUSZ ROBAK2
Profil ekspresji genów w przewlekłej białaczce limfocytowej
określany techniką mikromacierzy
Microarray gene expression profiling in B-cell chronic lymphocytic
leukemia
1
Zakład Biochemii Farmaceutycznej Uniwersytetu Medycznego w Łodzi,
Kierownik: Prof. dr hab. n. farm. Marek Mirowski
2
Katedra i Klinika Hematologii Uniwersytetu Medycznego w Łodzi,
Kierownik: Prof. dr hab. n. med. Tadeusz Robak
STRESZCZENIE
Analiza ekspresji genów (GE) przeprowadzona techniką mikromacierzy stała się istotnym elementem biomedycznych i klinicznych badań w chorobach rozrostowych układu chłonnego. Na
chwilę obecną metoda ta jest zbyt kosztowna i czasochłonna, aby zastosować ją w rutynowej
praktyce klinicznej.
Niniejsza praca jest przeglądem piśmiennictwa dotyczącego wyników badań ekspresji genów w
przewlekłej białaczce limfocytowej (PBL). Dostarczają one istotnych informacji na temat patogenezy tej choroby i mogą mieć zastosowanie w diagnostyce i leczeniu chorych na PBL.
SŁOWA KLUCZOWE: Ekspresja genu – Mikromacierz – Przewlekła białaczka limfocytowa
SUMMARY
Gene expression (GE) analyses using microarrays have become an important part of biomedical
and clinical research in lymphoid malignancies. However, the methods are time-consuming and
costly for routine clinical practice.
In the article we review the literature regarding GE data that may provide important information
regarding pathogenesis and may be extrapolated for use in diagnosis and treatment of patients
with chronic lymphocytic leukemia.
KEY WORDS: Gene expression – Microarray – Chronic lymphocytic leukemia
WSTĘP
Nowotwory wywodzące się z limfocytów B stanowią najliczniejszą grupę chorób
limfoproliferacyjnych. Cechuje je róŜnorodny przebieg kliniczny, a takŜe zmienna
wraŜliwość na leczenie. Przewlekła białaczka limfocytowa (PBL) jest chorobą o bardzo zróŜnicowanym przebiegu klinicznym. W części przypadków zaawansowanie
choroby w chwili rozpoznania jest niewielkie, a w trakcie obserwacji nie stwierdza się
56
I. FRANIAK-PIETRYGA i wsp.
progresji choroby nawet przez wiele lat. Chorzy ci najczęściej nie wymagają początkowo równieŜ terapii. Z kolei u innych chorych choroba juŜ od momentu rozpoznania
ma charakter agresywny, prowadząc do zgonu w krótkim czasie [1–4].
Dotychczas nie udało się ustalić optymalnego sposobu postępowania terapeutycznego w PBL. Podejmowane są próby indywidualizowania terapii, w zaleŜności od
stopnia zaawansowania klinicznego choroby, wieku oraz stanu ogólnego pacjenta.
Próbuje się takŜe ustalić inne czynniki o charakterze prognostycznym, które najlepiej
przewidywałyby przebieg choroby i rokowanie u poszczególnych chorych [1–5].
W ostatnich latach dokonał się istotny postęp w badaniach nad PBL. Optymalna,
rutynowa diagnostyka białaczek opiera się o zastosowanie metod, do których zalicza
się m.in. cytomorfologię, immunofenotypowanie, cytogenetykę, fluorescencyjną hybrydyzację in situ (FISH) i reakcję łańcuchowej polimerazy (PCR). Na podstawie tych
metod badawczych moŜna opracować algorytmy diagnostyczne pozwalające na wyodrębnienie w poszczególnych jednostkach chorobowych przypadków o odmiennym
rokowaniu [1]. JednakŜe ciągle poszukuje się nowych czynników prognostycznych
ułatwiających podejmowanie właściwych decyzji terapeutycznych. Technologia mikromacierzy umoŜliwia równoległe badanie ekspresji około 50 000 genów, co czyni tę
metodę istotną w diagnostyce chorób limfoproliferacyjnych. Jej zastosowanie umoŜliwia analizę ekspresji genów na poziomie genomu [6–8].
Techniczne aspekty metody mikromacierzy
Obecnie na rynku dostępne są róŜne platformy mikromacierzy: z sondami cDNA
(komplementarny DNA) związanymi z membraną (wcześniej nazywane makromacierzami, ze względu na niską gęstość umieszczonych na nich próbek), z oligonukleotydowymi sondami cDNA, lub fragmentami genomu związanymi z podłoŜem szklanym
[9]. Pomimo, Ŝe technika mikromacierzy istnieje w wielu róŜnych odmianach, główna
zasada działania jest taka sama dla wszystkich. Badany jest cały transkryptom komórki, po izolacji całkowitego RNA, które następnie przepisywane jest w procesie odwrotnej transkrypcji na cDNA. W mikromacierzach cDNA ten właśnie materiał jest
wyznakowywany barwnikiem fluorescencyjnym i nanoszony na mikromacierz, a kaŜde
jednoniciowe cDNA w tej mieszaninie hybrydyzuje tylko z sondą o komplementarnej
sekwencji. Mozaika kolorowych punktów, która powstaje po hybrydyzacji, jest punktem wyjścia do oceny aktywności genów w badanym materiale. Zastosowanie czułych
technik detekcji fluorescencji umoŜliwia precyzyjną identyfikację kaŜdego składnika
badanej mieszaniny i ocenę ilościową jego wiązania [7, 8]. Uzyskiwane dane są gromadzone i opracowywane za pomocą zaawansowanych programów komputerowych
[10].
Mikromacierze zawierają dokładnie spozycjonowane próbki ssDNA (ang. single
stranded DNA – jednoniciowy DNA) przeznaczone do specyficznego monitorowania
ekspresji poziomu tysięcy genów w równoległym oznaczeniu. Komercyjne mikromacierze, na przykład, HG-U133 Plus 2.0 (Affymetrix, Santa Clara, California, USA)
Profil ekspresji genów w PBL
57
zawierają ok. 42 000 genów reprezentujących prawdopodobnie w większości ludzki
genom [7, 8].
Jak zatem naleŜy interpretować fakt, Ŝe na niektórych mikromacierzach znajduje
się 30 000–50 000 róŜnych sekwencji ludzkich genów, podczas gdy liczba genów
człowieka oscyluje wokół 25 000? Zwykle obecnych jest więcej sond niŜ jedna dla
danego genu, czasem sekwencja wybrana dla danej sondy nie jest optymalna i w rzeczywistości nie odpowiada znanemu genowi tylko sekwencji EST (ang. expressed
sequence tags), które są krótkimi fragmentami genów, sklonowanymi, zsekwencjonowanymi i zdeponowanymi w publicznych genowych bazach danych, a której ostateczne znaczenie biologiczne nie jest zdefiniowane. Określenie „gen” dla wykrywanych
sekwencji na mikromacierzach nie powinno więc być traktowane absolutnie dosłownie, poniewaŜ za pomocą macierzy ekspresyjnych wykrywane są nie tyle same geny,
co ich transkrypty [11].
Jak interpretować wyniki badań na mikromacierzach DNA
JeŜeli więc oceniamy obecność transkryptów genów w badanych komórkach, to
pomiar mikromacierzy daje informację o ich ekspresji. Zatem na podstawie tego badania moŜna ocenić, czy transkrypcja danego genu przebiegła intensywnie, czy teŜ przeciwnie, doszło do zahamowania ekspresji genu. PoniewaŜ badanie wykonywane jest
równolegle dla wielu tysięcy genów, otrzymany zestaw wyników nazywamy profilem
ekspresji genu (GEP, ang. gene expression profiling), a mikromacierz na której badanie
jest wykonywane – mikromacierzą ekspresyjną [11, 12]. Profil ekspresji genów określa
zarówno pełen zestaw pomiarów ekspresji genów, jak równieŜ pewną jego wyselekcjonowaną część, charakterystyczną dla danego typu komórki czy tkanki lub jej stanu
czynnościowego. Mniejsze zestawy genów o szczególnym znaczeniu nazywane są teŜ
klasyfikatorami molekularnymi, jeŜeli wyodrębniane są w celach klasyfikacji badanych tkanek lub podpisami/sygnaturami molekularnymi (ang. molecular signature),
jeŜeli słuŜą do opisywania wybranych cech badanych komórek [13].
Analiza danych z mikromacierzy
Opracowanie ogromnej ilości wyników generowanych w technice mikromacierzy
stanowi prawdziwe wyzwanie, a klasyczne metody statystyczne nie są wystarczające
do oceny tych wyników. Metody analizy, w których kaŜdy gen rozpatruje się w oderwaniu od pomiarów ekspresji innych genów, są w tym przypadku nieadekwatne. Dlatego na potrzeby analizy mikromacierzy opracowano wiele nowych testów statystycznych lub zaadaptowano wcześniej istniejące metody matematyczne, rzadko dotychczas
wykorzystywane w praktyce biologicznej [11, 14].
Jedna z obecnie stosowanych metod, nazwana klasteryzowaniem (stworzenie zbiorów podobieństwa), grup wraz z genami, oparta jest na ich wzajemnym podobieństwie
[15]. Ta technika rozpatruje kaŜdy obiekt analizy jako matematyczny element, mierzy
odległość poszczególnych elementów i konwertuje je do współczynników korelacji.
58
I. FRANIAK-PIETRYGA i wsp.
W bioinformatyce uŜywane są obecnie dwie analizy klasterowe – supervised i unsupervised. Analiza supervised grupuje chorych zgodnie z uprzednio zdefiniowaną charakterystyką i w ten sposób umoŜliwia korelację wyników ze znanymi juŜ parametrami,
takimi jak cechy kliniczne, cytomorfologiczne czy teŜ cytogenetyczne. Analiza unsupervised jest uŜywana do testowania hipotezy, czy specyficzne cechy, np. aberracje
genetyczne, są równieŜ odzwierciedleniem na poziomie ekspresji genu bez wcześniejszego zdefiniowania grupy [16–18].
Do opracowywania analiz mikromacierzy, poza analizą klasterową, uŜywa się
równieŜ analizy składowych głównych (principal component analysis) czy teŜ bardzo
podobną do niej dekompozycję wartości szczególnych (singular value decomposition)
[19]. Na szczególną uwagę zasługują metody oparte na testach permutacyjnych, a
przede wszystkim przygotowana specjalnie dla badań mikromacierzowych analiza
znaczenia mikromacierzy (significance analysis of microarrays). Bardzo przydatne dla
analizy mikromacierzy okazały się techniki bioinformatyczne oparte o sieci neuronowe
z grupy tzw. maszyn wektorów podpieranych (supported vector machines), które w
analizach profilu ekspresji genów traktowane są ostatnio jako techniki z wyboru w
szczególności przy selekcji optymalnych sygnatur czy klasyfikatorów molekularnych i
następowego weryfikowania ich przydatności [14, 20].
Profil ekspresji genów w PBL
PBL jest chorobą nowotworową, w której dochodzi do akumulacji długo Ŝyjących
limfocytów B we krwi, szpiku i obwodowych narządach limfatycznych. Pod względem
klinicznym charakteryzuje się ona zwiększeniem bezwzględnej liczby limfocytów we
krwi obwodowej, nacieczeniem szpiku, postępującym powiększeniem węzłów chłonnych, wątroby i śledziony. Na skutek zakłócenia interakcji komórkowych dochodzi
takŜe do upośledzenia odporności humoralnej jak i komórkowej. Choroba ta charakteryzuje się duŜym zróŜnicowaniem przebiegu klinicznego – od nagłej progresji ze złym
rokowaniem do łagodnego przebiegu i średniej długości Ŝycia (5–8 lat). Badania nad
profilem ekspresji genów w PBL oraz ostrych białaczkach pozwoliły na wyodrębnienie
genów o potencjalnym znaczeniu rokowniczym [1, 21].
Odmienność genetyczna białaczkowych komórek typu B od zdrowych limfocytów
Stosując róŜne mikromacierze (oligonukleotydowe i cDNA na płytkach szklanych i
membranach nylonowych), Zent i wsp. (2003) wyodrębnili 10 genów, które róŜniły się
ekspresją w limfocytach białaczkowych i limfocytach zdrowych [22]. Dalsze badania
prowadzone przez Wanga i wsp. (2004) ostatecznie pozwoliły na wyszczególnienie
kilku genów, których ekspresja towarzyszyła białaczkowym limfocytom B: FCER2
(CD23), FGR, TNFRSF1B, CCR7, IL4R, PTPN12, FMOD, TMEM1, CHS1 i ZNF266
[23].
Gen FCER2 kodujący CD23 receptor na limfocytach B zlokalizowany w rejonie
konserwatywnej sekwencji Fc cząsteczki przeciwciała o niskim powinowactwie do IgE
Profil ekspresji genów w PBL
59
jest uznanym markerem diagnostycznym dla PBL [24]. FGR jest członkiem rodziny
genu c-Src kodującym cytoplazmatyczną kinazę tyrozynową i nie wykazuje ekspresji
w prawidłowych komórkach typu B krwi obwodowej. Jego ekspresja jest jednak
obecna na limfocytach typu B strefy płaszcza w chorobie limfoproliferacyjnej oraz na
limfocytach zainfekowanych wirusem Epsteina-Barra [25]. TNFRSF1B jest czynnikiem zaleŜnym od receptora TNF odpowiadającym m.in. za wzmoŜoną proliferację
[26, 27]. IL-4R (receptor IL-4) jest obecny na wielu hematopoetycznych i niehematopoetycznych komórkach, w tym na limfocytach białaczkowych. Obecność tego genu
chroni komórki przed apoptozą w warunkach in vitro nie nasilając jednocześnie ich
proliferacji [28, 29]. CCR7 jest specyficznym receptorem dla chemokin (SLC i EBI1)
pochodzących z tkanki limfoidalnej [30, 31]. NaleŜy do rodziny receptorów białek G i
ulega znacznej ekspresji w komórkach PBL [32]. PTPN12 jest cytozolowym białkiem
o właściwościach fosfatazy tyrozynowej hamującym aktywację limfocytów [33, 34].
FMOD jest proteoglikanem, który moŜe regulować aktywność TGFβ. Dotychczas nie
stwierdzono jego aktywności w komórkach hematopoetycznych [35]. TMEM1 koduje
białko wykazujące częściową homologię z białkami kanału sodowego [36]. CHS1 jest
genem charakterystycznym w syndromie Chediak-Higashi [37]. ZNF266 jest białkiem
wykazującym charakter palców cynkowych o niezidentyfikowanej jak dotąd funkcji
[38].
Charakterystyka molekularna PBL na podstawie profilu ekspresji genu
Komórki białaczkowe PBL pod względem immunologicznym i genotypowym najbardziej przypominają dojrzałe komórki pamięci B CD5+, jednak dotychczas nie znaleziono ich zdrowych odpowiedników [32, 39]. PBL jest modelowym przykładem
zaburzeń regulacji procesów apoptozy, czego głównym przejawem jest nadekspresja
genu BCL-2 hamującego to zjawisko i akumulacja białka Bcl-2 [1].
Geny, które w PBL wykazują podwyŜszoną ekspresję w porównaniu z limfocytami
prawidłowymi typu B nazwano sygnaturami molekularnymi [39]. NaleŜą do nich takie
geny jak CD5, IL2RA (CD25) i BCL-2, jak równieŜ inne geny takie jak WNT3, TITIN,
ROR1, MRC-OX2. Gen WNT3 jest prawdopodobnie odpowiedzialny za proliferację
prekursorowych komórek B w PBL, ale funkcja ta nie została jeszcze do końca potwierdzona [6, 40]. Natomiast geny ROR1 i MRC-OX2 kodują białka powierzchniowe,
dlatego teŜ moŜna je wykorzystać przy wyborze celowanej terapii humanizowanymi
przeciwciałami monoklonalnymi [40, 41]. Zent i wsp. (2003) porównując profil ekspresji 6800 genów u nie leczonych chorych na PBL i chorych na szpiczaka mnogiego
obserwowali wysoką ekspresję genu dla czynnika martwicy nowotworu (TNF), oraz
genów szlaku dla receptora TNF (LTB, TRAF5, TNFRSF9, TNFSF7 oraz LITAF). Wysoką ekspresję zaobserwowano równieŜ w rodzinie genu IAP (BIRC1) i XIAP. Ekspresja genu BCL-2 była równieŜ znacznie wyŜsza u chorych na PBL w porównaniu do
chorych na szpiczaka mnogiego [22].
60
I. FRANIAK-PIETRYGA i wsp.
Profil ekspresji genów w PBL
61
Mutacja genu dla IgVH , ZAP70 i inne czynniki prognostyczne w PBL
W ciągu ostatnich kilku lat zmienił się sposób postrzegania biologii PBL. W zaleŜności od pochodzenia białaczkowych limfocytów B o fenotypie CD5+ moŜna wyodrębnić dwie postacie choroby na podstawie oceny regionu zmiennego genu dla łańcucha cięŜkiego immunoglobulin – IgH (IgVH). Typ I choroby charakteryzuje się występowaniem niezmutowanego genu dla cięŜkich łańcuchów, podczas gdy w typie II obserwuje się obecność zmutowanego regionu zmiennego dla łańcucha cięŜkiego immunoglobulin [1, 4]. Obecność typu I związana jest z gorszym rokowaniem w porównaniu z typem II. Chorzy z niezmutowanym genem IgVH mają zwykle bardziej zaawansowany okres kliniczny choroby w chwili rozpoznania. Występują u nich anomalie
cytogenetyczne o niekorzystnym rokowaniu. Chorzy tacy wymagają szybkiego włączenie terapii, a czas całkowitego przeŜycia jest istotnie krótszy (mediana 79–119 miesięcy) niŜ u chorych ze zmutowanym IgVH (mediana 293 miesiące) [21, 42–44].
Podtypy PBL, określone na podstawie obecności lub braku mutacji IgVH, charakteryzowała zmienna ekspresja grupy genów związanych z aktywacją limfocytów B
stymulowanych pobudzeniem receptora BCR (ang. B-cell antygen receptor). Geny,
które podczas stymulacji ulegały nadekspresji, miały o wiele wyŜszą ekspresję w typie
PBL z niezmutowanym IgVH, natomiast geny, które podczas stymulacji BCR ulegały
wyciszeniu, miały niŜszą ekspresję w tym samym typie PBL. Obserwacja ta sugeruje,
Ŝe stymulacja limfocytów B przez BCR moŜe pełnić rolę w patogenezie PBL, a utrzymująca się stymulacja BCR w podtypie PBL z niezmutowanym IgVH wiąŜe się z bardziej progresywną postacią choroby [44, 45].
W PBL profil ekspresji genu jest niezaleŜny od obecności mutacji IgVH. Wyjątek
stanowi niewielka podgrupa genów, łącznie z ZAP-70, IM1286077 i genem pektyny
typu C, która jest powiązana z obecnością zmutowanego allelu genu IgVH [46–48].
Klein i wsp. (2001) oraz Rosenwald i wsp. (2001) wykazali, Ŝe niezaleŜnie od
obecności mutacji IgVH, limfocyty białaczkowe charakteryzują się odmiennym panelem ekspresji genów w porównaniu z osobami zdrowymi, a takŜe jest porównywany z
innymi rozrostami nowotworowymi B-komórkowych [32, 39].
Rosenwald i wsp. (2001) i Weistner i wsp. (2003) zaobserwowali, Ŝe PBL z niezmutowanym IgVH, w stosunku do przypadków ze stwierdzoną mutacją, charakteryzował się ponad 4-krotnym wzrostem ekspresji ZAP70 [39, 49].
Wyniki te potwierdzili Ferrer i wsp. (2004), a dodatkowo w grupie chorych z
obecną mutacją IgVH zidentyfikowali szereg genów o zwiększonej ekspresji (RAF1,
PAX5, TCF1, CD44, SF1, S100A12, NUP214, DAF, GLVR1, MKK6, AF4, CX3CR1,
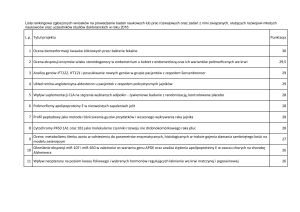
NAFTC1, HEX) (Tabela 1). Wykazali oni, Ŝe mutacji genu IgVH towarzyszy ekspresja
RAF1, PAX5, podczas gdy ekspresja ZAP70 jest charakterystyczna dla braku mutacji
tego genu [50]. ZauwaŜono takŜe, Ŝe zwiększona ekspresja ZAP70 jest powiązana z
szybką progresją choroby, skróceniem średniego czasu przeŜycia niezaleŜnie od początkowego stopnia zaawansowania klinicznego choroby [1, 21, 50].
Badania Haslinger i wsp. (2004) pokazały ponadto, Ŝe panel ekspresji genów w
przypadkach z mutacją IgVH zaleŜny jest od płci [51].
62
I. FRANIAK-PIETRYGA i wsp.
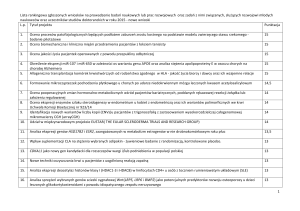
Tabela 2. Geny o niskiej ekspresji w PBL
Table 2. Genes underexpressed in CLL
Zent i wsp. (2003)
Wang i wsp. (2004)
(mikromacierz Affymetrix HuGeneFL)
(mikromacierz Affymetrix GeneChip)
MADH7
DAPK1
TYMS
VDR
GCSH
CRIP2
CD38
BLR1
RGS2
CDC2
SLC1A4
PPP1R16B
ADF
CENPF
GLRX
NEK2
BTG3
EGFL5
COX5A
PLCG2
ADE2H1
RGS13
Chorzy na PBL ze zmutowanym genem IgVH w chwili rozpoznania są zazwyczaj
we wczesnym stadium zaawansowania klinicznego choroby wg klasyfikacji Rai lub
Bineta. Często stwierdza się u nich występowanie delecji 13q14, a takŜe brak zmian w
zakresie genu supresorowego P53. Chorzy ci nie wymagają zazwyczaj natychmiastowego rozpoczęcia terapii [21]. Dotychczasowe badania potwierdzają rokownicze znaczenie obecności mutacji genu IgVH u chorych na przewlekłą białaczkę limfocytową
[1, 48]. Niestety szersze zastosowanie tego testu w praktyce klinicznej jest trudne z
powodu wysokiego kosztu badania i ograniczonej dostępności.
Porównawcze badania ekspresji genów metodą mikromacierzy znalazły teŜ zastosowanie w określaniu rokowania u chorych na PBL. Stratowa i wsp. (2001) określili
poziom ekspresji 1024 genów na macierzy cDNA u chorych na PBL. Zmniejszona
ekspresja genów kodujących interleukinę 1β (IL-1β), IL-8 i L-selektynę w komórkach
białaczkowych korelowała z istotnym skróceniem całkowitego czasu przeŜycia w porównaniu z chorymi wykazującymi wyŜszy współczynnik ekspresji wymienionych
genów. Odmienna obserwacja dotyczyła genu TCL1, gdyŜ wyŜsza ekspresja związana
była z krótszym okresem przeŜycia [52].
Zent i wsp. (2001) wykorzystali technikę mikromacierzy do róŜnicowej, porównawczej analizy ekspresji genów w przypadkach nie leczonej, agresywnej postaci PBL
oraz postaci o łagodnym przebiegu. Zidentyfikowali 16 genów przydatnych do zróŜnicowania tych dwóch postaci choroby [53].
Oporność na leczenie i wpływ na wybór terapii
Morales i wsp. (2005) w porównawczych badaniach ekspresji genów związanych
z procesem apoptozy wykorzystali komercyjną mikromacierz hybrydyzacyjną zawierającą 96 genów (www.SuperArray.com). Przebadali dwie grupy chorych na PBL.
Pierwsza obejmowała chorych wcześniej nie leczonych z łagodną postacią choroby,
z limfocytami wraŜliwymi w warunkach in vitro na indukcję apoptozy przez fludarabinę. Do drugiej grupy naleŜeli chorzy z białaczką progresywną i chemiooporną. Spo-
Profil ekspresji genów w PBL
63
śród kilku genów wyodrębnili gen BFL-1, który cechował się zwiększoną ekspresją
w drugiej grupie chorych [54].
Badania porównawczej analizy ekspresji genów prowadzono takŜe na komórkach
wyizolowanych z krwi obwodowej dwóch grup chorych ze zdiagnozowaną PBL.
Pierwsza z nich nie podlegała leczeniu, w przypadku drugiej zastosowano nieskuteczną
terapię fludarabiną, cyklofosfamidem czy chlorambucilem. Obie pule poddawano następnie naświetlaniu promieniowaniem gamma a następnie przeprowadzono porównawczą analizę ekspresji genów (mikromacierz Affymetrix z 7070 genami). Wyselekcjonowano grupę genów, które wykazały ponad 2-krotny wzrost ekspresji w limfocytach opornych na leczenie (TR3, HLA-DQA1, MTMR6, c-MYC, c-REL, cIAP1, FMOD,
MIP1α, BLK) (Vallat i wsp., 2003). Badanie to zdefiniowało nowe potencjalne markery molekularne, które mogą być specyficzne dla opornych podtypów PBL i które mogłyby mieć związek z przebiegiem klinicznym choroby [55].
Rosenwald i wsp. (2004) przeprowadzili porównawczą analizę ekspresji genów za
pomocą mikromacierzy zawierającej 17 856 klonów cDNA u chorych na PBL, leczonych fludarabiną. W badaniach in vivo wyselekcjonowali 27 genów, które ulegały
zwiększonej ekspresji (od 3 do 5 razy) po zastosowaniu fludarabiny. Sekwencje nukleotydowe 18 z nich zostały odnalezione w banku genów, a 9 kolejnych okazało się nowymi sekwencjami o jeszcze nie poznanej funkcji. Wśród nich wyróŜniono geny zaleŜne od P53 oraz geny zaangaŜowane w procesy naprawcze DNA. Podobny panel
ekspresji genów zaobserwowano po inkubacji komórek PBL z fludarabiną w warunkach in vitro. Ponadto naświetlanie komórek promieniowaniem gamma powodowało
podobne zmiany w ekspresji genów [56]. PoniewaŜ stosowanie fludarabiny in vivo
indukuje odpowiedź ekspresji genu zaleŜną od P53, autorzy sugerują, Ŝe leczenie fludarabiną moŜe doprowadzić do wyselekcjonowania komórek białaczkowych z mutacją
tego genu, które są oporne na większość leków i wiąŜą się z agresywnym przebiegiem
klinicznym. Leczenie chorych na PBL fludarabiną daje moŜliwość wyselekcjonowania
zmutowanych subklonów P53, które mogłyby dawać oporność krzyŜową z innymi
cytostatykami [56].
PODSUMOWANIE
Analizując otrzymane wyniki uzyskane metodą mikromacierzy moŜna wyodrębnić
co najmniej 5 genów o wysokiej ekspresji (IL-4R, FGR, BCL-2, FMOD oraz FCER2
(CD23)) (Tabela 1) oraz 20 genów o najniŜszej ekspresji w komórkach PBL (Tabela
2). Geny IL4R i BCL-2 związane są z upośledzonym mechanizmem apoptozy. FMOD
kodujący fibromodulinę często jest obecny w grupie chorych opornych na chemioterapię. FGR związany jest z brakiem obecności mutacji dla genu IgVH, a co za tym idzie
z bardziej progresywną postacią choroby. Informacje o zwiększonej bądź zmniejszonej
ekspresji poszczególnych genów mogą być bardzo pomocne w róŜnicowaniu agresywnej postaci choroby od PBL o łagodnym przebiegu, w wyborze właściwej terapii,
a takŜe w prognozowaniu przebiegu choroby i opracowaniu nowych testów diagnostycznych.
64
I. FRANIAK-PIETRYGA i wsp.
Ostatnie badania z uŜyciem analizy mikromacierzy DNA pozwoliły na wyodrębnienie kilkuset nowych genów, wykazujących zwiększoną ekspresję w PBL. Liczba
nowo zidentyfikowanych genów daje moŜliwość wyodrębnienia nowego profilu molekularnego PBL w powiązaniu z przebiegiem klinicznym choroby, co daje nowe szanse
diagnostyczne i terapeutyczne.
Praca naukowa finansowana ze środków na naukę w latach 2007–2010 jako
projekt badawczy (nr tematu 504-01-024).
PIŚMIENNICTWO
1.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
Dighiero G, Hamblin TJ. Chronic lymphocytic leukemia. Lancet 2008; 371: 1017–1029.
Robak T. The serach for optimal treatment in chronic lymphocytic leukemia. Acta Haematol Pol
2003; 34: 1–13.
Robak T. Alemtuzumab for B-cell chronic lymphocytic leukemia. Expert Rev Anticancer Ther 2008;
8: 1033 – 1051.
Lewandowski K, Matuszak M. Czynniki prognostyczne w przewlekłej białaczce limfatycznej Bkomórkowej. Współczesna onkologia 2003; 7: 470 – 475.
Brugiatelli M, Mannica D, Neri S i wsp. Recent update of prognosis and Stasing of chronic lymphocytic leukemia. Hematol 2003; 88: 30 – 31.
Zawada M, Czekalska S, Socha T i wsp. Technika mikromacierzy – podstawy metody oraz moŜliwości zastosowania w hematologii. Acta Haematol Pol 2004; 35: 505 – 523.
Lockhart DJ, Dong H, Byrne MC i wsp. Expression monitoring by hybridization to high-density
oligonucleotide arrays. Nat Biotechnol 1996; 14: 1675 – 1680.
Southern E, Mir K, Shchepinov M. Molecular interactions on microarrays. Nat Genet 1999; 21: 5 – 9.
Mirowski M, Bartkowiak M. Mikroprocesory DNA w badaniach biomedycznych. Postepy Biochemii
2000; 46: 272-281.
Nilsson B, Andersson A, Johansson M i wsp. Cross-platform classification in microarray-based leukemia diagnostics. Haematologica 2006, 91: 821 – 824.
Jarząb B, Gubała E, Lange D. Mikromacierze DNA i profil ekspresji genów raka brodawkowatego
tarczycy. Endokrynologia Polska 2005; 3: 294 – 301.
Greenfield A. Applications of DNA microarray to the transcriptional analysis of mammalian genomes. Mammalian Genome 2000; 11: 609 – 613.
Shaffer AL, Rosenwald A, Hurt EM i wsp. Signatures of the immune response. Immunity 2001; 15:
375 – 385.
Haferlach T, Kohlmann A, Bacher U i wsp. Gene expression profiling for the diagnosis of acute
leukemia. Br J Cancer 2007; 96: 535 – 540.
Eisen MB, Spellman PT, Brown PO i wsp. Cluster analysis and display of genome-wide expression
patterns. Proc Natl Acad Sci U S A 1998; 95: 14863 – 14868.
Brazma A, Vilo J. Gene expression data analysis. FEBS Letters 2000: 17 – 24.
Burke HB. Discovering patterns in microarray data. Mol Diagn 2000; 5: 349 – 357.
Getz G, Levine E, Domany E. Coupled two-way clustering analysis of gene microarray data. Proc
Natl Acad Sci U S A 2000; 97: 12079 – 12084.
Simek K, Kimmel M. A note on estimation of dynamics of multiple gene expression based on singular value decomposition. Mathematical Biosciences 2002; 182: 183 – 199.
Mohr S, Leikauf GD, Keith G i wsp. Microarrays as Cancer Keys: An Array of possibilities. J Clin
Oncol 2002; 20: 3165 – 3175.
Profil ekspresji genów w PBL
65
21. Robak T. Recent progress in the management of chronic lymphocytic leukemia. Cancer Treat Rev
2007; 33: 710 – 728.
22. Zent CS, Zhan F, Schichman SA i wsp. The distinct gene expression profiles of chronic lymphocytic
leukemia and multiple myeloma suggest different anti-apoptotic mechanisms but predict only some
differences in phenotype. Leuk Res 2003; 27: 765 – 774.
23. Wang J, Coombes KR, Highsmith WE i wsp. Differences in gene expression between B-cell chronic
lymphocytic leukemia and normal B cells: a meta-analysis of three microarray studies. Bioinformatics
2004; 20: 3166–3178.
24. Ikuta K, Takami M, Kim C i wsp. Human lymphocyte Fc receptor for IgE: sequence homology of its
cloned cDNA with animal lectins. Proc Natl Acad Sci USA 1987; 84: 819–823.
25. Link D, Zutter M. The proto-oncogene c-fgr is expressed in normal mantle zone B lymphocytes and is
developmentally regulated during myelomonocytic differentiation in vivo. Blood 1995; 85: 472–479.
26. Waage A, Espevik T. TNF receptors in chronic lymphocytic leukemia. Leuk Lymphoma 1994; 13:
41–46.
27. Trentin L, Zambello R, Agostini C i wsp. Expression and functional role of tumor necrosis factor
receptors on leukemic cells from patients with type B chronic lymphoproliferative disorders. Blood
1993; 81: 752–758.
28. Imani F, Rager K, Catipovic B i wsp. Interleukin-4 (IL-4) induces phosphatidylinositol 3-kinase (p85)
dephosphorylation. Implications for the role of SHP-1 in the IL-4 induced signals in human B cells. J
Biol Chem 1997; 272: 7927–7931.
29. Kay N, Han L, Bone N i wsp. Interleukin 4 content in chronic lymphocytic leukemia (CLL) B cells
and blood CD8+ T cells from B-CLL patients: impact on clonal B cell apoptosis. Br J Haematol
2001; 112: 760–767.
30. Yoshida R, Nagira M, Kitaura M i wsp. Secondary lymphoid-tissue chemokine is a functional ligand
for the CC chemokine receptor CCR7. J Biol Chem 1998; 273: 7118–7122.
31. Hasegawa H, Nomura T, Kohno M i wsp. Increased chemokine receptor CCR7/EBI1 expression
enhances the infiltration of lymphoid organs by adult T cell leukemia cells. Blood 2000; 95: 30–38.
32. Klein U, Tu Y, Stolovitzky GA i wsp. Gene expression profiling of B cell chronic lymphocytic leukemia reveals a homogeneous phenotype related to memory B cells. J Exp Med 2001; 194: 1625–
1638.
33. Davidson D, Veillette A. PTP-PEST, a scaffold protein tyrosine phosphatase, negatively regulates
lymphocyte activation by targeting a unique set of substrates. EMBO J 2001; 20: 3414–3426.
34. Yang Q, Co D, Sommercorn J i wsp. Cloning and expression of PTP-PEST. A novel, human nontransmembrane protein tyrosine phosphatase. J Biol Chem 1993; 268: 6622–6628.
35. Hildebrandt A, Romaris M, Rasmussen L i wsp. Interaction of the small of the small interstitial proteoglycans biglycan, decorin and fibromodulin with transforming growth factor beta. Biochem J
1994; 302: 527–534.
36. Yamakawa K, Mitchell S, Hubert R i wsp. Isolation and characterization of a candidate gene for
progressive myoclonus epilepsy on 21q22.3. Hum Mol Genet 1995; 4: 706–716.
37. Perou C, Leslie J, Green W i wsp. The Beige/Chediak-Higashi syndrome gene encodes a widely
expressed cytosolic protein. J Biol Chem 1997; 272: 29790– 29794.
38. Abrink M, Aveskogh M, Hellman L. Isolation of cDNA clones for 42 different Kruppel-related zinc
finger proteins expressed in the human monoblast cell line U-937. DNA Cell Biol 1995; 14: 125 –
136.
39. Rosenwald A, Alizadeh AA, Widhopf G i wsp. Relation of gene expression phenotype to immunoglobulin mutation genotype in B cell chronic lymphocytic leukemia. J Exp Med 2001; 194: 1639 –
1647.
40. Reya T, O’Riordan M, Okamura R. Wnt signaling regulates B lymphocyte proliferation through a
LEF-1 dependent mechanism. Immunity 2000; 13: 15 – 24.
41. Orchard JA, Ibbotson RE, Davis Z i wsp. ZAP-70 expression and prognosis in chronic lymphocytic
leukaemia. Lancet 2004; 363: 105 – 111.
66
I. FRANIAK-PIETRYGA i wsp.
42. Hamblin TJ, Davis Z, Gardiner A i wsp. Unmutated Ig V(H) genes are associated with a more aggressive form of chronic lymphocytic leukemia. Blood 1999; 94: 1848 – 1854.
43. Krober A, Seiler T, Benner A i wsp. V(H) mutation status, CD38 expression level, genomic aberrations, and survival in chronic lymphocytic leukemia. Blood 2002; 100: 1410 – 1416.
44. Oscier DG, Gardiner AC, Mould SJ i wsp. Multivariate analysis of prognostic factors in CLL: clinical
stage, IGVH gene mutational status, and loss or mutation of the p53 gene are independent prognostic
factors. Blood 2002; 100: 1177 – 1184.
45. Staudt LM. Gene expression profiling. Ann Rev Med 2002; 53: 303 – 318.
46. Monserrat E, Gine E, Bosch F. Redefining prognostic elements in chronic lymphocytic leukemia.
Hematol J. 2003; 4: 180 – 182.
47. Kanti Rai, Chiorazzi N. Determining the clinical course and outcome in chronic lymphocytic leukemia. N Engl J Med 2003; 348: 1797 – 1799.
48. Bosch F, Villamor N. ZAP-70 expression in chronic lymphocytic leukemia: a new parameter for an
old disease. Hematologica 2003; 88: 724 – 726.
49. Weistner A, Rosenwald A, Barry TS i wsp. ZAP-70 expression identifies a chronic lymphocytic
leukemia subtype with unmutated immunoglobulin genes, inferior clinical outcome, and distinct gene
expression profiles. Blood 2003; 101: 4944 – 4951.
50. Ferrer A, Ollila J, Tobin G i wsp. Different gene expression in immunoglobulin-mutated and immunoglobulin-unmutated forms of chronic lymphocytic leukemia. Cancer Genet Cytogenet 2004; 153:
69 – 72.
51. Haslinger C, Schweifer N, Stilgenbauer S i wsp. Microarray gene expression profiling of B-cell
chronic lymphocytic leukemia subgroups defined by genomic aberrations and VH mutation status. J
Clin Oncol. 2004; 22: 3937 – 3949.
52. Stratowa C, Loffler G, Lichter P i wsp. cDNA microarray gene expression analysis of B-cell chronic
lymphocytic leukemia proposes potential new prognostic markers involved in lymphocyte trafficking.
Int J Cancer 2001; 91: 474 – 480.
53. Zent CS, Chen JB, Evans R i wsp. Predicting disease progression in chronic lymphocytic leukaemia
from gene expression profiles. In: Program and abstracts of the 43rd Annual Meeting of the American
Society of Hematology; December 7-11, 2001; Orlando, Fla. Abstract 1970.
54. Morales AA, Olsson A, Celsing F i wsp. High expression of bfl-1 contributes to the apoptosis resistant phenotype in B-cell chronic lymphocytic leukemia. Int J Cancer 2005; 113: 730 – 737.
55. Vallat L, Magdelenat H, Merle-Beral H i wsp. The resistance of B-CLL cells to DNA damageinduced apoptosis defined by DNA microarrays. Blood 2003; 101: 4598 – 4606.
56. Rosenwald A, Chuang EY, Davis RE i wsp. Fludarabine treatment of patients with chronic lymphocytic leukemia induces a p53-dependent gene expression response. Blood 2004; 104: 1428 – 143.
Praca wpłynęła do Redakcji 6.10.2008 r. i została zakwalifikowana do druku 7.01.2009 r.
Adres do korespondencji:
Dr Ida Franiak-Pietryga
Zakład Biochemii Farmaceutycznej
Uniwersytet Medyczny w Łodzi
ul. Muszyńskiego 1
90-151 Łódź
e-mail: [email protected]